肺炎克雷伯菌是常见的机会致病菌之一,主要感染免疫力低下且患有严重基础疾病的患者,导致肺炎、败血症、化脓性肝脓肿等严重感染[1]。长期以来碳青霉烯类药物是治疗产超广谱β-内酰胺酶(extended-spectrum beta-lactamases, ESBLs)肺炎克雷伯菌相关感染的有效抗生素。然而,碳青霉烯类药物的频繁使用,导致肺炎克雷伯菌对此类药物的耐药率升高[2]。在我国,耐碳青霉烯类肺炎克雷伯菌(carbapenem-resistant Klebsiella pneumoniae, CRKP)检出率从2005年的3.00%增加到2017年的20.90%[3],给临床治疗带来挑战。本研究挑选出我国2017—2018年细菌耐药监测研究(CARST)中CRKP菌株,并对其进行药物敏感性分析和耐药基因检测,旨在为针对CRKP感染的抗菌药物选择提供数据支持。
1 材料与方法 1.1 材料 1.1.1 菌株来源北京大学临床药理研究所2017—2018年CARST中收集的CRKP,筛选条件:亚胺培南、美罗培南、厄他培南任一不敏感的非重复性临床分离株。
1.1.2 试验药物抗菌药物包括:哌拉西林(美国TargetMol公司)、舒巴坦、磷霉素氨丁三醇(山西仟源医药集团股份有限公司)、他唑巴坦、头孢西丁、头孢唑林、头孢噻肟、头孢呋辛、头孢他啶、头孢曲松、头孢哌酮、庆大霉素、阿米卡星(中国食品药品检定研究院)、头孢吡肟(中美上海施贵宝公司)、亚胺培南、厄他培南(美国默沙东制药有限公司)、美罗培南[住友制药(苏州)有限公司];氨曲南(上海新亚药业有限公司)、替加环素[美国辉瑞公司(意大利)]、磺胺甲基异
除多粘菌素E外,其他药物采用标准琼脂二倍稀释法。琼脂二倍稀释法试验细菌悬液采用多点接种仪接种,每一点接种量为1×104 CFU;多粘菌素E采用微量肉汤二倍稀释法,细菌最终浓度为5×105 CFU/mL。多黏菌素E、磷霉素氨丁三醇使用欧盟药敏试验标准委员会(EUCAST) 推荐折点,替加环素使用美国食品药品监督管理局(FDA)推荐折点,其他药敏结果根据美国临床实验室标准化协会(CLSI,2019)标准,判定敏感性和耐药性,其中头孢哌酮/舒巴坦参照头孢哌酮的标准判读。大肠埃希菌ATCC 25922和大肠埃希菌ATCC 35218被用作药敏试验的质量控制标准菌株。
1.2.2 耐药基因检测将细菌接种于中国兰琼脂平板上,37℃温箱孵育16~20 h。次日挑取单个菌落三区划线于中国兰平板分纯,37℃温箱孵育16~20 h,分离得到单个纯种菌落。第三日采用煮沸法提取DNA模板。使用聚合酶链反应(polymerase chain reaction, PCR)检测编码碳青霉烯酶(blaKPC、blaNDM、blaIMP、blaVIM、blaOXA)和其他广谱、超广谱β-内酰胺酶(blaTEM、blaSHV、blaCTX-M)基因,引物序列设计以及扩增条件参照文献[4-8],具体见表 1,每批PCR试验均带有阴性和阳性对照。反应停止后,取3 μL于1%琼脂糖凝胶电泳(含1/10 000 GelRed染料)。阳性产物送北京诺赛基因公司测序,将测序结果提交到BLAST(http://blast.ncbi.nlm.nih.gov/)进行比对序列以确定耐药基因亚型。
| 表 1 CRKP耐药基因引物序列及退火温度 Table 1 Primer sequence and annealing temperature of CRKP resistance genes |
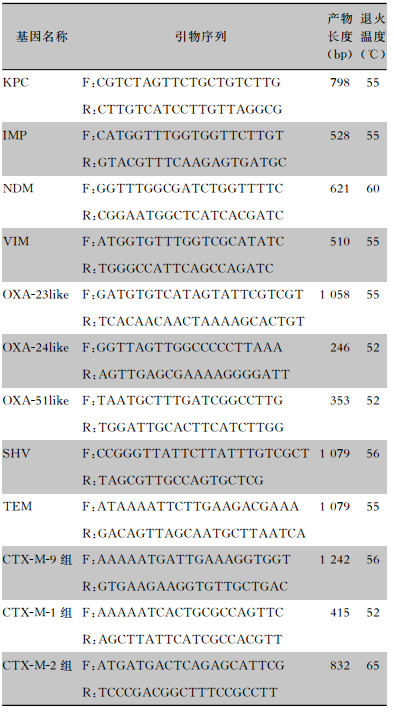
|
应用SPSS 20.0软件进行分析,药敏结果采用χ2检验进行比较,P < 0.01为差异有统计学意义。
2 结果 2.1 菌株临床分布和药敏结果CARST由北京大学临床药理所牵头,收集全国19所三级甲等医院临床分离致病菌,监测耐药状况。从北京大学临床药理研究所保存的2017—2018年CARST菌株中, 选取亚胺培南、美罗培南、厄他培南任一不敏感的非重复肺炎克雷伯菌129株, 作为本次试验的CRKP菌株。标本主要来源于血(44.19%),其次为痰、尿、引流液、分泌物、胆汁。129株CRKP,内科占38.76%,ICU占29.46%,外科占27.13%,其他科室占4.65%;儿童占16.28%,成年人占44.19%,老年人占39.53%;男性患者多于女性患者(60.47% VS 39.53%), 见表 2。CRKP对多粘菌素E敏感率最高(80.62%),其次为替加环素(79.07%)、磷霉素氨丁三醇(51.16%)、米诺环素(51.16%)、复方磺胺甲
| 表 2 CRKP在临床标本及科室中分布构成比 Table 2 Constituent ratios of CRKP in clinical specimens and departments |
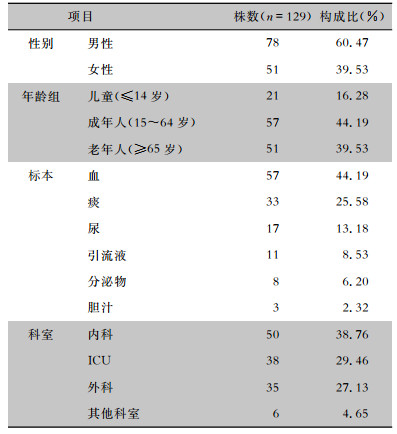
|
| 表 3 CRKP对抗菌药物的耐药率和敏感率(%) Table 3 Resistance and susceptibility rates of CRKP to antimicrobial agents(%) |
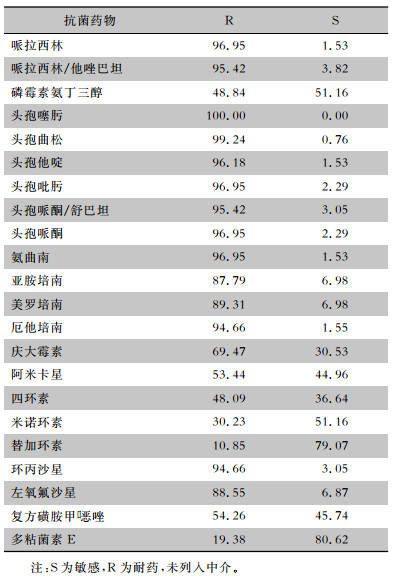
|
129株CRKP,共112株检测出碳青霉烯酶基因,涉及5种基因型,blaKPC-2、blaNDM-1、blaNDM-5、blaOXA51、blaIMP-4基因检出率分别为77.52%、6.20%、2.33%、2.33%、0.78%,其中3株同时含blaKPC-2和blaNDM基因。blaTEM检出率为51.94%,均为blaTEM-1;blaSHV检出率为64.34%,主要为blaSHV-11(18.60%)和blaSHV-12(27.91%),此外还有7株(5.43%)含blaSHV-1,3株(2.33%)含blaSHV-28;blaCTX-M检出率为53.49%,分别为blaCTX-M-9(43.41%)和blaCTX-M-15(10.08%),其中4株(3.10%)同时含blaCTX-M-9和blaCTX-M-15基因。80.36%的菌株携带ESBLs基因,32.14%的菌株同时携带2种或以上ESBLs基因。见表 4。
| 表 4 129株CRKP耐药基因检出情况 Table 4 Detection results of antimicrobial resistance genes in 129 strains of CRKP |
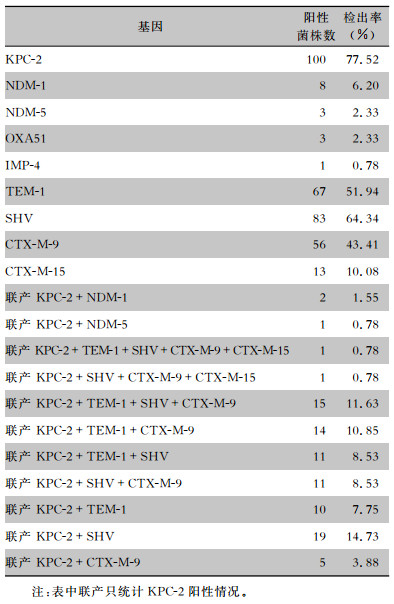
|
不产碳青霉烯酶菌株对β-内酰胺类药物中碳青霉烯类、β-内酰胺类复合剂及部分第三代头孢菌素敏感率高于产酶菌(P < 0.01)。在产碳青霉烯酶的CRKP中,相对blaKPC-2基因阳性菌,亚胺培南、美罗培南和氨曲南对blaNDM基因阳性菌株具有更好抗菌作用,MIC50和/或MIC90值低于blaKPC-2基因阳性菌。对于非β-内酰胺类药物,blaKPC-2基因阳性菌对磷霉素氨丁三醇均表现出较低敏感率(P < 0.01);不产碳青霉烯酶菌株对多粘菌素E敏感率低于产酶菌(P < 0.01)。见表 5。
| 表 5 blaKPC-2和blaNDM基因阳性菌株与不产碳青霉烯酶菌株的药敏结果 Table 5 Antimicrobial susceptibility testing result of blaKPC-2 and blaNDM gene positive as well as carbapenemase gene negative bacteria strains |
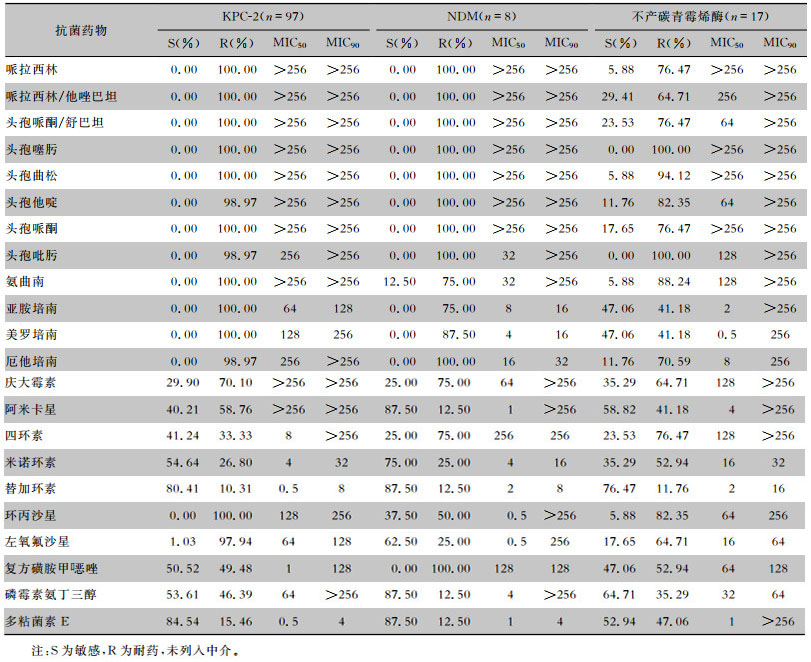
|
产碳青霉烯酶肺炎克雷伯菌通常对大多数β-内酰胺类和氟喹诺酮类抗菌药物耐药,治疗CRKP感染的选择仅限于多粘菌素E、替加环素、磷霉素氨丁三醇、米诺环素和一些氨基糖苷类药物[9],与本研究体外药敏结果较为一致。在CISL 2020更新的折点中,关于多粘菌素E对于大肠埃希菌有中介(≤ 2 μg/mL)或耐药(≥ 4 μg/mL)的折点,本研究对多粘菌素的折点使用EUCAST标准。所有CRKP菌株对多粘菌素E和替加环素具有较高的药物敏感性,与Lin等[10]研究结果较一致,但也应避免滥用此两种抗生素,因为CRKP对其耐药率可能会呈现逐渐上升的趋势[11],将会给临床医生的治疗带来挑战。本研究药敏结果显示,CRKP对复方磺胺甲
肺炎克雷伯菌对碳青霉烯类抗生素常见的耐药机制是其产碳青霉烯酶,广泛报道的碳青霉烯酶含A、B和D类。A类中KPC-2和KPC-3型是临床最常见的,在北美、南美、欧洲和亚洲均报告过医院暴发感染的情况,此外,也有GES型的报告;B类为金属-β-内酰胺酶,主要是NDM型、VIM型和IMP型,其中NDM型在全球广泛传播令人担忧,水解的底物包括β-内酰胺类抗生素,但对于氨曲南没有水解活性;D类通常能水解苯唑西林的OXA酶[13-14]。本研究中,检测出的碳青霉烯酶基因主要为blaKPC-2(77.52%)基因,与国内一项研究报告[15]中blaKPC-2(73.54%,517/703)是我国肺炎克雷伯菌携带的最常见的碳青霉酶基因结论一致。blaNDM是大肠埃希菌携带的主要碳青霉烯酶基因,本次检测出11株blaNDM基因阳性CRKP,其他碳青霉烯酶基因的检出率则低很多。同时本研究数据显示,blaKPC-2和blaNDM基因阳性菌株有明显耐药性差异,blaKPC-2基因阳性菌株对绝大多数所测药物耐药率高于blaNDM基因阳性菌株,表现出泛耐药特性,只对四环素和复方磺胺甲
ESBLs是由质粒介导的超广谱β-内酰胺酶,能够使细菌对青霉素类、第一至第四代头孢菌素类和氨曲南耐药;ESBLs最流行的是SHV和CTX-M两种类型[16]。本研究碳青霉烯酶基因阳性菌株中,80.36%(90/112)同时携带ESBLs基因,主要是blaSHV-12和blaCTX-M-9基因,32.14%(36/112)同时携带2种或以上ESBLs基因,与CRKP耐药基因以KPC-2型为主,同时携带ESBLs基因的报道[17-18]相似。
综上论述,我国临床CRKP分离株耐药问题严重且携带多种耐药基因,CRKP携带的主要碳青霉烯酶基因是blaKPC-2,最常见的ESBLs基因是blaSHV-12和blaCTX-M-9基因,需要加强CRKP中碳青霉烯酶的监测。本文特色是试验菌株来自全国不同地区,并且同时检测了碳青霉烯酶基因与ESBLs基因,药敏试验涵盖较全的临床治疗药物,但不足之处是耐药菌株未进行同源性检测,可在后续CRKP遗传背景中深入地研究。
| [1] |
Podschun R, Ullmann U. Klebsiella spp. as nosocomial pathogens: epidemiology, taxonomy, typing methods, and pathogenicity factors[J]. Clin Microbiol Rev, 1998, 11(4): 589-603.[J]. Clin Microbiol Rev, 1998/, 11(4): 589-603. DOI:10.1128/CMR.11.4.589 |
| [2] |
Su JC, Guo QL, Li Y, et al. Comparison of empirical therapy with cefoperazone/sulbactam or a carbapenem for bloodstream infections due to ESBL-producing Enterobacteriaceae[J]. J Antimicrob Chemother, 2018, 73(11): 3176-3180. DOI:10.1093/jac/dky323 |
| [3] |
Hu FP, Zhu DM, Wang F, et al. Current status and trends of antibacterial resistance in China[J]. Clin Infect Dis, 2018, 67(Suppl 2): S128-S134. |
| [4] |
杨启文. 中国碳青霉烯耐药肠杆菌科菌流行病学及耐药机制研究[D]. 北京: 北京协和医学院, 2015.
|
| [5] |
Poirel L, Walsh TR, Cuvillier V, et al. Multiplex PCR for detection of acquired carbapenemase genes[J]. Diagn Microbiol Infect Dis, 2011, 70(1): 119-123. DOI:10.1016/j.diagmicrobio.2010.12.002 |
| [6] |
Yousefi S, Farajnia S, Nahaei MR, et al. Detection of metallo-β-lactamase-encoding genes among clinical isolates of Pseudomonas aeruginosa in northwest of Iran[J]. Diagn Microbiol Infect Dis, 2010, 68(3): 322-325. DOI:10.1016/j.diagmicrobio.2010.06.018 |
| [7] |
Woodford N, Fagan EJ, Ellington MJ. Multiplex PCR for rapid detection of genes encoding CTX-M extended-spectrum β-lactamases[J]. J Antimicrob Chemother, 2006, 57(1): 154-155. DOI:10.1093/jac/dki412 |
| [8] |
马序竹, 吕媛, 张佳, 等. 耐亚胺培南鲍曼不动杆菌耐药性及OXA碳青霉烯酶检测[J]. 中国临床药理学杂志, 2011, 27(4): 268-271. DOI:10.3969/j.issn.1001-6821.2011.04.009 |
| [9] |
Thaden JT, Pogue JM, Kaye KS. Role of newer and re-emerging older agents in the treatment of infections caused by carbapenem-resistant Enterobacteriaceae[J]. Virulence, 2017, 8(4): 403-416. DOI:10.1080/21505594.2016.1207834 |
| [10] |
Lin L, Xiao XG, Wang XN, et al. In vitro antimicrobial susceptibility differences between carbapenem-resistant KPC-2-producing and NDM-1-producing Klebsiella pneumoniae in a teaching hospital in northeast China[J]. Microb Drug Resist, 2020, 26(2): 94-99. DOI:10.1089/mdr.2018.0398 |
| [11] |
Jeannot K, Bolard A, Plésiat P. Resistance to polymyxins in Gram-negative organisms[J]. Int J Antimicrob Agents, 2017, 49(5): 526-535. DOI:10.1016/j.ijantimicag.2016.11.029 |
| [12] |
Su JC, Li D, Guo QL, et al. In vitro bactericidal activity of trimethoprim-sulfamethoxazole/colistin combination against carbapenem-resistant Klebsiella pneumoniae clinical isolates[J]. Microb Drug Resist, 2019, 25(2): 152-156. DOI:10.1089/mdr.2018.0085 |
| [13] |
Hammoudi Halat D, Ayoub Moubareck C. The current burden of carbapenemases: review of significant properties and dissemination among Gram-negative bacteria[J]. Antibiotics (Basel), 2020, 9(4): 186. DOI:10.3390/antibiotics9040186 |
| [14] |
Pitout JD, Nordmann P, Poirel L. Carbapenemase-producing Klebsiella pneumoniae, a key pathogen set for global nosocomial dominance[J]. Antimicrob Agents Chemother, 2015, 59(10): 5873-5884. DOI:10.1128/AAC.01019-15 |
| [15] |
Zhang R, Liu LZ, Zhou HW, et al. Nationwide surveillance of clinical carbapenem-resistant Enterobacteriaceae (CRE) strains in China[J]. EBioMedicine, 2017, 19: 98-106. DOI:10.1016/j.ebiom.2017.04.032 |
| [16] |
Bhaskar BH, Mulki SS, Joshi S, et al. Molecular characterization of extended spectrum β-lactamase and carbapenemase producing Klebsiella pneumoniae from a tertiary care hospital[J]. Indian J Crit Care Med, 2019, 23(2): 61-66. DOI:10.5005/jp-journals-10071-23118 |
| [17] |
Wang Q, Wang XJ, Wang J, et al. Phenotypic and genotypic characterization of carbapenem-resistant Enterobacteriaceae: data from a longitudinal large-scale CRE study in China (2012-2016)[J]. Clin Infect Dis, 2018, 67(Suppl 2): S196-S205.. |
| [18] |
Zhao F, Zhang J, Fu Y, et al. Dissemination of extensively drug-resistant and KPC-2 producing Klebsiella pneumoniae isolated from bloodstream infections[J]. J Infect Dev Ctries, 2015, 9(9): 1016-1021. DOI:10.3855/jidc.6679 |


 ,
,