骨关节感染临床上较为常见,尽管诊疗水平不断提高,但仍有许多骨关节感染患者因此关节功能受限,生活质量下降,甚至最终残疾或死亡[1-2]。快速、准确的病原学诊断有助于抗感染治疗和手术方案的制定。目前,微生物培养是鉴定骨关节病原菌感染的主要方法,广泛应用于临床,但该方法耗时久,灵敏度相对较低,因此,迫切需要能更快、更准确鉴定病原体的新诊断方法。宏基因组二代测序(metagenomic next-generation sequencing,mNGS)是一种新型感染性疾病临床诊断技术,越来越多用于检测疑似感染病例中的病原体。这种无偏倚的检测方法不依赖于传统微生物培养,无需预设,可检测标本中所有核酸序列,通过识别脱氧核糖核酸(DNA)和/或核糖核酸(RNA)这些生物标志物来鉴定标本中的病原体,广泛鉴定微生物种类,从而诊断不同类型的感染[3-4]。本研究基于微生物测序技术,评估mNGS检测病原体的灵敏度和可靠性,并探讨其在骨关节感染病原学诊断中的临床应用价值。
1 对象与方法 1.1 研究对象纳入2021年1月—2022年6月收治于广东省某医院骨科病区,依据临床症状、体征、影像学CT检查及病原学结果诊断为疑似骨关节感染的患者。通过电子病历系统收集患者年龄、性别、基础疾病、临床症状、实验室检查、影像学CT检查、病原学结果等临床资料。本研究经医院伦理委员会批准,所有患者均签署知情同意书。
1.2 标本采集收集患者经手术途径获取的疑似感染病灶组织标本,或穿刺抽取的关节液、静脉血及骨髓。采样后,快速收集到无菌容器中并立即送检。
1.3 微生物培养研磨组织标本成组织匀浆,分别接种于血琼脂、巧克力及麦康凯平板行细菌培养,沙氏琼脂培养基行真菌培养,分枝杆菌(MGIT 960)培养系统行分枝杆菌培养,关节液标本直接注入需氧及厌氧血培养瓶送检、培养。
1.4 mNGS检测 1.4.1 标本处理和DNA提取采用离心沉淀、过滤、磁珠捕获等方法对不同类型标本进行预处理,去除细胞碎片和蛋白质等。用移液器吸取标本上清液至加样卡盒(含细胞/组织基因组提取纯化试剂盒及基因组DNA建库试剂盒)中,将卡盒放入NGS masterTM自动提取建库仪行核酸提取。
1.4.2 高人源标本去宿主化处理使用皂苷裂解高人源背景标本的人源细胞,使用脱氧核糖核酸酶(DNase)降解释放出的人源核酸,仅保留用于下游分析的完整微生物,以达到去宿主化目的。
1.4.3 DNA文库构建和测序通过自动核酸提取建库仪制备DNA文库,包括核酸提取、酶切片段化、补平加A、接头连接和纯化。文库经荧光PCR定量后进行等量混合和高通量测序。
1.4.4 生物信息学分析应用病原宏基因组数据分析软件Gentellix分析原始测序数据,并与参考数据库比对,以鉴定微生物种类。
1.5 统计学方法应用SPSS 23.0软件分析数据,采用χ2检验比较mNGS和微生物培养的病原体检出阳性率,以P≤0.05为差异有统计学意义。以临床确诊为金标准,计算并比较mNGS检测、微生物培养、影像学CT检查诊断骨关节感染的灵敏度、特异度、阳性预测值和阴性预测值。
2 结果 2.1 一般资料共纳入108例疑似骨关节感染患者,其中男性62例,女性46例,年龄21~92岁,共送检标本126份,大部分患者临床症状为感染部位疼痛。见表 1。
| 表 1 患者的基本情况和标本信息 Table 1 Basic information of patients and specimens |

|
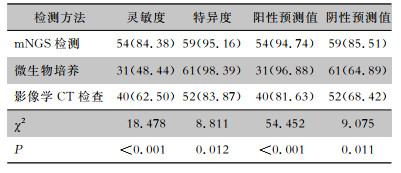
临床上,任一病原学检查(微生物培养、mNGS检测、病理学检查)发现病原体即确诊骨关节感染,据此,本研究病原学检查共检出阳性标本64份,阴性标本62份。mNGS检测的灵敏度和阴性预测值分别为84.38%、85.51%,均高于微生物培养(48.44%、64.89%)和影像学CT检查(62.50%、68.42%),差异均有统计学意义(均P<0.05)。微生物培养特异度和阳性预测值分别为98.39%、96.88%,均高于mNGS检测(分别为95.16%、94.74%) 和影像学CT检查(分别为83.87%、81.63%),差异均有统计学意义(均P<0.05)。见表 2。
| 表 2 三种检测方法的诊断效能比较[份(%)] Table 2 Comparison of diagnostic efficacy of three detection methods (No. of detected cases [%]) |
|
mNGS检出阳性标本54份,检出病原菌65种,以金黄色葡萄球菌和结核分枝杆菌为主;微生物培养检出阳性标本31份,检出病原菌36种,以金黄色葡萄球菌为主;两种检测方法阳性检测结果一致标本29份。mNGS检测阳性率(42.86%,54/126)高于微生物培养(24.60%,31/126),差异有统计学意义(P < 0.05)。mNGS检测结核分枝杆菌阳性率(9.52%,12/126)高于微生物培养(2.38%,3/126)。14.81%(8/54)的mNGS检测阳性标本检出两种及以上病原体。见图 1。
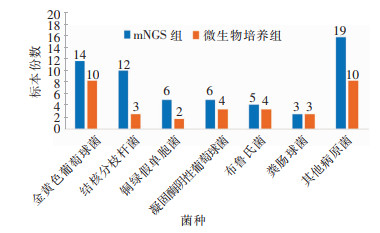 |
| 图 1 mNGS检测和微生物培养结果 Figure 1 mNGS detection and microbial culture results |
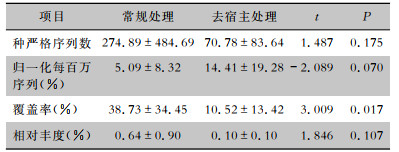
对其中56份高人源背景的标本进行去宿主处理。与常规处理相比,去宿主处理后mNGS的覆盖率有所降低(t=3.009,P=0.017),种严格序列数、归一化每百万序列、相对丰度各组比较,差异无统计学意义(P>0.05),见表 3。去宿主前后mNGS与临床诊断符合率分别为87.50%(49份)和78.57%(44份),差异无统计学意义(P=0.125)。
| 表 3 去宿主前后mNGS检测结果的差异 Table 3 Differences in mNGS results pre- and post-host removal |
|
近年来,mNGS技术在感染病原学领域的应用受到越来越多的关注和认可。mNGS使用随机引物进行无差别扩增,比传统检测方法灵敏度更高,是病原检测领域一项突破性技术。然而,其病原体单次检测费用较高,基于成本、医保方面的考虑,临床难以常规实施。但对于重症患者,尤其是重症监护病房(ICU)患者,快速全谱的病原体鉴定可争取靶向治疗时间,减少其他尝试性药物的使用,综合治疗费用可能更低,更具经济效益。大量研究评估了mNGS临床病原学诊断的效能,前期主要集中在病例报告或小规模病例,目前有了更大规模的研究。研究涉及的感染性疾病主要包括中枢神经系统感染、脓毒症、不明原因发热、心内膜炎、呼吸道感染、骨关节感染、眼部感染及组织感染等,随着涉及范围的拓展,mNGS逐渐显示出其特有的优越性[4]。在2018年的国际专家共识会议上,mNGS被明确推荐为诊断假体周围关节感染的重要补充检测方法[5-6]。
目前,传统微生物学培养是鉴定病原体的金标准,但由于取样前使用抗菌药物,标本采集和运送过程不规范,培养时间过短,以及苛养菌、特殊菌的存在等原因,微生物培养检测灵敏度不高[7-8]。本研究显示mNGS诊断骨关节感染的灵敏度和特异度分别为84.38%、95.16%,灵敏度高于微生物培养(48.44%)和影像学CT检查(62.50%),差异有统计学意义,与文献[9-11]报道一致。mNGS不仅可检测到微生物培养检测阳性的病原体,还能从其阴性标本中检测到病原体,而对明确培养出病原菌的标本进行mNGS检测,其序列数、相对丰度均较高。本研究25份微生物培养阴性标本经mNGS检测到病原菌,与文献[12-13]报道一致。mNGS的灵敏度也高于微生物培养,尤其针对微生物培养阴性标本。理论上,mNGS测序过程无偏倚,可以覆盖标本中所有已知基因序列的微生物,检测临床标本中所有病原体,尤其适用于临床上复杂的感染病例,且该检测足够敏感,可检测到标本中低水平的微生物核酸或残留核酸[14]。
本研究显示,mNGS检测阳性率(42.86%)高于微生物培养(24.60%),差异有统计学意义,检出病原体主要为金黄色葡萄球菌和结核分枝杆菌,与文献[15]相符。本研究还评估了mNGS对骨关节结核分枝杆菌感染的诊断效能,发现mNGS检测结核分枝杆菌的阳性率高于微生物培养法,与文献[16-17]报道一致。微生物培养往往只能检出单一病原体,这可能与不同种类病原微生物之间的生存竞争有关[18]。mNGS在混合感染或多种不明病原体检测方面比微生物培养更具优势,能检出更多混合菌。本研究14.81%的mNGS阳性标本检出两种及以上病原体,但需进一步区别感染、定植及污染。mNGS技术的高灵敏度易导致高假阳性率,临床一方面应分析mNGS检出的病原体及相关测序指标,同时还需结合患者临床症状及其他辅助诊断方法的检测结果综合判断,进一步区分污染与感染。
宿主核酸也是干扰mNGS检测病原体的重要因素。部分临床标本存在大量宿主细胞和宿主游离核酸,导致测序数据中大部分为宿主DNA信息,而病原微生物数据占比较少,此外,宿主DNA的非特异性序列也会干扰下游病原微生物的判断。因此,对高宿主背景的标本,须在提取过程中去除或部分去除宿主细胞或核酸,提高检测灵敏度。本研究对人源指数超过80%的同类标本采用差异化裂解方法进行去宿主处理。皂苷类化学物质选择性裂解人源细胞后,再通过核酸酶降解释放出的人源核酸,对微生物进行富集,富集效率可提高1 000倍[19]。但这种处理可能会损失某些细胞壁薄的微生物(如肺炎链球菌),从而产生假阴性结果,需予以注意[20]。本研究中,去宿主前后mNGS检测与临床诊断符合率分别为87.50%、78.57%,两种方法检测阳性率比较,差异无统计学意义。考虑去宿主处理的标本数较少,可能导致结果存在一定偏倚,需更大数据进行验证;另一方面,也需进一步优化去宿主方案。
本研究存在一定局限性。该研究为回顾性、单中心研究,变量较多,仍需开展多中心、大样本的前瞻性研究进一步证实结论。
综上所述,本研究系统比较了mNGS检测、微生物培养及影像学CT检查在检测骨关节感染病原体方面的效能,发现mNGS对病原体的检出率更高,可检测到被微生物培养法漏检的潜在病原体,有助于更多病原体的鉴别,同时,其诊断骨关节感染病原体灵敏度也高。作为传统检测方法(如微生物培养)的补充手段,mNGS具有重要临床价值。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Nadau E, Joseph C, Haraux E, et al. Clinical features and outcomes in children with bone and joint infections of the ankle or foot[J]. Arch Pediatr, 2020, 27(8): 464-468. DOI:10.1016/j.arcped.2020.08.005 |
| [2] |
沈建伟, 龙如超, 戚飞成. 骨关节术后医院感染病原学及其危险因素[J]. 中华医院感染学杂志, 2021, 31(6): 901-905. Shen JW, Long RC, Qi FC. Etiological characteristics and risk factors for postoperative nosocomial infection in orthopedic joint surgery patients[J]. Chinese Journal of Nosocomiology, 2021, 31(6): 901-905. |
| [3] |
Chiu CY, Miller SA. Clinical metagenomics[J]. Nat Rev Ge-net, 2019, 20(6): 341-355. DOI:10.1038/s41576-019-0113-7 |
| [4] |
Miller S, Chiu C. The role of metagenomics and next-generation sequencing in infectious disease diagnosis[J]. Clin Chem, 2022, 68(1): 115-124. |
| [5] |
Shohat N, Bauer T, Buttaro M, et al. Hip and knee section, what is the definition of a periprosthetic joint infection (PJI) of the knee and the hip? Can the same criteria be used for both joints?: Proceedings of international consensus on orthopedic infections[J]. J Arthroplasty, 2019, 34(2S): S325-S327. |
| [6] |
李韬, 高琪乐, 刘少华, 等. 宏基因组测序在骨关节感染诊断中的应用前景[J]. 中国矫形外科杂志, 2022, 30(10): 898-901. Li T, Gao QL, Liu SH, et al. The application prospects of metagenomic next-generation sequencing in the diagnosis of osteoarticular infections[J]. Orthopedic Journal of China, 2022, 30(10): 898-901. |
| [7] |
Yoon HK, Cho SH, Lee DY, et al. A review of the literature on culture-negative periprosthetic joint infection: epidemiology, diagnosis and treatment[J]. Knee Surg Relat Res, 2017, 29(3): 155-164. DOI:10.5792/ksrr.16.034 |
| [8] |
Palan J, Nolan C, Sarantos K, et al. Culture-negative periprosthetic joint infections[J]. EFORT Open Rev, 2019, 4(10): 585-594. |
| [9] |
薛白, 杨素珉, 张科翼, 等. 宏基因组二代测序技术对骨关节感染诊断的潜在应用价值[J]. 中华临床感染病杂志, 2021, 14(2): 127-132. Xue B, Yang SM, Zhang KY, et al. Application value of meta-genomic next generation sequencing in diagnosis of osteoarticular infections[J]. Chinese Journal of Clinical Infectious Diseases, 2021, 14(2): 127-132. DOI:10.3760/cma.j.issn.1674-2397.2021.02.009 |
| [10] |
Miao Q, Ma YY, Wang QQ, et al. Microbiological diagnostic performance of metagenomic next-generation sequencing when applied to clinical practice[J]. Clin Infect Dis, 2018, 67(Suppl-2): S231-S240. |
| [11] |
Huang ZD, Wu QQ, Fang XY, et al. Comparison of culture and broad-range polymerase chain reaction methods for diagnosing periprosthetic joint infection: analysis of joint fluid, periprosthetic tissue, and sonicated fluid[J]. Int Orthop, 2018, 42(9): 2035-2040. DOI:10.1007/s00264-018-3827-9 |
| [12] |
Huang ZD, Zhang ZJ, Yang B, et al. Pathogenic detection by metagenomic next-generation sequencing in osteoarticular infections[J]. Front Cell Infect Microbiol, 2020, 10: 471. DOI:10.3389/fcimb.2020.00471 |
| [13] |
Thoendel MJ, Jeraldo PR, Greenwood-Quaintance KE, et al. Identification of prosthetic joint infection pathogens using a shotgun metagenomics approach[J]. Clin Infect Dis, 2018, 67(9): 1333-1338. |
| [14] |
Geng SK, Mei Q, Zhu CY, et al. Metagenomic next-generation sequencing technology for detection of pathogens in blood of critically ill patients[J]. Int J Infect Dis, 2021, 103: 81-87. |
| [15] |
Zhao MW, Tang K, Liu FS, et al. Metagenomic next-generation sequencing improves diagnosis of osteoarticular infections from abscess specimens: a multicenter retrospective study[J]. Front Microbiol, 2020, 11: 2034. |
| [16] |
Zhou X, Wu HL, Ruan QL, et al. Clinical evaluation of diagnosis efficacy of active Mycobacterium tuberculosis complex infection via metagenomic next-generation sequencing of direct clinical samples[J]. Front Cell Infect Microbiol, 2019, 9: 351. |
| [17] |
Huang ZD, Zhang CJ, Hu DQ, et al. Diagnosis of osteoarticular tuberculosis via metagenomic next-generation sequencing: a case report[J]. Exp Ther Med, 2019, 18(2): 1184-1188. |
| [18] |
Simner PJ, Miller S, Carroll KC. Understanding the promises and hurdles of metagenomic next-generation sequencing as a diagnostic tool for infectious diseases[J]. Clin Infect Dis, 2018, 66(5): 778-788. |
| [19] |
Charalampous T, Kay GL, Richardson H, et al. Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection[J]. Nat Biotechnol, 2019, 37(7): 783-792. |
| [20] |
Street TL, Barker L, Sanderson ND, et al. Optimizing DNA extraction methods for nanopore sequencing of Neisseria gono-rrhoeae directly from urine samples[J]. J Clin Microbiol, 2020, 58(3): e01822-19. |



