2. 长江水利委员会长江医院中医科, 湖北 武汉 430019
2. Department of Traditional Chinese Medicine, Changjiang Water Resources Commission Changjiang Hospital, Wuhan 430019, China
肠球菌已成为导致医院感染的重要病原菌,其中又以粪肠球菌(Enterococcus faecalis,E. faecalis)和屎肠球菌(Enterococcus faecium,E. faecium)最常见。第一代恶唑烷酮类抗生素利奈唑胺(linezolid,LZD)于2000年首先在美国被批准用于临床,对大多数革兰阳性菌,特别是耐万古霉素肠球菌(vancomycin-resistant Enterococcus, VRE)显示出强大的抗菌活性,但应用于临床后不久就有耐利奈唑胺肠球菌的报道[1-2]。多重耐药肠球菌对公众卫生安全构成严重威胁,泰利唑胺(tedizolid,TZD)作为第二代恶唑烷酮类抗生素于2014年6月由美国食品和药物管理局批准用于临床, 治疗成人急性细菌性皮肤及软组织感染[3-5]。然而,近年来也有对泰利唑胺不敏感肠球菌出现的相关报道[6]。本研究调查了2011—2016年某院住院患者粪肠球菌对泰利唑胺的敏感性。尽管该院从未使用过泰利唑胺,但仍发现了泰利唑胺不敏感的粪肠球菌。因此,本研究对泰利唑胺不敏感粪肠球菌的耐药机制、多位点序列分型(multilocus sequence typing,MLST)分布等做了进一步的分析和探讨。
1 材料与方法 1.1 菌株来源收集2011年1月1日—2016年6月30日深圳市南山区人民医院临床分离的粪肠球菌菌株,剔除同一患者相同部位重复分离株。
1.2 菌株鉴定和药敏试验参照第9版《全国临床检验操作规程》,采用CATC肠球菌选择培养基对试验菌株进行分离培养,采用BD Phoenix-100全自动细菌鉴定/药敏系统进行菌株鉴定,以及常用抗菌药物(氨苄西林、红霉素、环丙沙星、利福平、四环素、呋喃西林、万古霉素和利奈唑胺)的敏感性检测。采用2016年版美国临床实验室标准化协会(CLSI)推荐的微量肉汤稀释法[7]检测试验菌株对泰利唑胺的最低抑菌浓度(MIC),并再次确认氨苄西林、万古霉素和利奈唑胺的药敏结果,以粪肠球菌ATCC 29212作为质控菌株。泰利唑胺药敏结果判断标准:敏感为MIC≤0.5 mg/L,不敏感为MIC>0.5 mg/L。
1.3 DNA提取和耐药基因检测耐药基因23S rRNA V区基因、耐药基因cfr、cfr(B)及optrA引物由北京六合华大基因公司合成[8-11]。根据试剂盒说明书提取菌株基因组DNA,-20 ℃保存备用。采用聚合酶链式反应(PCR)方法检测耐药基因,PCR体系(50 μL):Dream Taq Green PCR Master Mix(2×) 25 μL,上、下游引物各1 μL,DNA 2 μL,加dd H2O补至50 μL。PCR反应条件:95℃预变性3 min;95℃变性30 s,52℃退火30 s,72℃延伸1 min,共30个循环;72℃ 10 min,PCR产物于4℃保存。PCR反应产物均用1%的琼脂糖凝胶电泳,根据有无阳性扩增产物及目的基因片段长度进行判断分析。
1.4 MLST采用MLST分析肠球菌分离株的分子分型,参考粪肠球菌MLST数据库(http://efaecalis.mlst.net/)提供的分型方案,7个管家基因(gdh、gyd、pstS、gki、aroE、xpt和yqiL)[12-13]引物由北京六合华大基因公司合成。PCR反应体系为50 μL,内含2×ExTaq Mix 25 μL,10 μmol/L的正、反向引物各2.5 μL,模板DNA 2.0 μL,加ddH2O补至50 μL。PCR反应程序:94℃ 5 min变性,94℃ 30 s→52℃ 30 s→72℃ 60 s,30个循环,72℃延伸7 min。PCR产物经纯化后,进行双向测序。序列分析用SeqMan(DNAStar Inc.,Madison,WI,USA)软件对等位基因序列进行拼接和校正,将整理好的序列提交至MLST数据库,确定每个管家基因的序列号。7个管家基因序列号的组合即为该菌株序列型别(ST)。对于未能完全匹配的序列和ST,重复3次确认无误后记为NT。应用eBURST V3软件确定分离菌株与数据库其他菌株的关系,构建克隆群时以2个等位基因的差异为定义克隆群的标准。
1.5 统计学分析应用SPSS 19.0软件进行数据分析,率的比较采用卡方检验或Fisher确切概率法。P≤0.05为差异有统计学意义。
2 结果 2.1 细菌来源及患者分布共收集粪肠球菌289株,来源科室主要为外科(57.4%),其中泌尿外科检出最多(64株),其次为肝胆外科及重症监护病房(ICU),均检出31株。标本来源主要为中段尿(126株,43.6%),其次是伤口分泌物(60株,20.8%)、穿刺液(33株,11.4%)、血(30株,10.34%)、无菌体液(10株,3.5%)、呼吸道分泌物(9株,3.1%)、组织(6株,2.1%)、导管(2株,0.7%)。
2.2 粪肠球菌对泰利唑胺体外敏感性检出的粪肠球菌中大多对常用的多种抗菌药物耐药。其中对红霉素、利福平和四环素耐药最为突出,耐药率均在50%以上。泰利唑胺对粪肠球菌的敏感率为94.1%。粪肠球菌对氨苄西林、呋喃西林和万古霉素仍显示出很高的敏感性(敏感率为97.9%~99.7%)。而且,几乎所有的泰利唑胺非敏感粪肠球菌对氨苄西林、呋喃西林和万古霉素都是敏感的。粪肠球菌对常用抗菌药物的敏感性见表 1。
| 表 1 粪肠球菌对常用抗菌药物的敏感性 Table 1 Susceptibility of E. faecalis to commonly used antimicrobial agents |
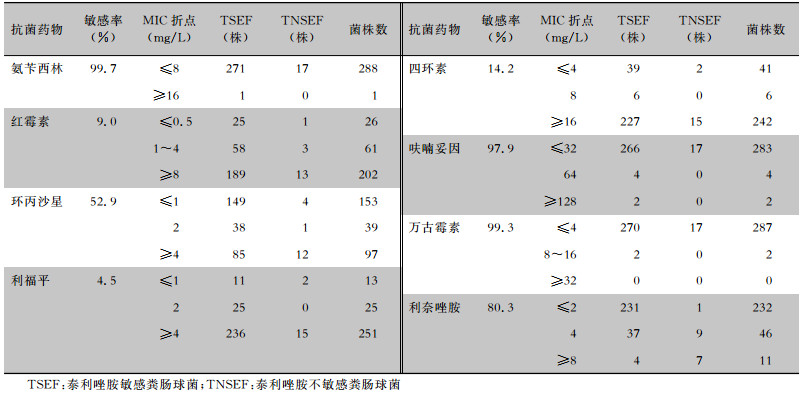
|
采用MLST技术对289株粪肠球菌进行同源性分析,共分为47个ST型(其中有48株的ST型与粪肠球菌MLST数据库不匹配,标记为NT)。优势ST分型为ST16和ST179,分别占29.1%(84株)和24.9%(72株),见图 1。在泰利唑胺不敏感粪肠球菌中,ST16的比例高于ST179(P<0.05),见图 2。
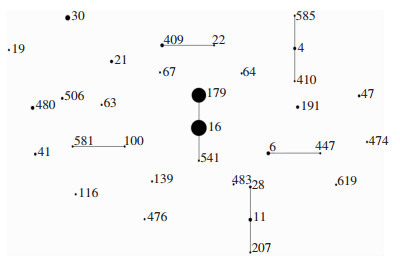 |
| 图 1 289株粪肠球菌MLST分布情况 Figure 1 Distribution of MLST of 289 E. faecalis strains |
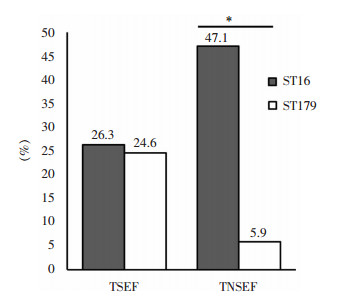 |
| TSEF:泰利唑胺敏感粪肠球菌;TNSEF:泰利唑胺不敏感粪肠球菌;*:差异有统计学意义(P<0.05) 图 2 泰利唑胺敏感及不敏感粪肠球菌菌株中ST16与ST179分型菌株占比的比较 Figure 2 Comparison of the proportion of ST16 and ST179 strains in TZD-susceptible and TZD-non-susceptible E. faecalis strains |
238株粪肠球菌中均未发现23S rRNA V区突变,也未检出cfr和cfr(B)基因。共检出11株携带optrA基因的粪肠球菌,其中9株对泰利唑胺不敏感。
3 讨论肠球菌是医院感染的主要病原体之一,因其具有获得编码抗生素耐药性基因的能力,且对多种抗菌药物天然耐药,给临床治疗造成许多困难。本研究中泰利唑胺对肠球菌MIC值与利奈唑胺相比更低,提示泰利唑胺具有更好的活性。与欧美国家的报道[14]不同,本研究中并未检出耐万古霉素粪肠球菌,而且几乎所有粪肠球菌对氨苄西林、呋喃妥因、万古霉素、泰利唑胺均有良好的敏感性。值得一提的是,本研究中检出的所有利奈唑胺或泰利唑胺不敏感的粪肠球菌菌株均对氨苄西林、万古霉素和呋喃妥因敏感,因此,在对粪肠球菌感染的治疗中这三种药相较于更昂贵的利奈唑胺或泰利唑胺是更好的选择。
既往研究[15]显示,粪肠球菌中ST16在医院感染中更为多见,而ST179在社区感染中较为多见。在本研究中,粪肠球菌的主要MLST为ST16和ST179,分型为ST16的粪肠球菌对泰利唑胺敏感率低于分型为ST179的菌株。提示社区感染的粪肠球菌比医院感染的菌株具有更高的泰利唑胺敏感性,但仍需进一步流行病学调查以证实。
有研究[16]提示23S rRNA V区突变可能是泰利唑胺耐药的主要机制,特别是2576核苷酸从A到T的突变是临床肠球菌中最常见的突变,但在本研究中并未发现23S rRNA V区突变菌株。本研究检出的17株泰利唑胺不敏感的粪肠球菌中有11株携带外源性质粒介导的耐药基因optrA(编码ABC家族F亚家族蛋白)。结合既往曾有报道[11]显示,携带optrA基因的粪肠球菌对泰利唑胺具有耐药性,推测携带optrA基因是本研究中粪肠球菌对泰利唑胺不敏感的主要机制。但另外6株泰利唑胺不敏感的粪肠球菌并未检出相关耐药基因,是否存在其他未知机制有待进一步研究。
多重耐药粪肠球菌的出现给临床治疗带来了很大的挑战,恶唑烷酮类药物的合理使用和适当的医院感染防控非常重要。在本研究中,泰利唑胺对携带optrA基因的粪肠球菌菌株未显示出明显的优势。随着利奈唑胺使用的日益增长,第二代恶唑烷酮类药物泰利唑胺不敏感粪肠球菌菌株的出现也有逐渐增加的风险。由于本研究为单中心数据,且该院尚未使用泰利唑胺,仍需要更多的临床试验进一步验证泰利唑胺对粪肠球菌感染的疗效。
| [1] |
Bourgeois-Nicolaos N, Massias L, Couson B, et al. Dose dependence of emergence of resistance to linezolid in Enterococcus faecalis in vivo[J]. J Infect Dis, 2007, 195(10): 1480-1488. DOI:10.1086/513876 |
| [2] |
蒲彰雅, 余治健, 陈重, 等. 利奈唑胺耐药粪肠球菌感染患者临床分离株与定植株同源性分析[J]. 中国感染控制杂志, 2017, 16(4): 343-345, 350. |
| [3] |
Rybak JM, Roberts K. Tedizolid phosphate: a next generation oxazolidinone[J]. Infect Dis Ther, 2015, 4(1): 1-14. DOI:10.1007/s40121-015-0060-3 |
| [4] |
Moellering RC Jr. Tedizolid: a novel oxazolidinone for gram-positive infections[J]. Clin Infect Dis, 2014, 58(1): S1-S3. |
| [5] |
Locke JB, Zurenko GE, Shaw KJ, et al. Tedizolid for the management of human infections: in vitro characteristics[J]. Clin Infect Dis, 2014, 58(Suppl 1): S35-S42. |
| [6] |
Klupp EM, Both A, Belmar Campos C, et al. Tedizolid susceptibility in linezolid- and vancomycin-resistant Enterococcus faecium isolates[J]. Eur J Clin Microbiol Infect Dis, 2016, 35(12): 1957-1961. DOI:10.1007/s10096-016-2747-0 |
| [7] |
Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing. 25th informational supplement, M100-S26[S]. Wayne, PA: CLSI, 2016.
|
| [8] |
Ntokou E, Stathopoulos C, Kristo I, et al. Intensive care unit dissemination of multiple clones of linezolid-resistant Enterococcus faecalis and Enterococcus faecium[J]. J Antimicrob Chemother, 2012, 67(8): 1819-1823. DOI:10.1093/jac/dks146 |
| [9] |
Liu Y, WangY, Wu C, et al. First report of the multidrug resistance gene cfr in Enterococcus faecalis of animal origin[J]. Antimicrob Agents Chemother, 2012, 56(3): 1650-1654. DOI:10.1128/AAC.06091-11 |
| [10] |
Deshpande LM, Ashcraft DS, Kahn HP, et al. Detection of a new cfr-like gene, cfr(B), in Enterococcus faecium isolates recovered from human specimens in the United States as part of the SENTRY Antimicrobial Surveillance Program[J]. Antimicrob Agents Chemother, 2015, 59(10): 6256-6261. DOI:10.1128/AAC.01473-15 |
| [11] |
Wang Y, Lv Y, Cai J, et al. A novel gene, optrA, that confers transferable resistance to oxazolidinones and phenicols and its presence in Enterococcus faecalis and Enterococcus faecium of human and animal origin[J]. J Antimicrob Chemother, 2015, 70(8): 2182-2190. DOI:10.1093/jac/dkv116 |
| [12] |
Bai B, Hu K, Li H, et al. Effect of tedizolid on clinical Enterococcus isolates: in vitro activity, distribution of virulence factor, resistance genes and multilocus sequence typing[J]. FEMS Microbiol Lett, 2018, 365(3). DOI:10.1093/femsle/fnx284 |
| [13] |
Nallapareddy SR, Duh RW, Singh KV, et al. Molecular typing of selected Enterococcus faecalis isolates: pilot study using multilocus sequence typing and pulsed-field gel electrophoresis[J]. J Clin Microbiol, 2002, 40(3): 868-876. DOI:10.1128/JCM.40.3.868-876.2002 |
| [14] |
O'Driscoll T, Crank CW. Vancomycin-resistant enterococcal infections: epidemiology, clinical manifestations, and optimal management[J]. Infect Drug Resist, 2015, 8: 217-230. |
| [15] |
Zheng JX, Bai B, Lin ZW, et al. Characterization of biofilm formation by Enterococcus faecalis isolates derived from urinary tract infections in China[J]. J Med Microbiol, 2017, 60: 60-67. |
| [16] |
Gawryszewska I, Żabicka D, Hryniewicz W, et al. Linezolid-resistant enterococci in Polish hospitals: species, clonality and determinants of linezolid resistance[J]. Eur J Clin Microbiol Infect Dis, 2017, 36(7): 1279-1286. DOI:10.1007/s10096-017-2934-7 |



