感染性心内膜炎(infective endocarditis, IE)是一种以心脏瓣膜受累为标志的感染性疾病, 其发病率约为3~10例/10万人[1]。未得到正确、及时的治疗, 超过1/3的IE患者会在诊断后的第一年内死亡[2]。IE患者的预后高度依赖早期、精准的抗菌药物治疗, 因此, 对引起IE病原菌的鉴定至关重要。目前, IE的诊断和抗菌药物治疗主要依赖于血培养及基于培养菌株的药敏试验结果。然而, 采集血培养前使用抗菌药物, 某些苛养菌或者常规方法难于培养的微生物引起的IE, 血培养常呈现阴性的结果[3-4]。在这种情况下, 可能会出现误诊或抗菌药物使用不合理的情况, 严重影响IE患者的预后。近十几年来, 分子生物学技术发展迅速, 基于细菌保守序列16S rRNA聚合酶链反应(PCR)技术已经被用于心脏换瓣术后摘除的瓣膜组织中病原体的检测, 其灵敏度优于传统的培养方法, 对常规可培养或者苛养菌也有较强的检测能力。然而, 基于16S rRNA的PCR技术存在天然的缺陷, 如不能检测真菌, 对混合感染也是无能为力[5-6]。近几年来, 以二代测序(next-generation sequencing, NGS)为代表的宏基因组学已经被用于直接检测临床标本中的病原菌[7]。评估NGS技术直接检测心脏瓣膜病原菌的研究在国内外较少见, 本研究以改良DUKE标准为"金标准", 直接提取心脏瓣膜组织中的DNA, 旨在评价NGS技术直接检测心脏瓣膜病原菌的能力, 为IE的诊断和术后抗菌治疗提供帮助。
1 材料与方法 1.1 标本来源及前处理标本来自中国医学科学院阜外医院2017年4月-2018年2月经心脏瓣膜置换术或者心脏瓣膜成形术后摘除的瓣膜组织, 其中28例来自成人外科中心, 2例来自小儿外科中心。31例标本中, 根据IE诊断的"金标准"改良Duke标准, 20例为确诊IE(D.IE)患者, 4例为可能IE(P.IE)患者, 阴性对照为剩余的7例排除IE(re- jected IE)的心脏瓣膜病患者。实验室收到摘除的瓣膜组织后, 立即在生物安全柜里用无菌的眼科手术剪把组织剪碎并随机分为两份, 一份用于常规增菌培养, 另一份(约35 mg)放置于-80℃冰箱冷冻保存, 供NGS测序及Sanger测序验证使用。
1.2 主要仪器与试剂哥伦比亚血平板、麦康凯平板、脑心浸液(赛默飞世尔生物化学公司), Heal Force HF90型CO2孵箱(上海力申科学仪器公司), -80℃低温冰箱(日本三洋公司), 高压蒸汽灭菌器(山东新华公司), BACTEK FX400全自动血培养仪及配套血培养瓶(美国BD公司), Vitek 2 Compact全自动微生物分析仪及配套鉴定药敏卡, VITEK MSTM质谱仪(法国生物梅里埃公司), QIAamp DNA Mini Kit(德国Qiagen公司), Premix TaqTM、DL2000 marker(大连TaKaRa公司), 琼脂糖凝胶(西班牙Biowest公司), GTQ-Cy- cler 96扩增仪(德国海恩公司), DYY-6C型电泳仪(北京六一仪器厂), GIS-2010凝胶图像分析仪(上海天能公司), 引物及扩增产物测序由中美泰和生物公司完成, 二代测序及生物信息学分析由深圳华大基因研究所完成。
1.3 瓣膜培养瓣膜组织在培养前, 先用无菌的眼科手术剪尽可能的剪碎, 然后加入5 mL脑心浸液增菌, 放置35℃ CO2孵箱中培养7 d。每日评估标本的增菌情况, 若有浑浊及时转种于哥伦比亚血平板、巧克力平板及麦康凯平板(赛默飞世尔公司)。未出现浑浊的标本, 在第7天也转种于血平板、巧克力平板及麦康凯平板。培养出的微生物使用MAL- DI-TOF质谱仪(法国VITEK公司)进行鉴定, 使用VITEK 2 Compact全自动药敏分析仪进行药敏试验(法国VITEK公司)。
1.4 血培养血培养的采集参照美国临床实验室标准化协会(CLSI)的血培养临床实践指南(Princi- ples and procedures for blood cultures; Approved Guideline:M47A)。对疑似IE的发热患者, 在使用抗菌药物之前或者下次使用抗菌药物之前(对已使用抗菌药物的患者)从不同穿刺位点平均采集3套血培养, 采集的血液被立即注入需氧瓶和厌氧瓶(成人每瓶8~10 mL血液, 儿童每瓶1~3 mL血液)。血培养瓶在2 h内被放入全自动血培养仪(美国BD公司)培养7 d, 对培养阳性的标本及时转种到哥伦比亚血平板、巧克力平板及麦康凯平板。微生物的鉴定和药敏同瓣膜组织培养。
1.5 NGS及生信分析冷冻的标本恢复常温后, 用无菌的眼科手术剪剪碎。称取约25 mg的瓣膜组织加入蛋白酶K, 消化过夜后使用QIAamp DNA Mini Kit提取DNA, 整个操作严格遵照厂家的说明书进行。提取的DNA经超声波处理被破碎成200~300 bp的片段, 随后开始文库构建和测序, 具体如下:使用Agilent 2100 Bioanalyzer质控文库插入片段大小, Qubit dsDNA HS Assay Kit (Thermo Fisher Scientific Inc.)质控DNA文库浓度, 经环化形成单链环形结构。环化后的文库经滚环复制生成DNA纳米球。制备好的DNA纳米球加载至测序芯片, 使用BGISEQ-500进行测序。测序数据下机后去除低质量的和长度小于35 bp的数据, 以获得高质量的数据。通过BWA比对, 将高质量数据中与人参考基因组序列相同的数据去除, 剩下的数据用SNAP等软件进一步分析, 去除低复杂度reads后与NCBI的NT数据库比对, 并将比对结果进行Taxonomy注释、分类, 以及统计作图。
1.6 Sanger测序验证血培养、瓣膜培养和/或NGS检测结果为阳性的标本进行Sanger测序验证。Sanger测序所用的DNA同NGS。引物由中美泰和生物公司合成。全序列引物为27f(序列为: 5'-AGAGTTTGATCCTGGCTCAG-3')和1492r (序列为:5'-GGTTACCTTGTTACGACTT-3'), 扩增产物长度约1 500 bp。扩增体系为50 μL: 25 μL Premix TaqTM, 10 μmol/L上、下游引物各2 μL, 2 μL DNA模板, 以灭菌双蒸水补足体积。反应条件为:96℃ 150 s; 96℃ 30 s, 55℃ 30 s, 72℃ 90 s, 共30个循环; 72℃ 7 min。扩增产物经10 g/L琼脂糖凝胶电泳观察, 阳性扩增产物送中美泰和公司进行测序, 测序结果在NCBI Blast(http://blast.ncbi.nlm.nih.gov/B-last.cgi?PROGRAM5blastn &PAGE_TYPE5BlastSear-ch&LINK_LOC5blasth ome)在线比对, 以相似度最高者为准。比对相似度至少97%方可认为同一菌种, 不能归到种水平的只归到属水平。
1.7 统计学分析应用MedCalc统计软件进行分析, 采用灵敏度、特异度、阳性预测值、阴性预测值表示。
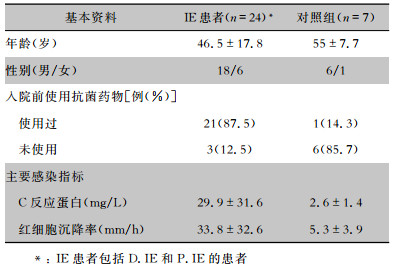
2 结果 2.1 患者基本资料24例IE患者中, 根据改良Duke标准, 20例为D.IE, 4例为P.IE患者, 其中男性占75.0%, 平均年龄46.5岁。87.5%的IE患者在入院前使用过抗菌药物, 非IE患者仅占14.3%。IE患者的C反应蛋白和红细胞沉降率均高于非IE患者(均P<0.05)。见表 1。IE患者瓣膜赘生物分布为主动脉瓣12例(50.0%), 二尖瓣4例(16.7%), 主动脉瓣+二尖瓣3例(12.5%)、主动脉瓣+三尖瓣2例(8.3%), 未见3例(12.5%)。对照组均未见瓣膜赘生物。
| 表 1 IE患者及对照组的基本资料 Table 1 Basic data of IE patients and control group |
|
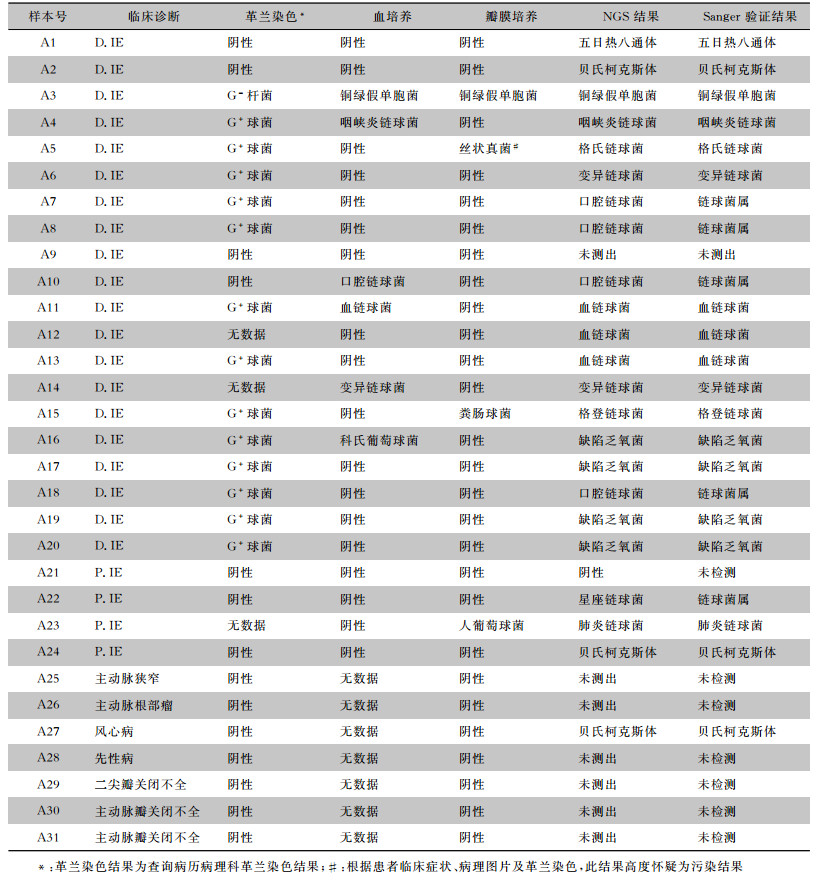
31例患者中有3例无革兰染色结果。在18例确诊IE患者中, 14例革兰染色阳性, 阳性率为77.8%, 其中13例(92.9%)为革兰阳性(G+)球菌, 1例为革兰阴性(G-)杆菌。见表 2。
| 表 2 31例患者临床诊断及主要实验室检测结果 Table 2 Clinical diagnosis and main laboratory test results of 31 patients |
|
20例D.IE患者中, 6例患者血培养阳性, 阳性率为30.0%, 其中4株为草绿色链球菌, 铜绿假单胞菌和科氏葡萄球菌各1株; 在D.IE患者中, 3例瓣膜培养阳性, 分别为铜绿假单胞菌、丝状真菌和粪肠球菌, 阳性率为10.0%(A5样本从临床症状、病理图片及革兰染色结果看, 丝状真菌高度怀疑为污染菌, 未计入阳性率统计)。见表 2。
2.4 NGS结果20例D.IE患者中, 19例NGS检测阳性, 阳性率为95.0%, 其中链球菌16株, 五日热八通体、贝氏柯克斯体、铜绿假单胞菌各1株。4例P.IE患者中, 3例NGS阳性, 其中链球菌2株, 贝氏柯克斯体1株。排除IE的7例病例中, NGS检测阳性1例, 为贝氏柯克斯体。所有NGS结果与Sanger测序验证的属一致率为100.0%, 种一致率为79.2%。
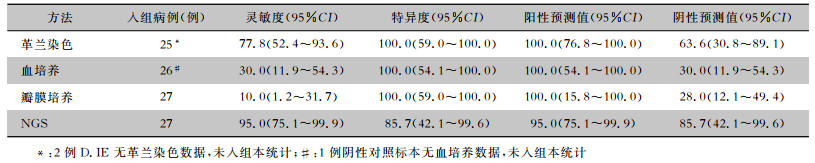
2.5 4种方法的诊断性能革兰染色、血培养、瓣膜培养、NGS诊断IE的灵敏度分别为77.8%、30.0%、10.0%及95.0%, 特异度分别为100.0%、100.0%、100.0%及85.7%, 阳性预测值分别为100.0%、100.0%、100.0%及95.0%, 阴性预测值分别为63.6%、30.0%、28.0%及85.7%。见表 3。
| 表 3 不同方法诊断IE的诊断效能(%) Table 3 Diagnostic efficacy of different diagnostic methods for IE(%) |
|
在临床实践中, IE的早期诊断及针对性治疗一直是个难题。通过查询病历及电话回访, 发现高达87.5%的患者收入本院前在下级医院或者家中服用过抗菌药物, 可能是IE早期症状与普通感染性疾病(如感冒)相似, 造成临床医生或者患者误判。采集血培养前使用抗菌药物极大地影响了IE患者血培养的阳性率, 可能是本院IE血培养阳性率低于国际平均水平的主要原因[8]。这种状况更可突显NGS技术在检测IE病原菌中的优势。从理论上讲, 只要样本中还存在病原菌的核酸物质, NGS技术就可以从临床样本中直接检测出包括细菌、真菌、病毒、寄生虫等所有类型的病原体[9]。当然, 这也预示着NGS检测出的病原体有可能是残留的已死亡的病原体[10-11]。即使检出的是已死亡的病原体, 对于IE患者瓣膜置换术后针对性的预防治疗也至关重要。IE患者瓣膜置换术后若不接受系统的、规范的抗菌治疗就会存在很大的复发风险。在研究中还发现相比血液、胸腔积液和尿液等无菌体液[12-13], 心脏瓣膜组织中病原体的核酸更为富集, 本研究中的很多样本病原体的reads数能达到上千甚至十万个数量级。
本研究中1例样本(A5)的瓣膜培养结果为丝状真菌, 而革兰染色、NGS结果及Sanger测序均为G+球菌, 临床表现也不支持真菌感染, 此结果应为假阳性, 很可能是由实验室培养或者转种过程中污染导致的。样本A16、A23的培养结果均为凝固酶阴性葡萄球菌, NGS和Sanger测序是一致的, 均为链球菌。凝固酶阴性葡萄球菌虽然也可引起IE, 但是作为皮肤表面的定植菌, 链球菌更容易被认为是IE的病原菌[14]。1例D.IE(A9)的病例革兰染色、血培养、瓣膜培养、NGS及Sanger测序却均为阴性, 可能与患者处于炎症恢复期和/或者样本取材的部分不合理有关。1例D.IE的病例(A15), 瓣膜培养经VITEK 2 Compact鉴定药敏仪和VITEK MS质谱仪双重鉴定均为粪肠球菌, NGS和Sanger验证却均为格登链球菌, 可能与肠球菌、链球菌的基因序列存在较高同源性有关(早期的细菌学分类将肠球菌归为D群链球菌)[15]。1例阴性对照样本(A27), 临床诊断为风心病, 血培养、瓣膜培养均为阴性, 但是NGS检测出贝氏柯克斯体(907条reads)。通过查询病历, 与病理科医生和临床医生深入探讨, 一致认为此患者可能为风心病引起的IE患者, 临床存在漏诊的可能。如果此病例为真阳性的话, 本研究计算的NGS检测病原菌的灵敏度和特异度将会更高。
总而言之, 相比传统培养方法, NGS在诊断IE时具有很多优势[16], 具体如下:(1)NGS检测IE患者病原体的灵敏度远高于传统培养方法, 尤其是对难于培养的苛养菌。本研究中NGS检测的苛养菌占总数的34.8%(包括五日热八通体1例、贝氏柯克斯体3例、缺陷乏氧菌4例)。随着NGS技术在检测IE病原体上的广泛应用, 更多未知的病原体可能会被检出, 甚至会颠覆人们对IE病原谱的传统认识。(2)随着NGS技术的成熟, NGS检测病原体的周期将会缩短至2 d, 远低于传统培养所需的5~ 7 d, 甚至是更长时间。(3)NGS技术能够检测包括细菌、真菌、病毒、寄生虫在内的所有病原体, 克服了基于16S rRNA的传统PCR方法只能检测细菌, 对真菌或者混合感染无能为力的缺点。国外文献[17]报道, 约2%的IE是由真菌引起的。(4)随着大数据的应用和生物信息学的发展, NGS技术在检测病原体的同时还能检测耐药基因, 对于培养结果阴性的IE患者尤为重要, 因为NGS不仅可以用于IE的诊断, 还可对IE患者的抗菌治疗提供帮助。当然, NGS作为一项新技术也存在很多局限性, 如NGS的生信分析缺乏统一的标准, 不能确定检测的细菌是否已经死亡, 以及测序结果与疾病之间的关系尚不明确等问题。相信随着国家相关标准的形成, NGS完整基因数据库的建立及NGS技术的进一步发展, 这些不足或许会得到改善或者被克服, NGS在IE的诊断和抗菌治疗上将发挥更大的作用。
| [1] |
Thuny F, Grisoli D, Collart F, et al. Management of infective endocarditis:challenges and perspectives[J]. The Lancet, 2012, 379(9819): 965-975. DOI:10.1016/S0140-6736(11)60755-1 |
| [2] |
Thuny F, Di Salvo G, Belliard O, et al. Risk of embolism and deathin in fective endocarditis:prognostic value of echocardio-graphy:a prospective multicenter study[J]. Circulation, 2005, 112(1): 69-75. DOI:10.1161/CIRCULATIONAHA.104.493155 |
| [3] |
Houpikian P, Raoult D. Blood culture-negative endocarditis in a reference center:etiologic diagnosis of 348 cases[J]. Medi-cine (Baltimore), 2005, 84(3): 162-173. DOI:10.1097/01.md.0000165658.82869.17 |
| [4] |
Poesen K, Pottel H, Colaert J, et al. Epidemiology of infec-tive endocarditis in a large Belgian non-referral hospital[J]. Acta Clin Belg, 2014, 69(3): 183-190. DOI:10.1179/0001551214Z.00000000046 |
| [5] |
Peeters B, Herijgers P, Beuselinck K, et al. Comparison of PCR-electrospray ionization mass spectrometry with 16S rRNA PCR and amplicon sequencing for detection of bacteria in excised heart valves[J]. J Clin Microbiol, 2016, 54(11): 2825-2831. DOI:10.1128/JCM.01240-16 |
| [6] |
Shrestha NK, Ledtke CS, Wang H, et al. Heart valve culture and sequencing to identify the infective endocarditis pathogen in surgically treated patients[J]. Ann Thorac Surg, 2015, 99(1): 33-37. DOI:10.1016/j.athoracsur.2014.07.028 |
| [7] |
Capobianchi MR, Giombini E, Rozera G. Next-generation se-quencing technology in clinical virology[J]. Clin Microbiol In-fect, 2013, 19(1): 15-22. DOI:10.1111/1469-0691.12056 |
| [8] |
Marín M, Muñoz P, Sánchez M, et al. Molecular diagnosis of infective endocarditis by real-time broad-range polymerase chain reaction (PCR) and sequencing directly from heart valve tissue[J]. Medicine (Baltimore), 2007, 86(4): 195-202. DOI:10.1097/MD.0b013e31811f44ec |
| [9] |
Fukui Y, Aoki K, Okuma S, et al. Metagenomic analysis for detecting pathogens in culture-negative infective endocarditis[J]. J Infect Chemother, 2015, 21(12): 882-884. DOI:10.1016/j.jiac.2015.08.007 |
| [10] |
Hasman H, Saputra D, Sicheritz-Ponten T, et al. Rapid whole-genome sequencing for detection and characterization of microorganisms directly from clinical samples[J]. J Clin Microbiol, 2014, 2(1): 139-146. |
| [11] |
Köser CU, Ellington MJ, Cartwright EJ, et al. Routine use of microbial whole genome sequencing in diagnostic and public health microbiology[J]. PLoS Pathog, 2012, 8(8): e1002824. DOI:10.1371/journal.ppat.1002824 |
| [12] |
Guan H, Shen A, Lv X, et al. Detection of virus in CSF from the cases with meningoencephalitis by next-generation sequencing[J]. J Neurovirol, 2016, 22(2): 240-245. DOI:10.1007/s13365-015-0390-7 |
| [13] |
Long Y, Zhang Y, Gong Y, et al. Diagnosis of sepsis with cell-free DNA by next-generation sequencing technology in ICU patients[J]. Arch Med Res, 2016, 47(5): 365-371. DOI:10.1016/j.arcmed.2016.08.004 |
| [14] |
黄俊, 刘甜, 蒋祖勋, 等. 感染性心内膜炎730例病原菌构成及药敏分析[J]. 岭南心血管病杂志, 2013, 19(5): 568-571. DOI:10.3969/j.issn.1007-9688.2013.05.012 |
| [15] |
Garrity GM, Bell JA, Lilburn TG.伯杰氏细菌鉴定手册: 第9版[M].中国科学院微生物研究所"伯杰氏细菌手册"翻译组, 译.北京: 科学出版社, 2004.
|
| [16] |
Fukui Y, Aoki K, Okuma S, et al. Metagenomic analysis for detecting pathogens in culture-negative infective endocarditis[J]. J Infect Chemother, 2015, 21(12): 882-884. DOI:10.1016/j.jiac.2015.08.007 |
| [17] |
Murdoch DR, Corey GR, Hoen B, et al. Clinical presentation, etiology, and outcome of infective endocarditis in the 21st century:the international collaboration on endocarditis-prospective cohort study[J]. Arch Intern Med, 2009, 169(5): 463-473. DOI:10.1001/archinternmed.2008.603 |



