2. 成都医学院药学院, 四川 成都 610500;
3. 南充市中心医院药学部, 四川 南充 637000
2. School of Pharmacy, Chengdu Medical College, Chengdu 610500, China;
3. Department of Pharmacy, Nanchong Central Hospital, Nanchong 637000, China
鲍曼不动杆菌(Acinetobacter baumannii) 是医院感染中常见的革兰阴性条件致病菌,是全球6大超级细菌-ESKAPE (Enterococcus faecium、Staphy-lococcus aureus、Klebsiella pneumoniae、Acinetobacter baumannii、Pseudomonas aeruginosa、Entero- bacter species)的成员之一[1-2]。近年来,随着抗菌药物的广泛使用,鲍曼不动杆菌的耐药率不断上升,出现多重耐药、泛耐药,甚至是全耐药。2017年世界卫生组织(WHO)发布首份抗生素耐药“重点病原体”清单,列出了对人类健康构成最大威胁的急需开发新抗生素的12种重点耐药细菌,耐碳青霉烯类鲍曼不动杆菌是其中之首,它的出现给抗感染治疗带来重重困难[3-5]。鲍曼不动杆菌的耐药机制主要包括药物灭活酶的产生、药物外排泵的表达、药物作用靶点改变、生物被膜的形成等[6-8]。近年研究发现,细菌在抗菌药物等外界环境压力下,会产生特异的调控性非编码RNAs(ncRNAs),在伴侣蛋白Hfq的作用下,与靶基因的mRNA互补配对或通过其构像的变化调控相关耐药基因(药物外排泵、生物被膜形成、毒力因子、细胞膜通透性、细胞壁的合成)的表达水平,从而帮助细菌适应外界环境压力[8-11]。此文针对鲍曼不动杆菌的调控性ncRNAs对耐药性的调控作用,以及其作为药物靶点的研究进展进行综述。
1 调控性ncRNAs的分类及作用机制细菌调控性ncRNAs是一类广泛存在于原核生物基因组中能被转录但不编码蛋白质的长度为50~300 nt的RNA分子。细菌ncRNAs主要位于基因间区,某些还可位于编码基因的5’端非翻译区(5’UTR)和3’端非翻译区(3’UTR),能折叠成稳定的茎环或发夹结构,与靶基因的mRNA互补或直接与靶蛋白结合, 导致mRNA或蛋白的功能衰减或丧失。根据调控ncRNAs的转录和调控方式不同,本文将其分为3类即小RNAs(sRNAs)、5’UTR或3’UTR调控元件、成簇规律间隔短回文重复序列(CRISPR)[12-14]。
sRNAs多数位于基因间隔区,自身拥有独立转录的启动子和终止子,转录开始于一段能折叠成稳定茎环结构的序列,终止于Rho不依赖的转录终止子,以顺式或反式作用调节靶基因的表达[15-17]。靶基因的5’UTR或者3’UTR调控元件,以顺式作用调控靶基因的表达[18],核糖体停靠机制以及核糖开关通过靶基因的5’UTR或者3’UTR的茎环或发夹结构构像改变使核糖体结合位点和起始密码子暴露或隐藏,从而调控细菌对抗生素的敏感性[19-22]。CRISPR序列由众多短而保守且含有回文序列的重复序列区(repeat,转录后可以形成具有稳定发卡结构的tracRNA)和间隔区(spacer,被细菌俘获的部分外源DNA序列,转录后可形成成熟的crRNA)组成。当这些外源遗传物质再次入侵时,crRNA、tracRNA形成sgRNA复合体,与外源遗传物质互补配对并引导cas剪切蛋白破坏外源遗传物质[23-24]。ncRNAs不需要翻译,在转录后水平发挥作用,作用速度比蛋白产生速度快,能更快地调节诸如群体感应、生物被膜、毒力因子、药物外排泵等相关耐药基因的表达[25],是细菌对抗菌药物产生耐药的重要参与者,因此调控性ncRNAs有希望成为对抗细菌耐药的新靶点。
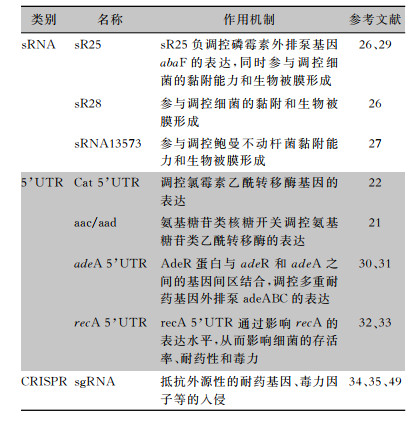
2 调控性ncRNAs对鲍曼不动杆菌耐药性及毒力的调控作用 2.1 调控性sRNAs对鲍曼不动杆菌生物被膜形成和黏附能力的调控作用生物被膜是鲍曼不动杆菌对抗菌药物高度耐药的重要原因,也是其重要的毒力因子,而调控性sRNAs可以通过调控细菌生物被膜的形成从而影响细菌耐药性和毒力。付竹青[26]研究发现,在鲍曼不动杆菌17978中AbsR25和AbsR28的缺失不仅能抑制其对细胞的黏附,还能显著减少细菌生物被膜的形成以及提高大蜡螟的存活率。研究者发现,在鲍曼不动杆菌17978中有9个sRNAs只在生物被膜状态下高表达,而其中sRNA13573的高表达又可增强鲍曼不动杆菌生物被膜形成的能力和对人肺上皮细胞A549的黏附能力[27]。在鲍曼不动杆菌中,sRNAs作用于靶基因时,常需要分子伴侣Hfq的协助,以加快sRNAs与靶mRNA二聚体的形成,Kuo等[28]证明,鲍曼不动杆菌17978在Hfq缺失的情况下生物被膜形成量减少,对细胞的黏附能力也减弱,而且参与生物被膜形成的相关基因,如外膜蛋白ompA、菌毛合成系统csuA/B基因的表达水平也显著下调。
2.2 调控性sRNAs和5’UTR调控元件对鲍曼不动杆菌外排泵的调控作用鲍曼不动杆菌中的药物外排泵种类多而且底物广,其高表达是细菌耐药重要机制之一。研究[29]发现,AbsR25的靶基因是外排泵主要易化家族(MFS)中的一种新型的磷霉素外排泵基因abaF,在不同亚抑菌浓度的磷霉素条件下AbsR25负调控abaF基因的表达。在双组份调控体adeRS与adeABC外排泵操纵子中,adeR与adeA基因之间存在基因间区,而且Marchand等[30]研究显示,adeR与adeA基因间区存在adeA 5’UTR元件。Wen等[31]研究证实,AdeR蛋白与该基因间区中的一对10 bp直接重复序列结合,从而调控adeABC的表达,提示adeR 5’UTR元件在调节adeABC的表达中起着关键作用。
2.3 5’UTR调控元件对鲍曼不动杆菌耐药酶的调控作用在氯霉素的诱导下,氯霉素乙酰转移酶cat基因mRNA的5’UTR调控元件通过核糖体停靠机制使其二级结构改变,导致mRNA的核糖体结合位点和起始密码子暴露,使下游cat基因高表达,从而使细菌对氯霉素产生抗性。aac/aad氨基糖苷类抗生素核糖开关在氨基糖苷类抗生素的诱导下,aac/aad基因mRNA的5’UTR调控元件的二级结构改变,导致mRNA的核糖体结合位点和起始密码子暴露,使下游aac/aad基因高表达,介导氨基糖苷类抗生素核糖耐药[21-22]。
2.4 5’UTR调控元件对鲍曼不动杆菌基因重组酶的调控作用RecA是参与基因同源重组和重组修复的主要酶,当鲍曼不动杆菌处于抗菌药物等压力条件下或受到耐药基因入侵导致DNA损伤时,产生的基因间隙会被RecA重组酶修复。研究人员通过构建鲍曼不动杆菌recA基因缺失的菌株发现,细菌对β-内酰胺类、氟喹诺酮类、亚胺培南、复方磺胺甲口恶唑、多粘菌素的敏感性均显著增强,recA基因可在鲍曼不动杆菌的抗菌药物耐药、应答环境压力、毒力因子等方面发挥作用[32]。Ching等[33]研究发现,在recA的5’端存在具有茎环结构的recA 5’UTR元件,破坏recA 5’UTR元件的茎环结构可降低细菌对利福平抗性的获得概率,以及细菌在紫外线诱变损伤情况下的存活率,证实recA 5’UTR元件能稳定recA的转录,从而应对环境压力造成的DNA损伤,以及获取影响耐药等相关基因。
2.5 CRISPR抵抗外源耐药基因对鲍曼不动杆菌的入侵CRISPR的间隔序列包含了部分耐药基因序列,可作为防御外源核酸侵袭的特定“武器”[34]。付恒霞等[35]研究62株临床耐药鲍曼不动杆菌,无CRISPR系统的菌株中β-内酰胺酶、乙酰转移酶、核苷酸转移酶及16sRNA甲基化酶等耐药基因的携带率均高于有CRISPR系统菌株,表明CRISPR一定程度上可以抵御外源耐药对细菌的入侵。
3 靶向调控性ncRNAs的抗感染策略 3.1 Hfq靶点在调控性sRNAs参与的细菌耐药网络中,Hfq发挥着至关重要的作用,Hfq与sRNA结合后可避免sRNA被核糖核酸酶降解,有助于sRNA的稳定,同时促进sRNA-mRNA结合生成环状蛋白质复合物[36-37]。此外,Hfq作为鲍曼不动杆菌对外界环境压力的相关应答物质,对帮助细菌在外界环境压力下生长繁殖具有重要意义[28, 38]。El-Mowafi等[39]发现,RI20这种物质可以抑制Hfq与sRNA的相互作用,从而抑制革兰阴性致病菌毒力基因的表达,提高细菌对H2O2与抗菌药物的敏感性。因此,Hfq抑制剂的研发及运用有望成为对抗细菌耐药的新策略。
| 表 1 调控性ncRANs对鲍曼不动杆菌耐药性或毒力的调控机制 |
|
嘌呤合成是细菌繁殖的必需过程,嘌呤核糖开关广泛存在于细菌当中。鸟嘌呤类似物5,6-三氨基嘧啶-4-酮配体(PC12)可通过结合嘌呤核糖开关抑制金黄色葡萄球菌的生长繁殖[40-41]。类似的核糖开关如黄素单核苷酸(FMN)核糖开关,FMN类似物配体在对抗大肠埃希菌感染时表现出良好的活性[42-44]。核糖开关成为解决日益严重耐药问题的新出口,而这些配体物质与抗菌药物的联合运用也是一种抗感染治疗的新策略。在鲍曼不动杆菌中为了防止配体与诱导耐药基因表达的核糖开关(aac/aad)结合,以期设计产生与核糖开关竞争结合但不改变核糖开关结构,不与耐药基因核糖开关相互作用的新型小分子物质[45]。
3.3 CRISPR/Cas9基因编辑技术在对抗耐药基因方面,改造的CRISPR/Cas9基因编辑技术已成功应用于细菌。Citorik等[46]构建了靶向β-内酰胺酶1(NDM-1)基因的CRISPR/Cas9系统,帮助细菌对抗NDM-1。Kim等[47]运用CRISPR/Cas9系统恢复了产超广谱β-内酰胺酶(ESBLs)细菌对抗菌药物的敏感性。Sun等[48]运用CRISPR/Cas9基因编辑技术成功敲除了大肠埃希菌中粘菌素耐药基因mcr-1,使其恢复了对多粘菌素的敏感性。宁夏医科大学总医院研究人员[49]发明了CRISPR/Cas9系统,成功敲除鲍曼不动杆菌的OXA-23基因,恢复了其对亚胺培南的敏感性。将改造的CRISPR/Cas9系统植入耐药细菌,并破坏相关耐药基因,使耐药菌重新对抗菌药物敏感,也是一种有效的抗感染策略。
4 结论鲍曼不动杆菌中的调控性ncRNAs(sRNAs、5’UTR或3’UTR调控元件、CRISPR)具有稳定的茎环或发夹结构,均具有高度保守性,这种特殊的结构与其调控细菌的耐药性、毒力因子等密切相关[50-52]。ncRNAs在调控鲍曼不动杆菌生物被膜、药物外排泵、药物灭活酶、毒力因子等的表达,以及抵抗外源耐药基因的入侵等方面起着关键的作用,从而影响细菌的耐药性和毒力。因此,ncRNAs极具作为药物靶点的潜在价值,此外针对Hfq、核糖开关等靶点研发新的抗菌物质,以及开发出适用于鲍曼不动杆菌的CRISPR/Cas生物靶向制剂,都是解决鲍曼不动杆菌耐药问题的新思路。但是目前对鲍曼不动杆菌中的调控性ncRNAs的研究还处在初期探索阶段,大量的调控性ncRNAs,以及其靶基因还有待进一步发现,调控性ncRNAs还通过哪些调控方式参与细菌的耐药网络也有待进一步深入研究。近年来,随着筛选、鉴定ncRNAs,以及预测其靶基因等的分子生物信息学方法与技术的发展[11, 53],将会有更多的调控性ncRNAs及其调控细菌耐药的相关作用机制会被发现。
| [1] |
Santajit S, Indrawattana N. Mechanisms of antimicrobial resistance in ESKAPE pathogens[J]. Biomed Res Int, 2016, 2016: 2475067. |
| [2] |
陈佰义, 何礼贤, 胡必杰, 等. 中国鲍曼不动杆菌感染诊治与防控专家共识[J]. 中国医药科学, 2012, 2(8): 3-8. |
| [3] |
Antunes LC, Visca P, Towner KJ. Acinetobacter baumannii: evolution of a global pathogen[J]. Pathog Dis, 2014, 71(3): 292-301. DOI:10.1111/2049-632X.12125 |
| [4] |
Lin F, Xu Y, Chang YW, et al. Molecular characterization of reduced susceptibility to biocides in clinical isolates of Acinetobacter baumannii[J]. Front Microbiol, 2017, 8: 1836. DOI:10.3389/fmicb.2017.01836 |
| [5] |
Tacconelli E, Carrara E, Savoldi A, et al. Discovery, research, and development of new antibiotics: the WHO priority list of antibiotic-resistant bacteria and tuberculosis[J]. Lancet Infect Dis, 2018, 18(3): 318-327. DOI:10.1016/S1473-3099(17)30753-3 |
| [6] |
刘秋萍, 徐凌. 鲍曼不动杆菌耐药机制的研究进展[J]. 中国抗生素杂志, 2018, 43(10): 1179-1187. DOI:10.3969/j.issn.1001-8689.2018.10.004 |
| [7] |
Pakharukova N, Tuittila M, Paavilainen S, et al. Structural basis for Acinetobacter baumannii biofilm formation[J]. Proc Natl Acad Sci U S A, 2018, 115(21): 5558-5563. DOI:10.1073/pnas.1800961115 |
| [8] |
Felden B, Cattoir V. Bacterial adaptation to antibiotics through regulatory RNAs[J]. Antimicrob Agents Chemother, 2018, 62(5): e02503-17. |
| [9] |
Holmqvist E, Wagner EGH. Impact of bacterial sRNAs in stress responses[J]. Biochem Soc Trans, 2017, 45(6): 1203-1212. DOI:10.1042/BST20160363 |
| [10] |
郑薇, 凌保东, 贾旭, 等. 核糖体停靠与抗生素耐药[J]. 中国抗生素杂志, 2014, 39(3): 161-170. DOI:10.3969/j.issn.1001-8689.2014.03.001 |
| [11] |
Kröger C, MacKenzie KD, Alshabib EY, et al. The primary transcriptome, small RNAs and regulation of antimicrobial resistance in Acinetobacter baumannii ATCC 17978[J]. Nucleic Acids Res, 2018, 46(18): 9684-9698. DOI:10.1093/nar/gky603 |
| [12] |
Jia X, Ling BD, Li XZ. Influence of regulatory RNAs on antimicrobial resistance and efflux mechanisms[M]//Li XZ, Elkins C, Zgurskaya H. Efflux-mediated antimicrobial resistance in bacteria. Switzerland: Springer, 2016: 625-648.
|
| [13] |
Repoila F, Darfeuille F. Small regulatory non-coding RNAs in bacteria: physiology and mechanistic aspects[J]. Biol Cell, 2009, 101(2): 117-131. DOI:10.1042/BC20070137 |
| [14] |
徐巧林, 宋艳钊, 郭岩彬. 细菌非编码RNA及其分子伴侣Hfq[J]. 微生物学报, 2018, 58(9): 1511-1520. |
| [15] |
Waters LS, Storz G. Regulatory RNAs in bacteria[J]. Cell, 2009, 136(4): 615-628. DOI:10.1016/j.cell.2009.01.043 |
| [16] |
Aiba H. Mechanism of RNA silencing by Hfq-binding small RNAs[J]. Curr Opin Microbiol, 2007, 10(2): 134-139. DOI:10.1016/j.mib.2007.03.010 |
| [17] |
Steuten B, Schneider S, Wagner R. 6S RNA: recent answers-future questions[J]. Mol Microbiol, 2014, 91(4): 641-648. DOI:10.1111/mmi.12484 |
| [18] |
Naville M, Gautheret D. Transcription attenuation in bacteria: theme and variations[J]. Brief Funct Genomics, 2010, 9(2): 178-189. DOI:10.1093/bfgp/elq008 |
| [19] |
Ramu H, Mankin A, Vazquez-Laslop N. Programmed drug-dependent ribosome stalling[J]. Mol Microbiol, 2009, 71(4): 811-824. DOI:10.1111/j.1365-2958.2008.06576.x |
| [20] |
He WZ, Zhang XH, Zhang J, et al. Riboswitch control of induction of aminoglycoside resistance acetyl and adenyl-transferases[J]. RNA Biol, 2013, 10(8): 1266-1273. DOI:10.4161/rna.25757 |
| [21] |
Jia X, Zhang J, Sun WX, et al. Riboswitch control of aminogly-coside antibiotic resistance[J]. Cell, 2013, 152(1-2): 68-81. DOI:10.1016/j.cell.2012.12.019 |
| [22] |
Schwarz S, Kehrenberg C, Doublet B, et al. Molecular basis of bacterial resistance to chloramphenicol and florfenicol[J]. FEMS Microbiol Rev, 2004, 28(5): 519-542. DOI:10.1016/j.femsre.2004.04.001 |
| [23] |
Michaux C, Verneuil N, Hartke A, et al. Physiological roles of small RNA molecules[J]. Microbiology (Reading), 2014, 160(Pt 6): 1007-1019. |
| [24] |
Richter C, Chang JT, Fineran PC. Function and regulation of clustered regularly interspaced short palindromic repeats (CRISPR)/CRISPR associated (Cas) systems[J]. Viruses, 2012, 4(10): 2291-2311. DOI:10.3390/v4102291 |
| [25] |
Shimoni Y, Friedlander G, Hetzroni G, et al. Regulation of gene expression by small non-coding RNAs: a quantitative view[J]. Mol Syst Biol, 2007, 3: 138. DOI:10.1038/msb4100181 |
| [26] |
付竹青. 鲍曼不动杆菌sRNA AbsR25和AbsR28功能的研究[D]. 湛江: 广东医科大学, 2016.
|
| [27] |
álvarez-Fraga L, Rumbo-Feal S, Pérez A, et al. Global assessment of small RNAs reveals a non-coding transcript involved in biofilm formation and attachment in Acinetobacter baumannii ATCC 17978[J]. PLoS One, 2017, 12(8): e0182084. DOI:10.1371/journal.pone.0182084 |
| [28] |
Kuo HY, Chao HH, Liao PC, et al. Functional characterization of Acinetobacter baumannii lacking the RNA chaperone Hfq[J]. Front Microbiol, 2017, 8: 2068. DOI:10.3389/fmicb.2017.02068 |
| [29] |
Sharma A, Sharma R, Bhattacharyya T, et al. Fosfomycin resistance in Acinetobacter baumannii is mediated by efflux through a major facilitator superfamily (MFS) transporter-AbaF[J]. J Antimicrob Chemother, 2017, 72(1): 68-74. DOI:10.1093/jac/dkw382 |
| [30] |
Marchand I, Damier-Piolle L, Courvalin P, et al. Expression of the RND-type efflux pump AdeABC in Acinetobacter baumannii is regulated by the AdeRS two-component system[J]. Antimicrob Agents Chemother, 2004, 48(9): 3298-3304. DOI:10.1128/AAC.48.9.3298-3304.2004 |
| [31] |
Wen YR, Ouyang ZL, Yu Y, et al. Mechanistic insight into how multidrug resistant Acinetobacter baumannii response regulator AdeR recognizes an intercistronic region[J]. Nucleic Acids Res, 2017, 45(16): 9773-9787. DOI:10.1093/nar/gkx624 |
| [32] |
Aranda J, Bardina C, Beceiro A, et al. Acinetobacter baumannii RecA protein in repair of DNA damage, antimicrobial resistance, general stress response, and virulence[J]. J Bacte-riol, 2011, 193(15): 3740-3747. DOI:10.1128/JB.00389-11 |
| [33] |
Ching C, Gozzi K, Heinemann B, et al. RNA-mediated cis regulation in Acinetobacter baumannii modulates stress-induced phenotypic variation[J]. J Bacteriol, 2017, 199(11): e00799-16. |
| [34] |
Touchon M, Cury J, Yoon EJ, et al. The genomic diversification of the whole Acinetobacter genus: origins, mechanisms, and consequences[J]. Genome Biol Evol, 2014, 6(10): 2866-2882. DOI:10.1093/gbe/evu225 |
| [35] |
付恒霞, 毕春霞, 张蒙蒙, 等. 不动杆菌CRISPR系统基因结构的分析及其与耐药性关系的研究[J]. 中国病原生物学杂志, 2018, 13(10): 1073-1077, 1083. |
| [36] |
Yamada J, Yamasaki S, Hirakawa H, et al. Impact of the RNA chaperone Hfq on multidrug resistance in Escherichia coli[J]. J Antimicrob Chemother, 2010, 65(5): 853-858. DOI:10.1093/jac/dkq067 |
| [37] |
Simonsen KT, Nielsen G, Bjerrum JV, et al. A role for the RNA chaperone Hfq in controlling adherent-invasive Escherichia coli colonization and virulence[J]. PLoS One, 2011, 6(1): e16387. DOI:10.1371/journal.pone.0016387 |
| [38] |
Sharma A, Dubey V, Sharma R, et al. The unusual glycine-rich C terminus of the Acinetobacter baumannii RNA chape-rone Hfq plays an important role in bacterial physiology[J]. J Biol Chem, 2018, 293(35): 13377-13388. DOI:10.1074/jbc.RA118.002921 |
| [39] |
El-Mowafi SA, Alumasa JN, Ades SE, et al. Cell-based assay to identify inhibitors of the Hfq-sRNA regulatory pathway[J]. Antimicrob Agents Chemother, 2014, 58(9): 5500-5509. DOI:10.1128/AAC.03311-14 |
| [40] |
Mulhbacher J, Brouillette E, Allard M, et al. Novel riboswitch ligand analogs as selective inhibitors of guanine-related metabolic pathways[J]. PLoS Pathog, 2010, 6(4): e1000865. DOI:10.1371/journal.ppat.1000865 |
| [41] |
Ster C, Allard M, Boulanger S, et al. Experimental treatment of Staphylococcus aureus bovine intramammary infection using a guanine riboswitch ligand analog[J]. J Dairy Sci, 2013, 96(2): 1000-1008. DOI:10.3168/jds.2012-5890 |
| [42] |
Langer S, Hashimoto M, Hobl B, et al. Flavoproteins are potential targets for the antibiotic roseoflavin in Escherichia coli[J]. J Bacteriol, 2013, 195(18): 4037-4045. DOI:10.1128/JB.00646-13 |
| [43] |
Howe JA, Xiao L, Fischmann TO, et al. Atomic resolution mechanistic studies of ribocil: a highly selective unnatural ligand mimic of the E. coli FMN riboswitch[J]. RNA Biol, 2016, 13(10): 946-954. DOI:10.1080/15476286.2016.1216304 |
| [44] |
盛树悦, 陈悦, 张兴梅, 等. 核糖开关及其在抗菌药物方面的研究进展[J]. 生物技术通报, 2017, 33(1): 114-119. |
| [45] |
刘男男, 凌保东. 核糖开关在逆转细菌耐药性机制中的潜在应用[J]. 中华临床医师杂志(电子版), 2013, 7(6): 2673-2676. |
| [46] |
Citorik RJ, Mimee M, Lu TK. Sequence-specific antimicro-bials using efficiently delivered RNA-guided nucleases[J]. Nat Biotechnol, 2014, 32(11): 1141-1145. DOI:10.1038/nbt.3011 |
| [47] |
Kim JS, Cho DH, Park M, et al. CRISPR/Cas9-mediated re-sensitization of antibiotic-resistant Escherichia coli harboring extended-spectrum β-lactamases[J]. J Microbiol Biotechnol, 2016, 26(2): 394-401. DOI:10.4014/jmb.1508.08080 |
| [48] |
Sun LC, He T, Zhang LL, et al. Generation of newly disco-vered resistance gene mcr-1 knockout in Escherichia coli using the CRISPR/Cas9 system[J]. J Microbiol Biotechnol, 2017, 27(7): 1276-1280. DOI:10.4014/jmb.1611.11021 |
| [49] |
宁夏医科大学总医院. 一种利用CRISPR-Cas9清除鲍曼不动杆菌耐药性基因的方法: 中国, CN201610978586.8[P]. 2017-03-29.
|
| [50] |
Oliva G, Sahr T, Buchrieser C. Small RNAs, 5' UTR elements and RNA-binding proteins in intracellular bacteria: impact on metabolism and virulence[J]. FEMS Microbiol Rev, 2015, 39(3): 331-349. DOI:10.1093/femsre/fuv022 |
| [51] |
Weinberg Z, Wang JX, Bogue J, et al. Comparative genomics reveals 104 candidate structured RNAs from bacteria, archaea, and their metagenomes[J]. Genome Biol, 2010, 11(3): R31. DOI:10.1186/gb-2010-11-3-r31 |
| [52] |
Meyer MM. The role of mRNA structure in bacterial translational regulation[J]. Wiley Interdiscip Rev RNA, 2017, 8(1): e1370. |
| [53] |
李娜, 张智, 王岱. 病原菌非编码sRNA的功能及研究新方法[J]. 中国生物化学与分子生物学报, 2017, 33(3): 214-219. |



