肝脓肿和肾脓肿是常见的腹腔内感染性疾病,可进一步导致脓毒性休克并危及生命,且这类疾病的发病率有逐年增加趋势[1-3]。临床上主要依赖传统的细菌培养确定脓肿的致病菌,对于罕见的、生长缓慢的和不可培养的细菌均无法通过传统的细菌培养检测出来[4]。近年来,16S rDNA测序技术已经用于胸腔感染、胆道感染及脑脓肿的致病菌检测,且诊断效能优于传统培养[3],但在肝、肾脓肿中却鲜有报道。肝脓肿的致病菌多经门静脉或胆道途径入肝[5],而肾脓肿则多为血源性或泌尿道逆行感染[6],两者的常见致病菌存在差异。16S rDNA技术能分析微生物群体的多样性、丰富度及群体结构等,为深入分析肝、肾脓肿的病原菌构成提供了新的方法[7]。本研究对肝脓肿和肾脓肿引流液进行16S rDNA测序,旨在比较16S rDNA测序及传统培养方法在肝、肾脓肿病原菌检测中的价值,分析肝脓肿和肾脓肿中病原微生物的构成差异,了解肝、肾脓肿病原菌的特点。
1 对象与方法 1.1 研究对象2019年12月—2020年12月共32例腹腔脓肿患者纳入本研究,其中肝脓肿24例,肾脓肿8例。患者年龄及性别分布见表 2。32例患者其中男性18例(肝脓肿17例,肾脓肿1例),女性14例(肝脓肿7例,肾脓肿7例); 肝脓肿组年龄为29~80岁,平均年龄(60.54±14.28)岁;肾脓肿组年龄为37~76岁,平均年龄(51.75±14.82)岁。所有患者均进行经皮穿刺引流联合抗菌药物治疗,常规应用的抗生素为头孢哌酮钠/舒巴坦钠或美罗培南;对于头孢类抗生素过敏的患者,应用盐酸莫西沙星。
1.2 标本收集经皮穿刺引流脓肿时,使用超声引导下穿刺脓肿,抽出脓液,一式两份,分别进行传统培养和16S rDNA测序。患者的标本编号为肝脓肿(liver abscess,LA)和肾脓肿(renal abscess,RA)。
1.3 传统培养脓肿引流液常规接种培养后进行菌种鉴定,相关操作均由医院检验科微生物室完成。其中菌种鉴定采用VITEK全自动分析仪(法国生物梅里埃公司),结果参照美国临床和实验室标准协会(CLSI)标准进行判定。
1.4 16S rDNA测序使用E.Z.N.A. Stool DNA试剂盒(D4015, Omega, Inc.,USA)从标本中提取DNA,-80℃保存,由浙江杭州联川科技有限公司进行PCR检测。使用引物341F (5’-CCTACGGGNGGCWGCAG-3’) 和805R (5’-GACTACHVGGGTATCTAATCC-3’)[8]对16S rDNA (V3+V4)可变区进行PCR扩增。纯化后的PCR产物采用Quant-iTPicoGreen dsDNA Assay Kit在Qbit荧光定量系统上对文库进行定量,合格的文库浓度应在2 nM以上。使用NovaSeq测序仪进行2×250 bp的双端测序。以SILVA数据库注释结果为准。Alpha多样性和Beta多样性通过抽平(将所有标本的序列数抽取至最少序列标本的序列数)的方式进行归一化, 物种注释使用相对丰度进行归一化处理。Alpha多样性以及Beta多样性均用QIIME2平台进行流程分析,图片由R (v3.5.2)包绘制。物种注释采用QIIME2的插件feature-classifier进行序列比对,比对数据库为SILVA和NT-16S数据库。
1.5 统计学分析应用SPSS 25.0软件包进行分析,符合正态分布的计量资料以均数±标准差(x±s)表示,组间比较采用t检验或秩转换的非参数检验;非正态分布的计量资料以中位数和四分位数间距[M(P25,P75)]表示,组间比较采用秩转换的非参数检验;计数资料以例数(%)表示,组间比较采用Fisher’s确切概率法检验,P≤0.05为差异具有统计学意义。
2 结果 2.1 16S rDNA测序与细菌培养结果比较32例标本,26例(81.25%)患者脓肿引流液细菌培养阳性,而16S rDNA测序阳性率为100%,定义16S的单独致病菌为reads>96%[4]。见表 1。
| 表 1 标本细菌培养和16S rDNA测序结果比较 Table 1 Bacterial culture and 16S rDNA sequencing results of specimens |
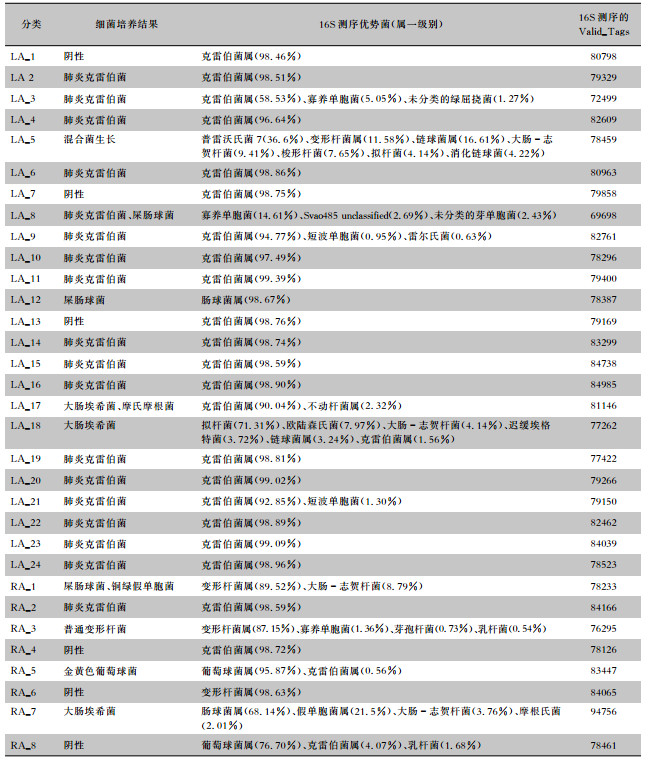
|
| 表 2 肝脓肿组和肾脓肿组患者临床特征比较 Table 2 Comparison of clinical characteristics of patients in liver abscess group and renal abscess group |

|
肝肾脓肿患者均有发热、腹痛、乏力等不适,两组患者在性别、腰痛症状方面差异均有统计学意义(均P<0.05),在基础疾病及脓肿影像表现方面差异均无统计学意义(均P>0.05)。
2.3 肝、肾脓肿检出病原菌差异Chao1指数反映标本的物种丰富程度,分析显示,肝脓肿组Chao1指数与肾脓肿组,差异无统计学意义,提示肝脓肿组的物种丰度与肾脓肿组相近。见图 1。堆叠柱状图中可以看出肝脓肿和肾脓肿在属一级别的菌种组成存在差异,其中横坐标为组别,纵坐标为相对丰度,不同的颜色代表了不同的菌属。见图 2。主成分分析(principal component analysis, PCA):肝脓肿组和肾脓肿组组内标本离得很近,但两组间距离相对较远,进一步表明两组间微生物组成差异明显。见图 3。
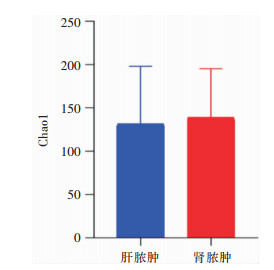 |
| 图 1 肝、肾脓肿组Chao1指数比较 Figure 1 Comparison of Chao1 index between liver and renal abscess groups |
 |
| 图 2 肝、肾脓肿组检出病原菌在属一级别的堆叠柱状图 Figure 2 Stacked bar chart at the genus level of pathogens in liver and renal abscess groups |
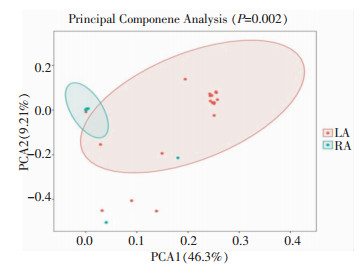 |
| 图 3 肝、肾脓肿组检出病原菌PCA Figure 3 Principal component analysis on pathogens in liver and renal abscess groups |
采用线性判别式分析(linear discriminant analysis, LDA)进一步分析肝、肾脓肿病原存在差异的物种,LDA图提示,肝脓肿组菌群以变形菌纲、肠杆菌目、肠杆菌科、克雷伯菌属为主要特征差异细菌菌类;肾脓肿组菌群以丛毛单胞菌科、分枝杆菌科、变形杆菌属、短芽孢杆菌属、解纤维素根瘤菌属、毛螺菌属、纤毛菌属、嗜糖假单胞菌属、分枝杆菌属、Thermicanus菌属以及Saccharimonadaceae菌属为主要特征差异细菌菌类。见图 4。
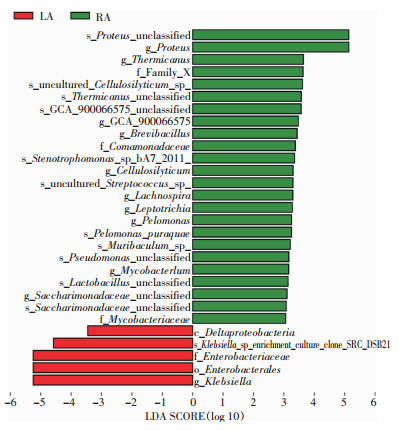 |
| 图 4 肝、肾脓肿组检出病原菌LDA Figure 4 Linear discriminant analysis on pathogens in liver and renal abscess groups |
目前,临床上仍主要采用传统培养方法鉴定细菌。然而培养的细菌鉴定成功率却比较低,因为标本中存在生长缓慢或不可培养的细菌。除此之外,传统培养比较耗时,不利于临床工作的进一步开展。随着DNA测序和PCR技术的日益普及,16S rDNA测序逐渐成为一种强有力的菌群鉴定方法,其比传统的细菌培养准确性更高,能鉴定出的菌种更多。本研究将16S rDNA测序用于肝、肾脓肿的病原菌检测,结果表明该技术对病原菌的检测能力优于传统培养。另外,对于传统培养获得病原菌结果的标本,16S rDNA还能够鉴定出优势菌以外的其他菌属,有利于更加深入了解脓肿内的微生态。
本研究显示,肺炎克雷伯菌是导致肝脓肿的主要病原菌,与以往研究[1, 10-11]相符。16S rDNA测得的结果显示,引起肝脓肿的其他致病菌还包括肠球菌属、普雷沃氏菌属、寡养单胞菌属、拟杆菌属等,这些细菌大多是肠道定植菌。肝脏受肝动脉及门静脉的双重供血,并经胆道与胃肠道相通,受细菌感染的机会较大[12-14]。本组肝脓肿病例可能大多是因肠道微生物突破肠黏膜屏障,经门静脉途径导致的。本研究中8例肾脓肿患者中除RA2、RA4的优势菌属为克雷伯菌属外,其余几个标本的优势菌属为葡萄球菌属、变形杆菌属,以及肠球菌属。其中克雷伯菌属及变形杆菌属是革兰阴性杆菌,是泌尿系统感染的常见致病菌,可逆行导致肾脓肿[10, 15],葡萄球菌属和肠球菌属也是引起尿路感染的常见细菌[2, 16]。由此推测本组肾脓肿主要为泌尿系统逆行感染所致。
利用16S rDNA测序数据进一步分析肝、肾脓肿内的菌群特征。Alpha多样性中的Chao1指数提示肝、肾脓肿的菌群丰度相近;Beta多样性分析中,PCA能明显区别两类脓肿的菌群,提示菌群构成存在显著差异;LDA进一步探索差异的菌群,结果发现肾脓肿组主要的病原菌为变形杆菌属等,而肝脓肿组主要的病原菌为克雷伯菌属以及肠杆菌属等,可能是由两种脓肿发病途径不同所致。不同的菌群结构将引起不同的代谢特征,以及不同的临床症状。本研究为后续基于脓肿的菌群特征,研究脓肿的临床特点提供了新的思路。
当然,16S rDNA测序也存在一些不足。首先,16S rDNA测序无法提供细菌的药敏试验结果。其次,基因测序是一种相对昂贵的鉴定方法,因此不建议将其作为临床常规检查,但是对于传统培养阴性,并且需要进一步明确致病菌以指导临床用药的情况,基因测序是一种好的选择。另外,16S rDNA技术仅能在属水平上区分细菌,未具体到种水平。下一步考虑进行宏基因组测序,以将病原菌构成精确到种一级,并进行相关功能学研究[17-18]。
综上所述,16S rDNA测序可检测传统培养阴性的病原菌,肝、肾脓肿病原菌组成存在差异,16S rDNA测序是深入了解肝肾脓肿病原菌特点的有效方法。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
吴元清, 林杰, 邱堃, 等. 96例细菌性肝脓肿患者病原学及临床特征[J]. 中华医院感染学杂志, 2021, 31(3): 413-418. Wu YQ, Lin J, Qiu K, et al. Etiological and clinical characteristics of 96 patients with bacterial liver abscess[J]. Chinese Journal of Nosocomiology, 2021, 31(3): 413-418. |
| [2] |
Liu XQ, Wang CC, Liu YB, et al. Renal and perinephric abscesses in West China Hospital: 10-year retrospective-descriptive study[J]. World J Nephrol, 2016, 5(1): 108-114. DOI:10.5527/wjn.v5.i1.108 |
| [3] |
Bivand JM, Nygaard RM, Kommedal ∅. Characterization of abscesses from liver, pancreas and kidney using deep sequencing of the 16S rRNA gene[J]. Diagn Microbiol Infect Dis, 2021, 99(3): 115277. DOI:10.1016/j.diagmicrobio.2020.115277 |
| [4] |
Song YG, Shim SG, Kim KM, et al. Profiling of the bacteria responsible for pyogenic liver abscess by 16S rRNA gene pyrosequencing[J]. J Microbiol, 2014, 52(6): 504-509. DOI:10.1007/s12275-014-4241-7 |
| [5] |
郑伟, 陈竹凤, 杨文韬, 等. 细菌性肝脓肿合并血流感染的临床特点及其相关危险因素分析[J]. 中外医学研究, 2021, 19(13): 41-44. Zheng W, Chen ZF, Yang WT, et al. Clinical characteristics and related risk factors of bacterial liver abscess complicated with bloodstream infection[J]. Chinese and Foreign Medical Research, 2021, 19(13): 41-44. |
| [6] |
任大勇, 姜林, 刘敏. 肾脓肿误诊为肾肿瘤临床分析[J]. 临床误诊误治, 2020, 33(7): 11-14. Ren DY, Jiang L, Liu M. Clinical analysis of renal abscess misdiagnosed as renal tumor[J]. Clinical Misdiagnosis & Mistherapy, 2020, 33(7): 11-14. DOI:10.3969/j.issn.1002-3429.2020.07.004 |
| [7] |
Woo PCY, Lau SKP, Teng JLL, et al. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories[J]. Clin Microbiol Infect, 2008, 14(10): 908-934. |
| [8] |
Logue JB, Stedmon CA, Kellerman AM, et al. Experimental insights into the importance of aquatic bacterial community composition to the degradation of dissolved organic matter[J]. ISME J, 2016, 10(3): 533-545. DOI:10.1038/ismej.2015.131 |
| [9] |
Chang ZH, Zheng JH, Ma YJ, et al. Analysis of clinical and CT characteristics of patients with Klebsiella pneumoniae liver abscesses: an insight into risk factors of metastatic infection[J]. Int J Infect Dis, 2015, 33: 50-54. |
| [10] |
何蕾, 阴晴, 吴亮, 等. 肺炎克雷伯菌所致肝脓肿患者的临床特征及毒力基因检测[J]. 中国感染控制杂志, 2021, 20(7): 619-625. He L, Yin Q, Wu L, et al. Clinical characteristics and virulence genes of patients with liver abscess caused by Klebsiella pneumoniae[J]. Chinese Journal of Infection Control, 2021, 20(7): 619-625. |
| [11] |
姜英杰, 畅智慧, 周大昕, 等. 化脓性肝脓肿介入引流时间及影响因素分析[J]. 中国临床医学影像杂志, 2014, 25(6): 438-440. Jiang YJ, Chang ZH, Zhou DX, et al. Factors associated with drainage time of percutaneous catheter drainage for pyogenic liver abscess[J]. Journal of China Clinic Medical Imaging, 2014, 25(6): 438-440. |
| [12] |
封立魁, 周林, 杜国盛. 细菌性肝脓肿的病原学特点及治疗对策[J]. 武警医学, 2021, 32(6): 486-489. Feng LK, Zhou L, Du GS. Pathogenic characteristics and treatments of bacterial liver abscess[J]. Medical Journal of the Chinese People's Armed Police Forces, 2021, 32(6): 486-489. DOI:10.3969/j.issn.1004-3594.2021.06.007 |
| [13] |
莫贝贝, 庄丽维. 肠道微生态与肝脏疾病关系的研究进展[J]. 医学综述, 2021, 27(14): 2761-2766. Mo BB, Zhuang LW. Research progress in relationship between intestinal microecology and liver diseases[J]. Medical Recapitulate, 2021, 27(14): 2761-2766. DOI:10.3969/j.issn.1006-2084.2021.14.011 |
| [14] |
黄雪莹, 刘叶莹, 陈思思, 等. 肝硬化合并糖尿病患者肠道菌群研究进展[J]. 中国医药, 2021, 16(7): 1100-1103. Huang XY, Liu YY, Chen SS, et al. Research progress of intestinal flora in patients with cirrhosis complicated with diabetes mellitus[J]. China Medicine, 2021, 16(7): 1100-1103. |
| [15] |
Armbruster CE, Mobley HLT, Pearson MM. Pathogenesis of Proteus mirabilis infection[J]. EcoSal Plus, 2018, 8(1). DOI:10.1128/ecosalplus.ESP-0009-2017 |
| [16] |
岑叶平, 崔裕山, 常燕子, 等. 肠球菌属细菌的临床分布与耐药性分析[J]. 中国微生态学杂志, 2019, 31(10): 1215-1218. Cen YP, Cui YS, Chang YZ, et al. Clinical distribution and drug resistance of Enterococci[J]. Chinese Journal of Microecology, 2019, 31(10): 1215-1218. |
| [17] |
Giuffrè M, Campigotto M, Campisciano G, et al. A story of liver and gut microbes: how does the intestinal flora affect li-ver disease? A review of the literature[J]. Am J Physiol Gastrointest Liver Physiol, 2020, 318(5): G889-G906. |
| [18] |
敬晓棋, 李娟, 郝婷婷, 等. 宏基因组学技术在动物传染病原挖掘中的应用与进展[J]. 基因组学与应用生物学: 1-7. http://kns.cnki.net/kcms/detail/45.1369.Q.20210527.1622.006.html. Jing XQ, Li J, Hao TT, et al. Application and progress of metagenomics technology in animal infectious pathogen excavation[J]. Genomics and Applied Biology: 1-7. http://kns.cnki.net/kcms/detail/45.1369.Q.20210527.1622.006.html. |



