2. 宁夏医科大学公共卫生与管理学院, 宁夏 银川 750004;
3. 宁夏环境因素与慢性病控制重点实验室, 宁夏 银川 750004
2. School of Public Health and Management, Ningxia Medical University, Yinchuan 750004, China;
3. Ningxia Key Laboratory of Environmental Factors and Chronic Disease Control, Yinchuan 750004, China
人呼吸道合胞病毒(human respiratory syncytial virus, HRSV) 是婴幼儿急性下呼吸道感染(acute lower respiratory tract infection,ALRTI)和住院相关感染的主要病原体,5岁以下ALRTI死亡的儿童中约13%与之相关[1]。在老年人和免疫功能低下的成年人或既往有基础疾病的患者中,HRSV感染的发病率和病死率更高[2]。HRSV每年导致全球约3 310万例下呼吸道感染,320万例住院,149.4万例5岁以下儿童死亡[3],HRSV相关呼吸道感染是一个全球性的重大公共问题。HRSV由10个基因构成,至少可以编码11种蛋白, 包括9个结构蛋白(G、F、SH蛋白为RSV跨膜糖蛋白,N、P、L 3种蛋白相互作用构成核衣壳蛋白,M、M2-1、M2-2为基质蛋白)和2个非结构蛋白(NS1和NS2)。HRSV只有1个血清学,根据其对单克隆抗体的反应性、抗原性和遗传特异性,将呼吸道合胞病毒(RSV)分为RSVA和RSVB两个亚型,再根据G蛋白的第二高变区(the second hypervariable region in the G protein,HVR2)将HRSV-A划分为18个基因型,分别为ON1~2、GA1~7、SAA1~2、NA1~4、TN1~2、CBA[4-7],将HRSV-B划分为39个基因型分别为GB1~13、BA1~14、BAc、URU1~2、SAB1~4、CB1(GB5)、CBB、BA-CCA、BA-CCB、THB[5, 8-10]。本研究分析银川市4株HRSV全基因组序列,研究其基因特征及氨基酸变异,以期为HRSV疾病防控及疫苗研制提供理论参考。
1 对象与方法 1.1 研究对象收集2021年银川市2所流感监测哨点医院的流感样标本共1 100份,选择发热伴有严重呼吸道感染症状的标本共669份,采用实时荧光定量逆转录聚合酶链反应(real-time reverse transcription polymerase chain reaction, real time RT-PCR),筛选出HRSV阳性标本为后续二代测序做准备, 该研究的患者及其家属均对此知情并签署知情同意书。
1.2 主要仪器及设备硕世全自动核酸提取仪及核酸快速提取试剂盒均购自硕世生物科技股份有限公司,呼吸道6种病毒核酸检测试剂盒(包括HRSV)购自上海伯杰医疗有限公司,PCR扩增仪(7500 Fast Real-Time PCR System、QuantStudioTM 7)均购自美国ABI公司。
1.3 实验方法按照病毒核酸快速提取试剂盒说明书,取200 μL进行病毒总RNA的提取。采用RT-PCR方法检测6种呼吸道病毒核酸,依据试剂盒说明书进行操作。
1.4 基因测序通过呼吸道病毒检测,获得HRSV阳性标本,筛选出HRSV CT值<25的标本(CT值越小,病毒载量越高,二代全基因组测序成功率高),取1 000 μL临床标本送至上海伯杰医疗科技有限公司进行合胞病毒二代全基因组测序。
1.5 生物信息学分析使用MEGA7软件进行序列比对、亲缘关系进化树构建和遗传距离的计算分析,使用邻接法(neighbor-joining method, NJ)构建系统发育进化树,使用p-distance法估计遗传距离,Bootstrap值均选择1 000次引导重复测试准确性。使用DNAStar软件中MegAlign分析测序株与全球各代表株核苷酸和氨基酸序列的同源性。
1.6 全球代表株在NCBI中搜索并下载HRSV的A亚型和B亚型代表株。由于B亚型全基因组序列代表株检索出的代表株型别较少,故本研究选择将G基因片段序列整理下载。其中全球38条HRSVA亚型代表株来自于12个国家,历时36年(1984—2020年),包括标准株A2株(M74568.1),共有9种基因型别;全球35条HRSVB亚型代表株来自于16个国家,历时27年(1993年—2020年),包括标准株B1株(AF013254.1),共有14种基因型别。
2 结果 2.1 HRSV阳性情况选择的669份鼻咽拭子标本,采用RT-PCR检测6种呼吸道病毒核酸,获得HRSV阳性标本共60份,阳性率为8.97%。
2.2 系统进化分析60份HRSV阳性标本中,CT值<25的HRSV的标本共8份,进行A、B亚型分型后,得到5株A亚型,3株B亚型。最终符合测序要求且测序成功共4株(2株HRSVA,2株HRSVB),分别命名为YinChuanHRSVA-01、YinChuanHRSVA-02、YinChuanHRSVB-01、YinChuanHRSVB-02,长度分别为15 180、15 176、15 209、15 217 bp。
将2株HRSVA亚型的测序结果与全球38条A亚型代表株进行全基因组序列比对与分析,分别基于全基因组序列(图 1)、G蛋白基因组序列(图 2)、F蛋白基因组序列(图 3)构建亲缘关系进化树,在高变蛋白G基因的系统发育树上发现,银川2株HRSVA均与ON1基因型代表株在同一分支上,因此判定为ON1型,且与在全序列、保守蛋白F基因的亲缘关系进化树上结论一致。通过对G蛋白基因全长进行遗传距离分析发现,两条银川HRSVA间的核苷酸(氨基酸)遗传距离为0.001(0.003),这两条HRSVA与所有ON1型代表株的核苷酸(氨基酸)遗传距离为0.004~0.023(0.006~0.038),YinChuanHRSVA-01、YinChuanHRSVA-02分别与标准株A2的核苷酸(氨基酸)遗传距离为0.100 (0.157)、0.098(0.153),YinChuanHRSVA-01、YinChuanHRSVA-02与GA1代表株(KJ723474.1)的核苷酸(氨基酸)遗传距离最远,分别为0.125(0.181)、0.124(0.177)。
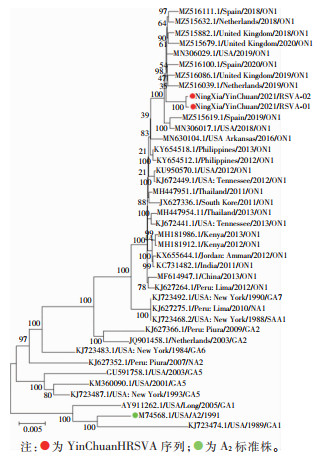 |
| 图 1 2条YinChuanHRSVA和38条全球代表株基于基因组全长序列构建系统进化树 Figure 1 Phylogenetic trees for 2 YinChuanHRSVA and 38 global representative strains constructed based on genomic full-length sequence |
 |
| 图 2 2条YinChuanHRSVA和38条全球代表株基于G蛋白基因组序列构建系统进化树 Figure 2 Phylogenetic trees for 2 YinChuanHRSVA and 38 global representative strains constructed based on genomic sequence of G protein |
 |
| 图 3 2条YinChuanHRSVA和38条全球代表株基于F蛋白基因组序列构建系统进化树 Figure 3 Phylogenetic trees for 2 YinChuanHRSVA and 38 global representative strains constructed based on genomic sequence of F protein |
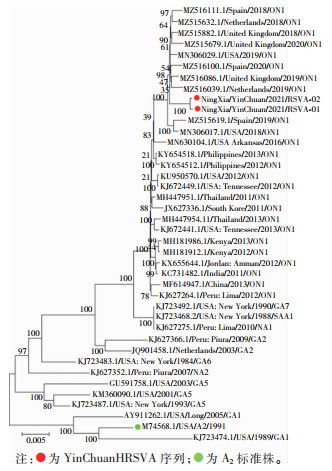
将2株HRSVB亚型的测序结果与全球35条B亚型代表株进行G蛋白基因组序列比对与分析,基于G蛋白基因组序列构建亲缘关系进化树见图 4,在高变蛋白G基因的系统发育树上发现,银川2株HRSVB均与BA9基因型代表株在同一分支上,判定为BA9型。分析G蛋白基因全长遗传距离发现,两条银川HRSVB间的核苷酸(氨基酸)遗传距离为0.002(0.003),这两条HRSVB与所有BA9型代表株的核苷酸(氨基酸)遗传距离为0.004~0.034(0.003~0.052),YinChuanHRSVB-01、YinChuanHRSVB-02分别与标准株B1的核苷酸(氨基酸)遗传距离为0.082(0.119)、0.082(0.122),YinChuanRSVB-01、YinChuanHRSVB-02与GB1代表株(M73545)的核苷酸(氨基酸)遗传距离最远,分别为0.085(0.158)、0.085(0.162)。
 |
| 图 4 2条YinChuanHRSVB和35条全球代表株基于G蛋白基因组序列构建系统进化树 Figure 4 Phylogenetic trees for 2 YinChuanHRSVB and 35 global representative strains constructed based on genomic sequence of G protein |
将银川2条HRSVA序列与38条HRSVA亚型的全球代表株进行全基因组序列、G蛋白基因组序列、F蛋白基因组序列核苷酸及氨基酸同源性分析。银川2条HRSVA之间全序列、G蛋白基因、F蛋白基因的核苷酸(氨基酸)同源性分别为99.9%(92.3%)、99.9%(99.4%)、100%(99.8%)。将银川2条HRSVA与各基因型别的平均核苷酸(氨基酸)同源性整理,结果发现,银川2条HRSVA在全序列和主要蛋白基因(F、G)上,都与ON1基因型的平均核苷酸(氨基酸)同源性最高,与GA1基因型的平均核苷酸(氨基酸)同源性最低。银川2条HRSVA全长序列与MN306029.1/USA/2019/ON1全长序列的核苷酸(氨基酸)最高,均为99.3%(91.2%),与KJ723474/USA/1989/GA1最低。见表 1。
| 表 1 YinChuanHRSVA与不同基因型代表株主要蛋白基因的核苷酸及氨基酸同源性分析 Table 1 Nucleotide and amino acid homology of main protein genes between YinChuanHRSVA and representative strains of different genotypes |

|
将银川2条HRSVB与35条HRSVB亚型的全球代表株进行G蛋白基因组序列核苷酸及氨基酸同源性分析(见图 5、6),银川2条HRSVB之间G蛋白基因序列的核苷酸(氨基酸)同源性为99.2%(98.4%),YinChuanHRSVB-01、YinChuanHRSVB-02序列均与MW160815.1/Australia/2017/BA9的G蛋白基因序列核苷酸(氨基酸)同源性最高,分别为97.8%(96.6%)、97.8%(96.3%), 均与JX908836.1/Brazil/2007/GB3的核苷酸(氨基酸)同源性最低,分别为67.4%(65.4%)、67.4%(65.1%)。
 |
| 图 5 YinChuanHRSVB与不同基因型代表株核苷酸同源性分析 Figure 5 Nucleotide homology between YinChuanHRSVB and representative strains of different genotypes |
 |
| 图 6 YinChuanHRSVB与不同基因型代表株氨基酸同源性分析 Figure 6 Amino acid homology between YinChuanHRSVB and representative strains of different genotypes |
与HRSVA原型株M11486株的G蛋白氨基酸序列比较,加拿大首株ON1型(JN257693.1)、YinChuanHRSVA-01、YinChuanHRSVA-02均插入了24个重复氨基酸(第285~307位),终止密码子均是TGA。与加拿大首株进行比较发现(见图 7),银川2条HRSVA在第113、131、178、257、258、266位均发生了突变,分别为T113I、V131D、N178G、E257K、H258Q、H266L,YinChuanHRSVA-01在第134位发生K突变为R(K134R)。
 |
| 图 7 HRSVA-ON1型G蛋白氨基酸序列比较 Figure 7 Comparison of amino acid sequences of HRSVA-ON1 G protein |
与HRSVB原型株CH-18537株的G蛋白氨基酸序列比较,BA型代表株(AY333364.1)、YinChuanHRSVA-01、YinChuanHRSVA-02均插入了20个重复氨基酸(第260—279位),与BA型代表株进行比较发现(见图 8),银川2条HRSVB的终止密码子为TAA,在第159位和第160位均出现氨基酸缺失,在G蛋白第二高变区的第254、270、276、290位均发生T突变为I(T254I、T270I、T276I、T290I)。
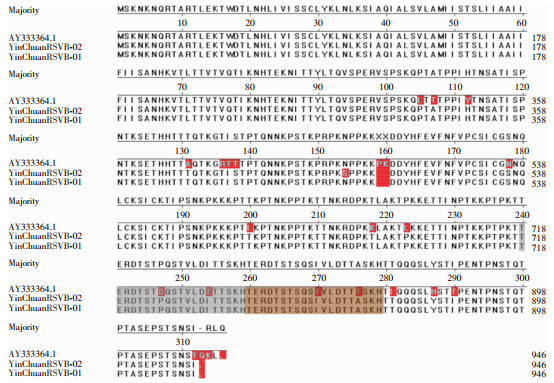 |
| 图 8 HRSVB-BA9型G蛋白氨基酸序列比较 Figure 8 Comparison of amino acid sequences of HRSVB-BA9 G protein |
HRSV是导致婴幼儿和免疫缺陷人群出现严重呼吸道感染的主要原因,所有年龄段的人群感染后都会出现不同程度的临床症状,如轻微的上呼吸道感染、严重的肺炎或细支气管炎[11-12]。HRSV的流行情况因地而异。本研究中人群HRSV阳性率为8.97%,与研究对象为全人群相关研究相比,HRSV阳性率低于北京市(15.30%)[13],高于西安市(4.51%)[14]、白银市(4.08%)[15]。HRSV导致的呼吸道感染症状与新型冠状病毒肺炎的症状相似,且HRSV的阳性率高,对新型冠状病毒肺炎疫情的日常防控造成了影响。目前,尚无针对HRSV治疗有效的临床方案和安全有效的疫苗,给社会带来了巨大的经济负担。加强对HRSV的分子流行病学及基因特征分析,能为临床诊断和疫苗研制提供科学依据。G蛋白(glycoprotein,黏附蛋白)和F蛋白(fusion protein,融合蛋白)是HRSV的主要表面糖蛋白,F蛋白基因序列在亚型间更保守,激发的中和抗体具有更广谱的保护作用,是HRSV预防及治疗性抗体、药物及疫苗开发的重要靶标,G蛋白基因在病毒与宿主细胞间的黏附和穿膜入胞过程中发挥重要作用,促进病毒与宿主细胞的吸附过程,G蛋白表面的B及T细胞抗原表位,能激发宿主的免疫应答并且产生保护性的中和抗体,由于G蛋白的遗传多样性,其相应的中和抗体大多没有广泛保护作用,也反映了G蛋白具有强大的免疫压力,因此研究G和F蛋白基因序列,对疫苗研制也至关重要。本研究对HRSVA亚型的全基因组、G和F蛋白基因,以及HRSVB的G蛋白基因分别构建了亲缘关系进化树及核苷酸(氨基酸)同源性分析,结果显示,两条银川HRSVA均属于ON1型,且均与美国(MN306029.1/USA/2019/ON1)基因全长序列同源性最高,两条银川HRSVB都均属于BA9型,且与澳大利亚(MW160815.1/Australia/2017/BA9)的G蛋白基因序列同源性最高。在系统进化树上还发现,基因型一致的序列更多的是通过时间(年份)聚集在一分支上,而不是地点聚集在一起,表明HRSV基因型在全球范围内快速传播的。
BA型于1999年首次在阿根廷报道,其特点是在B亚型G基因C-末端中插入了60个重复氨基酸序列,ON1型于2010年首次在加拿大报道,其特点是在A亚型G基因C-末端插入72个重复核苷酸序列。通过氨基酸变异分析发现,2条序列是ON1型,2条序列为BA型。此4条G蛋白氨基酸的突变主要集中在第一高变区和第二高变区,银川市ON1型的5个氨基酸变异位点(T113I、V131D、N178G、H258Q、H266L)与广州报道[16]的ON1型变异位点一致。银川市BA型6个氨基酸变异位点(K218T、L223P、S247P、T270I、I281T、H287Y)与沙特阿拉伯报道[17]的BA型变异位点一致,同时各地区出现不同的氨基酸变异位点,说明该病毒的变异位点存在地区特异性,应将相同变异位点与地区特异性变异位点相结合,研发出广泛适用于全国各地人群的疫苗。Liang等[18]对兰州HRSV的进化推测显示,过去GA2取代GA5成为优势基因型用时7年,HRSVB亚型中BA基因型取代非BA基因型用时5年,而ON1仅仅不到2年就完全取代NA1基因型。因此,在未来十年或更长时间内,BA基因型的传播有可能被ON1取代。
因地域和年份不同HRSV在流行中呈现出不同的变化,可表现为HRSVA、HRSVB亚型交替流行,即以其中一种亚型为主要流行亚型,也可以表现为HRSVA、HRSVB亚型同时为主要流行亚型。目前全球的流行亚型为HRSV-A中的ON1型和HRSV-B中的BA9型。本研究显示,2021年银川市HRSV出现了HRSVA和HRSVB共流行的情况。不足之处:本研究未对所有HRSV阳性标本进行A、B亚型分型,也未对RSVA和RSVB进一步分型。尚不能确定2021年银川市HRSV主要流行的优势基因型。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Jefferies K, Drysdale SB, Robinson H, et al. Presumed risk factors and biomarkers for severe respiratory syncytial virus disease and related sequelae: protocol for an observational multicenter, case-control study from the Respiratory Syncytial Virus Consortium in Europe (RESCEU)[J]. J Infect Dis, 2020, 222(Suppl 7): S658-S665. |
| [2] |
Colosia AD, Yang J, Hillson E, et al. The epidemiology of medically attended respiratory syncytial virus in older adults in the United States: a systematic review[J]. PLoS One, 2017, 12(8): e0182321. DOI:10.1371/journal.pone.0182321 |
| [3] |
Shi T, McAllister DA, O'Brien KL, et al. Global, regional, and national disease burden estimates of acute lower respiratory infections due to respiratory syncytial virus in young children in 2015: a systematic review and modelling study[J]. Lancet, 2017, 390(10098): 946-958. DOI:10.1016/S0140-6736(17)30938-8 |
| [4] |
Venter M, Madhi SA, Tiemessen CT, et al. Genetic diversity and molecular epidemiology of respiratory syncytial virus over four consecutive seasons in South Africa: identification of new subgroup A and B genotypes[J]. J Gen Virol, 2001, 82(Pt 9): 2117-2124. |
| [5] |
Duvvuri VR, Granados A, Rosenfeld P, et al. Genetic diversity and evolutionary insights of respiratory syncytial virus A ON1 genotype: global and local transmission dynamics[J]. Sci Rep, 2015, 5: 14268. DOI:10.1038/srep14268 |
| [6] |
Schobel SA, Stucker KM, Moore ML, et al. Respiratory syncytial virus whole-genome sequencing identifies convergent evolution of sequence duplication in the c-terminus of the G gene[J]. Sci Rep, 2016, 6: 26311. DOI:10.1038/srep26311 |
| [7] |
Song JH, Zhang Y, Wang HL, et al. Emergence of ON1 ge-notype of human respiratory syncytial virus subgroup A in China between 2011 and 2015[J]. Sci Rep, 2017, 7(1): 5501. DOI:10.1038/s41598-017-04824-0 |
| [8] |
Gimferrer L, Andrés C, Campins M, et al. Circulation of a novel human respiratory syncytial virus Group B genotype du-ring the 2014-2015 season in Catalonia (Spain)[J]. Clin Microbiol Infect, 2016, 22(1): 97. e5-97. e8. DOI:10.1016/j.cmi.2015.09.013 |
| [9] |
ábrego LE, Delfraro A, Franco D, 等. Genetic variability of human respiratory syncytial virus group B in Panama reveals a novel genotype BA14[J]. J Med Virol, 2017, 89(10): 1734-1742. |
| [10] |
Gaymard A, Bouscambert-Duchamp M, Pichon M, et al. Genetic characterization of respiratory syncytial virus highlights a new BA genotype and emergence of the ON1 genotype in Lyon, France, between 2010 and 2014[J]. J Clin Virol, 2018, 102: 12-18. DOI:10.1016/j.jcv.2018.02.004 |
| [11] |
Cane PA. Molecular epidemiology of respiratory syncytial virus[J]. Rev Med Virol, 2001, 11(2): 103-116. DOI:10.1002/rmv.305 |
| [12] |
Collins PL, Melero JA. Progress in understanding and controlling respiratory syncytial virus: still crazy after all these years[J]. Virus Res, 2011, 162(1-2): 80-99. DOI:10.1016/j.virusres.2011.09.020 |
| [13] |
李海明, 李俊, 王婷. 北京地区上呼吸道感染发生情况及病毒感染亚型分布调查[J]. 公共卫生与预防医学, 2022, 33(1): 128-131. Li HM, Li J, Wang T. Prevalence of upper respiratory tract infection and distribution of virus subtypes in Beijing Area[J]. Journal of Public Health and Preventive Medicine, 2022, 33(1): 128-131. DOI:10.3969/j.issn.1006-2483.2022.01.028 |
| [14] |
闫芍药, 原伟莉, 黎巧玲. 2016—2020年西安地区急性呼吸道感染患者病毒病原学特征分析[J]. 华南预防医学, 2021, 47(11): 1417-1419, 1423. Yan SY, Yuan WL, Li QL. Analysis of viral pathogenic chara- cteristics of patients with acute respiratory tract infection in Xi'an from 2016 to 2020[J]. South China Journal of Preventive Medicine, 2021, 47(11): 1417-1419, 1423. |
| [15] |
郭规, 曾同霞, 杨军鹏, 等. 2018—2020年白银市发热呼吸道症候群监测分析[J]. 疾病预防控制通报, 2021, 36(5): 38-40, 50. Guo G, Zeng TX, Yang JP, et al. Surveillance and analysis of febrile respiratory syndrome in Baiyin City from 2018 to 2020[J]. Bulletin of Disease Control & Prevention(China), 2021, 36(5): 38-40, 50. |
| [16] |
Li WW, Wang YL, Yu B, et al. Disease severity of respiratory syncytial virus (RSV) infection correlate to a novel set of five amino acid substitutions in the RSV attachment glycoprotein (G) in China[J]. Virus Res, 2020, 281: 197937. DOI:10.1016/j.virusres.2020.197937 |
| [17] |
Ahmed A, Haider SH, Parveen S, et al. Co-circulation of 72bp duplication group a and 60bp duplication group B respiratory syncytial virus (RSV) strains in Riyadh, Saudi Arabia during 2014[J]. PLoS One, 2016, 11(11): e0166145. DOI:10.1371/journal.pone.0166145 |
| [18] |
Liang X, Liu DH, Chen D, et al. Gradual replacement of all previously circulating respiratory syncytial virus A strain with the novel ON1 genotype in Lanzhou from 2010 to 2017[J]. Medicine (Baltimore), 2019, 98(19): e15542. DOI:10.1097/MD.0000000000015542 |



