感染性疾病作为全球十大死亡原因之一,病原菌的早期诊断对其临床治疗的开展至关重要[1-3]。而及时准确的诊断病原菌现今仍具有很大的挑战性。传统微生物培养、靶向核酸检测及抗原抗体检测都存在一定的局限性[4-6]。宏基因组测序(meta-genomic next-generation sequencing,mNGS)是基于高通量测序平台,直接对标本中的DNA或RNA进行无偏性测序,通过生物信息学分析,检测标本中可能含有的病原微生物(包括病毒、细菌、真菌和寄生虫)[7]。与传统基于培养学的病原检测技术相比,mNGS具有检测速度快、种类多、灵敏度高等特点,已逐渐成为临床疑难和未知病原微生物检验的重要手段[8-11]。为规范mNGS技术在感染性疾病诊断中的应用,国内临床指南和专家共识也在不断发布与更新[4, 12-14]。2016年以来,国内开展mNGS的第三方检验平台已达数十家,但各家平台mNGS检测产品的性能差异性较大,存在很多技术问题,且缺乏质量管理控制和评价体系,为医疗机构实验室本地化带来挑战[15-16]。为便于医疗机构更有效地开展mNGS本地化检测系统,丰富国内mNGS数据,本研究选取了国内临床应用较为成熟的三家mNGS检测平台,对其规范开展不同产品性能评估。
1 材料与方法 1.1 mNGS厂家研究选取3家mNGS临床应用成熟的平台,分别命名为A、B、C。其中A和B来自北京,C来自江苏。
1.2 测试样品测试样品包括参考品和临床标本。
1.2.1 参考品参考品主要由中国食品药品检定研究院提供,主要有D系列和M系列,规格为1 mL/管,见表 1、2。
| 表 1 D系列参考品 Table 1 References of D series |
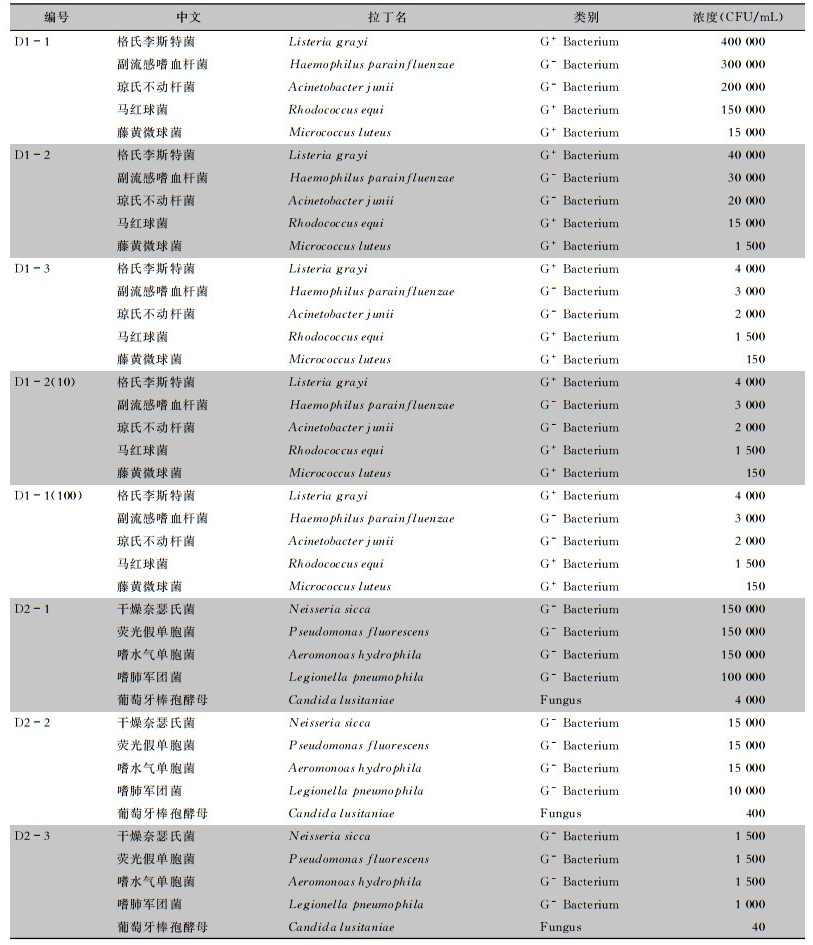
|
| 表 2 M系列参考品 Table 2 References of M series |
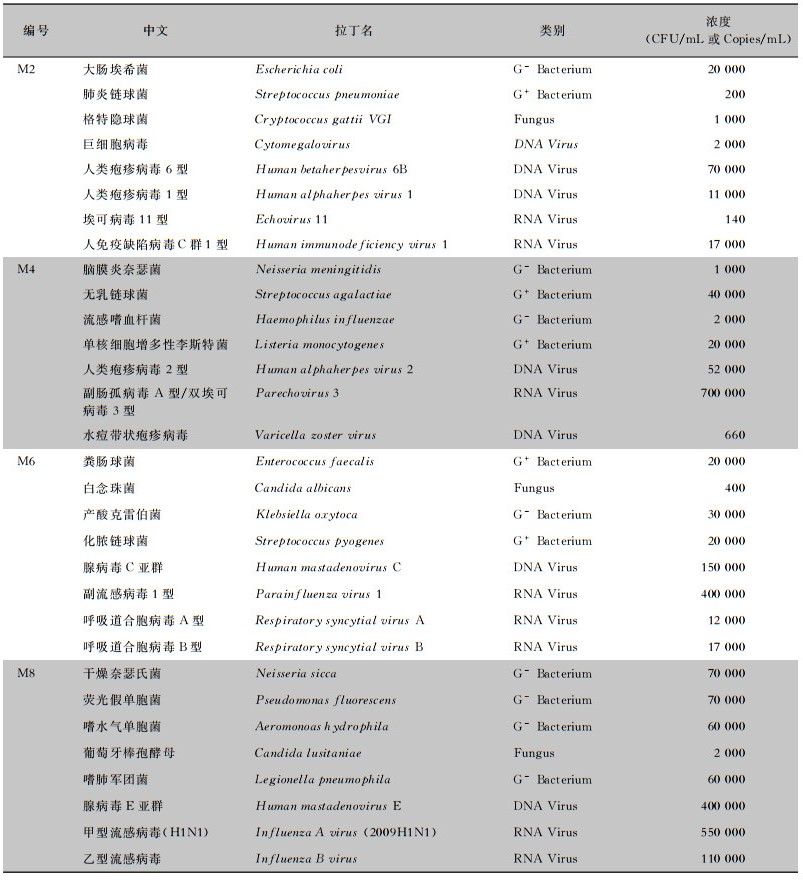
|
参考品覆盖革兰阳性菌、革兰阴性菌、真菌、DNA病毒和RNA病毒等不同种属病原体,所选病原体在病原种类、基因组大小、GC含量等方面覆盖广泛,可代表实际临床标本中病原体的多样性。D系列由人源背景1×105 cells/mL和不同病原体组成,M系列由背景菌(人乳头瘤病毒18型、吉拉尔迪贪铜菌、猪鼻支原体)和不同病原体组成,其中D0和M9是阴性参考品。D1系列有3支(D1-1、D1-2和D1-3),D2系列有3支(D2-1、D2-2和D2-3),M系类有4支(M2、M4、M6和M8)。另外,使用无菌无核酸酶水分别将D1-1和D1-2稀释100、10倍,制备成D1-1(100)和D1-2(10)。所有参考品进行随机编码,并随机分配给三家平台,见表 3。
| 表 3 参考品分配情况 Table 3 Distribution of references |
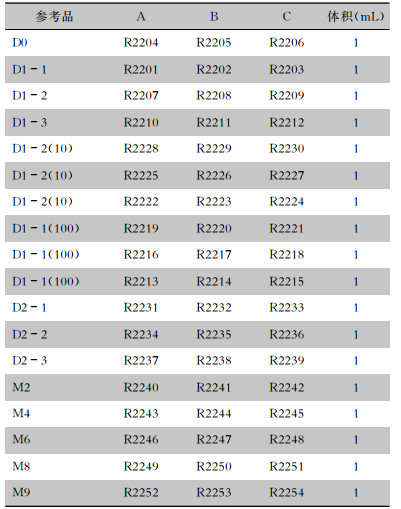
|
前瞻性收集8份临床标本,主要类型为胸/腹腔积液和肺泡灌洗液,其中4份培养检出病原菌,剩余4份培养未检出病原菌。每份临床标本分装成3管,5 mL/管,及时冻存在-80°冰箱。所有标本进行随机编号,并随机分配给不同测试平台。见表 4。
| 表 4 临床标本及分配情况 |
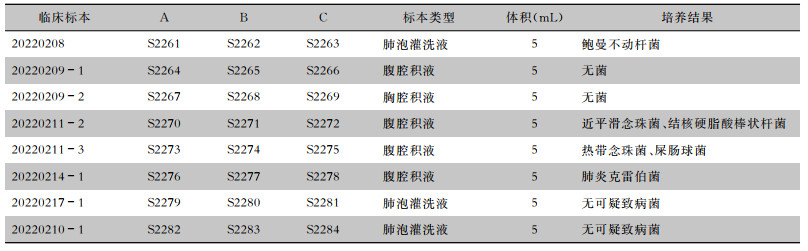
|
所有编号的参考品和临床标本分别分配给平台A、B、C,由不同平台工作人员取样,干冰运输至各自实验室安排后续样本提取、检测和出具报告。mNGS检测流程包括:首先对样本进行前处理,提取样本核酸。利用随机引物建库,对构建完成的文库进行质量评估,筛选出合格的DNA文库样本。将筛选后的DNA文库样本上机扩增测序,并对数据质量进行评估,去除低质量短片断的序列及匹配到宿主基因组的序列,最后比对到包括已知病毒、细菌、真菌和寄生虫的微生物参考基因组数据库中,检索所有序列匹配基因组目标的种属信息、匹配的序列数量、占总序列数比值等。
2 结果 2.1 检测周转时间(turn around time,TAT)研究汇总各家TAT时间,其中A平台在24 h内汇报结果,B和C平台的TAT分别为4、7 d。其中,B平台在样品DNA测序中分别做了去人源和未去人源化处理。
2.2 参考品检测性能通过研究阴性参考品和阳性参考品病原体的检出情况,评估各家mNGS检出灵敏度、特异度、阴性预测值和阳性预测值。参考品mNGS检测性能分析结果见表 5。
| 表 5 参考品mNGS检测结果汇总 Table 5 mNGS detection results of reference |
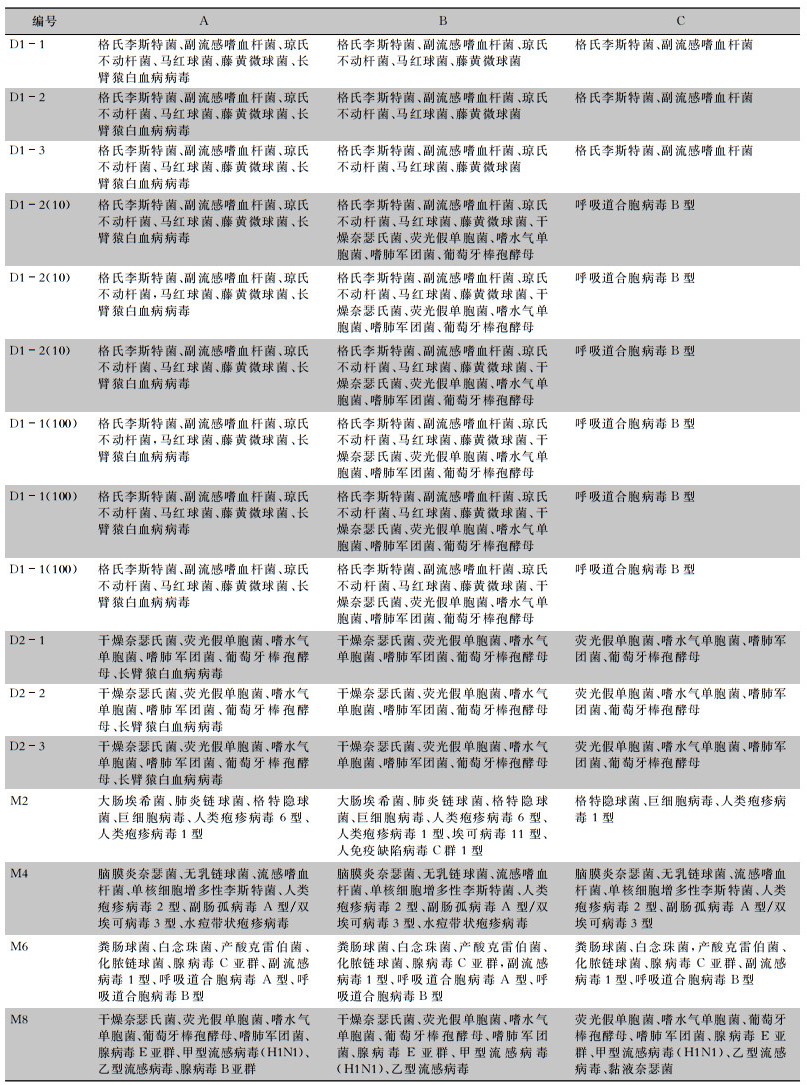
|
研究采用不同阳性参考品的不同病原体作为阳性金标准,计算不同mNGS平台对阳性参考品中不同病原体的阳性检出率(即检出灵敏度)。研究统计了91份参考品病原体,结果显示B(100%,91份)和A(97.8%,89份)的检出灵敏度远优于C(45.1%,41份)。结果见图 1。
 |
| 图 1 三家mNGS平台参考品的性能分析 Figure 1 Performance analysis on references of three mNGS platforms |
研究计算阴性参考品(D0和M9)病原体各家mNGS平台的检测情况,结果显示B和C平台无多报现象,特异度为100%,而A平台对D0多报了长臂猿白血病病毒,特异度为50%(1/2),见图 1。
2.2.3 阴性预测值在阴性参考品检测中,B和C平台都报告了D0和M9两个阴性结果,而A平台对D0多报了长臂猿白血病病毒。在阳性参考品中,A和C平台分别报了2个和50个阳性病原体为阴性。三家mNGS阴性预测值分别为B平台100%(2/2)、A平台25.0%(1/4)、C平台3.8%(2/52)。
2.2.4 阳性预测值在91个阳性参考品病原体检测中,B平台报告91个阳性和多报了30个结果(三次D1-2(10)和三次D1-1(100)重复均多报了5个病原菌),A平台报告89个阳性和多报13个阳性(M8多报了腺病毒B亚群,D系列均多报了长臂猿白血病病毒),C平台报告了41个阳性和多报3个病原体[D1-2(10)和D1-1(100)多报了呼吸道合胞病毒B型,M8多报了黏液奈瑟菌]。三家mNGS阳性预测值分别为B平台75.2%(91/121)、A平台87.3%(89/102)、C平台93.2%(41/44)。
2.3 临床标本检测性能研究以培养或2家mNGS平台同时检出的病原体为阳性金标准,分析诊断灵敏度和阳性预测值。鉴于本次研究未提供完全无菌临床体液标本,无法评估诊断特异性和阴性预测值。临床标本mNGS检测性能分析结果见表 6。
| 表 6 培养和mNGS临床标本病原体检测结果汇总 Table 6 Pathogen detection results of cultured and mNGS clinical specimens |
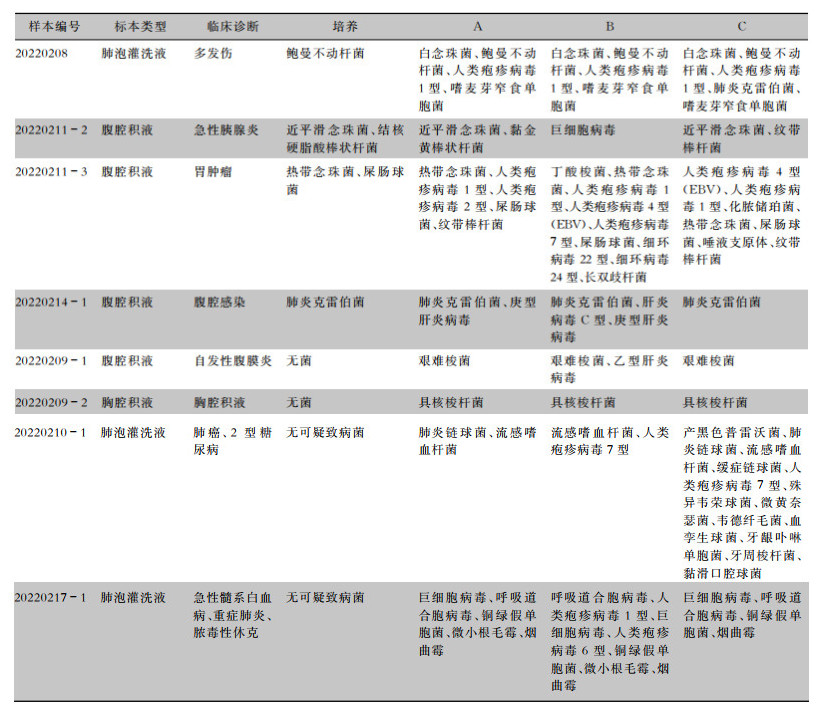
|
临床标本共检出23种病原体,不同mNGS平台的检出灵敏度分别为A平台87.0%(20种),B平台82.6%(19种)、C平台87.0%(20种),见图 2。
 |
| 图 2 三家mNGS平台临床标本的性能分析 Figure 2 Performance analysis on clinical specimens of three mNGS platforms |
mNGS相比于培养方法会发现更多的阳性病原体,本次评估研究发现不同mNGS平台在临床标本中分别报告了22个(A)、29个(B)、33个(C)阳性病原体,其中真阳性病原体分别为20个(A)、19个(B)和20个(C)。试验计算不同mNGS平台阳性预测值分别为A平台90.9%(20/22)、B平台65.5%(19/29)、C平台60.6%(20/33)。
3 讨论mNGS对于不明原因感染、混合及复杂病原体感染的诊断优势显著。2021年7月15日,国务院发布《关于支持浦东新区高水平改革开放打造社会主义现代化建设引领区的意见》[15]中提出,允许有条件的医疗机构按照相关要求开展自行研制体外诊断试剂试点。政策支持推动了mNGS实验室本地化的发展,建立完整的质量管理控制体系是重点更是难点[16]。
目前,采用mNGS进行感染性疾病诊断最大的瓶颈为污染引起的假阳性[12]。污染来源包括实验室试剂或环境中检测到的背景微生物和正常人体菌群,以及生信分析或数据库错误(例如错误注释),这些均可能导致假阳性[2, 12]。本研究在结果汇总时发现,各家在参考品检测中均出现了污染。A平台在对D系列参考品进行RNA测序时,多报了一种RNA病毒——长臂猿白血病病毒,这可能来自标本运输、核酸提取及实验室污染等过程[2, 17]。B平台在D1-1(100)和D1-2(10)重复检测样品中报告了D2系列全部的病原体,可能是实验室检测过程中污染导致。C平台在除了D1-1(100)和D1-2(10)外的其他参考品都报告了人乳头瘤病毒18型(M系列参考品的背景菌),怀疑存在标本交叉污染。因此,如何减少污染是mNGS检测技术在未来急需攻克的问题。
本研究结果显示,三家平台对细菌、真菌和病毒的检测效能也存在一定差异,可能与不同微生物种类本身结构的差异,核酸释放效率差异明显有关[4, 18]。另外,病毒的准确分型也十分考验宏基因组的生信流程[2]。对于临床标本20220211-3,B和C平台检出了人类疱疹病毒4型,也就是EB病毒,而A平台检出的是人类疱疹病毒2型。临床上这两种病毒引起的临床症状大相径庭。因此,提高病毒检测的灵敏度及特异度,仍是宏基因组临床应用的挑战之一。
本次研究评估比较了三家mNGS平台对于参考品和临床标本的检测性能。结果显示,在参考品评估中,B平台mNGS的灵敏度、特异度和阴性预测值最高,C平台mNGS的阳性预测值最高。而在临床标本评估中,A平台的灵敏度和阳性预测值的性能明显优于B和C。TAT显示A平台报告回报速度最快。三家平台均存在定植菌及背景菌被多报的现象,造成该现象可能的原因是:由于不同类型微生物基因组长度和测序平台等存在差异,无法建立统一的致病菌判断标准,导致某些背景菌被误判为致病菌。因此,在mNGS结果解读方面,实验室需综合测序目的、标本类型、技术特点等建立并验证阳性阈值及判断标准,根据微生物序列数目、基因组覆盖度、相对丰度和病原体的临床相关性等形成一套内部的结果解读规则[15],并与临床保持良好的沟通,才能得到一份合理的检测报告。
本研究也存在一定的局限性。首先,由于前瞻性收集病原菌阳性的临床血液和脑脊液标本的难度较大,本研究未开展血液和脑脊液临床标本的宏基因组评估。其次,由于临床标本的病原菌检测缺少金标准,单纯培养只能检出常见细菌和真菌,病毒和难培养的细菌无法检出,因此本研究选择了以培养或2家mNGS平台同时检出的病原体作为金标准,进而分析诊断灵敏度和阳性预测值。该方法的准确性尚缺乏定论。
感染性疾病的诊断是现今医学上的难点和重点,本研究丰富了国内宏基因组平台资料,为感染性疾病诊断新技术的进步和完善提供了一定的建议。另外,mNGS对于已知以及未知病原体都具有强大的检测能力,面对检出的大量病原体,如何正确地去看待及解读,使其更好地指导临床感染性疾病的诊治,这需要临床实验室工作人员加强相关知识的学习,联合强大的生物信息学团队,并与临床保持良好的沟通才能实现。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Gu W, Deng XD, Lee M, et al. Rapid pathogen detection by metagenomic next-generation sequencing of infected body fluids[J]. Nat Med, 2021, 27(1): 115-124. DOI:10.1038/s41591-020-1105-z |
| [2] |
刘东来, 张春涛, 王佑春, 等. 病原宏基因组高通量测序技术质量控制与评价的挑战和思考[J]. 生物工程学报, 2020, 36(12): 2598-2609. Liu DL, Zhang CT, Wang YC, et al. Challenges and considera-tions on quality control and evaluation of pathogen meta-genomic next-generation sequencing[J]. Chinese Journal of Biotechnology, 2020, 36(12): 2598-2609. DOI:10.13345/j.cjb.200377 |
| [3] |
McGill F, Heyderman RS, Panagiotou S, et al. Acute bacte-rial meningitis in adults[J]. Lancet, 2016, 388(10063): 3036-3047. DOI:10.1016/S0140-6736(16)30654-7 |
| [4] |
中华医学会检验医学分会. 高通量宏基因组测序技术检测病原微生物的临床应用规范化专家共识[J]. 中华检验医学杂志, 2020, 43(12): 1181-1195. Laboratory Medicine Society of Chinese Medical Association. Expert consensus on clinical standardized application of meta-genomics next-generation sequencing for detection of pathogenic microorganisms[J]. Chinese Journal of Laboratory Medi-cine, 2020, 43(12): 1181-1195. DOI:10.3760/cma.j.cn114452-20200903-00704 |
| [5] |
Schlaberg R, Chiu CY, Miller S, et al. Validation of meta-genomic next-generation sequencing tests for universal pathogen detection[J]. Arch Pathol Lab Med, 2017, 141(6): 776-786. DOI:10.5858/arpa.2016-0539-RA |
| [6] |
Greninger AL. The challenge of diagnostic metagenomics[J]. Expert Rev Mol Diagn, 2018, 18(7): 605-615. DOI:10.1080/14737159.2018.1487292 |
| [7] |
Gu W, Miller S, Chiu CY. Clinical metagenomic next-generation sequencing for pathogen detection[J]. Annu Rev Pathol, 2019, 14: 319-338. DOI:10.1146/annurev-pathmechdis-012418-012751 |
| [8] |
Wilson MR, Naccache SN, Samayoa E, et al. Actionable dia-gnosis of neuroleptospirosis by next-generation sequencing[J]. N Engl J Med, 2014, 370(25): 2408-2417. DOI:10.1056/NEJMoa1401268 |
| [9] |
Zheng Y, Qiu XJ, Wang T, et al. The diagnostic value of metagenomic next-generation sequencing in lower respiratory tract infection[J]. Front Cell Infect Microbiol, 2021, 11: 694756. DOI:10.3389/fcimb.2021.694756 |
| [10] |
Govender KN, Street TL, Sanderson ND, et al. Metagenomic sequencing as a pathogen-agnostic clinical diagnostic tool for infectious diseases: a systematic review and Meta-analysis of diagnostic test accuracy studies[J]. J Clin Microbiol, 2021, 59(9): e0291620. DOI:10.1128/JCM.02916-20 |
| [11] |
Han DS, Li ZY, Li R, et al. mNGS in clinical microbiology laboratories: on the road to maturity[J]. Crit Rev Microbiol, 2019, 45(5/6): 668-685. |
| [12] |
中华医学会检验医学分会临床微生物学组, 中华医学会微生物学与免疫学分会临床微生物学组, 中国医疗保健国际交流促进会临床微生物与感染分会. 宏基因组高通量测序技术应用于感染性疾病病原检测中国专家共识[J]. 中华检验医学杂志, 2021, 44(2): 107-120. Clinical Microbiology Group, Laboratory Medicine Branch, Chinese Medical Association, Clinical Microbiology Group, Microbiology and Immunology Branch, Chinese Medical Association, Clinical Microbiology and Infection Branch of China Healthcare International Exchange Promotion Association. Chinese expert consensus on metagenomics next-generation sequencing application on pathogen detection of infectious diseases[J]. Chinese Journal of Laboratory Medicine, 2021, 44(2): 107-120. DOI:10.3760/cma.j.cn114452-20201026-00794 |
| [13] |
宏基因组学测序技术在中重症感染中的临床应用共识专家组, 中国研究型医院学会脓毒症与休克专业委员会, 中国微生物学会微生物毒素专业委员会, 等. 宏基因组学测序技术在中重症感染中的临床应用专家共识(第一版)[J]. 中华危重病急救医学, 2020, 32(5): 531-536. Consensus Expert Group on the Clinical Application of Meta-genomic Sequencing Technology in Moderate and Severe In-fections, Sepsis and Shock Committee of Chinese Research Hospital Association, Microbial Toxin Committee of Chinese Society of Microbiology, et al. Expert consensus for the application of metagenomic next generation sequencing in the pathogen diagnosis in clinical moderate and severe infections (first edition)[J]. Chinese Critical Care Medicine, 2020, 32(5): 531-536. DOI:10.3760/cma.j.cn121430-20200228-00095 |
| [14] |
《中华传染病杂志》编辑委员会. 中国宏基因组学第二代测序技术检测感染病原体的临床应用专家共识[J]. 中华传染病杂志, 2020, 38(11): 681-689. Editorial Board of Chinese Journal of Infectious Diseases. Clini-cal practice expert consensus for the application of meta-genomic next generation sequencing[J]. Chinese Journal of Infectious Diseases, 2020, 38(11): 681-689. DOI:10.3760/cma.j.cn311365-20200731-00732 |
| [15] |
韩东升, 马筱玲, 吴文娟. 病原体宏基因组高通量测序医院实验室本地化之路: 现状和挑战[J]. 中华检验医学杂志, 2022, 45(2): 100-104. Han DS, Ma XL, Wu WJ. Current status and challenges of pathogen metagenomic high-throughput sequencing testing implemented in a clinical laboratory affiliated to a hospital[J]. Chinese Journal of Laboratory Medicine, 2022, 45(2): 100-104. |
| [16] |
刘东来, 沈舒, 田颖新, 等. 病原宏基因组高通量测序技术质量评价参考品的建立[J]. 分子诊断与治疗杂志, 2022, 14(5): 727-730. Liu DL, Shen S, Tian YX, et al. Establishment of reference panel for quality evaluation of pathogen metagenomic next-generation sequencing[J]. Journal of Molecular Diagnostics and Therapy, 2022, 14(5): 727-730. DOI:10.3969/j.issn.1674-6929.2022.05.004 |
| [17] |
Denner J. Transspecies transmission of gammaretroviruses and the origin of the gibbon ape leukaemia virus (GaLV) and the koala retrovirus (KoRV)[J]. Viruses, 2016, 8(12): 336. DOI:10.3390/v8120336 |
| [18] |
Liu DL, Zhou HW, Xu T, et al. Multicenter assessment of shotgun metagenomics for pathogen detection[J]. EBioMedicine, 2021, 74: 103649. DOI:10.1016/j.ebiom.2021.103649 |



