2. 宁夏病原微生物重点实验室, 宁夏 银川 750000
2. Ningxia Key Laboratory of Clinical and Pathogenic Microbiology, Yinchuan 750000, China
耐碳青霉烯类肠杆菌(carbapenem-resistant Enterobacterales, CRE)感染治疗可选择的药物非常少,病死率高,给公共卫生带来严峻挑战[1]。患者肠道CRE定植是发生CRE感染常见的来源[2],且多项研究[3-5]表明,肠道CRE定植会增加重症监护病房(intensive care unit, ICU)患者感染及死亡的风险。然而,CRE定植的机制及其发展为感染的机制尚不明确。
肠道微生物群的改变不仅与多种感染性疾病相关[6-7],而且肠道菌群失调与克罗恩病[8]和慢性阻塞性肺疾病[9]的进展相关。此外,有研究[10]证明,肠道微生物群与炎症相关,可以利用肠道微生物的特定分类和功能谱的鉴定区分细菌性肺炎和病毒性肺炎[11]。最近的一项研究[12]表明,通过改变肠道微生物的组分(粪便菌群移植)可有效去除肠道CRE定植。这些证据都提示肠道微生物可能在CRE定植及感染中扮演重要角色。因此,本研究的目的是基于16S rRNA测序分析CRE定植对ICU患者肠道微生物的影响。
1 对象与方法 1.1 研究对象收集2020年6—12月宁夏医科大学总医院入住ICU时间>48 h患者的粪便或肛拭子标本,每例收集一份,剔除同一患者的重复标本。纳入患者标准:在排除污染的情况下,粪便或肛拭子中检测出CRE但患者无感染相关的临床表现,则被认为CRE定植。排除标准:根据国家卫生部颁布的《医院感染诊断标准(试行)》[13]判定为CRE感染的患者。
1.2 研究方法 1.2.1 CRE筛查及鉴定用无菌棉拭子采集ICU患者新鲜的粪便或肛拭子标本,直接接种于含有2 μg/mL厄他培南的MacConkey平板上培养。若培养基上有菌落生长,对生长出的菌落进行分离纯化后采用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)进行菌种鉴定,并采用纸片扩散法对菌株进行药物敏感性试验;针对初步鉴定为CRE的菌株,采用聚合酶链式反应(PCR)方法扩增菌株16S rDNA片段,并采用微量肉汤稀释法最终确定CRE。PCR扩增产物测序结果在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)上进行比对。质控菌株大肠埃希菌ATCC 25922、肺炎克雷伯菌ATCC BAA-1705均来自该院医学实验中心。药敏试验结果判定标准依据美国临床实验室标准化协会(CLSI)指南[14]。引物序列为27F:AGAGTTTGATCMTGGCTCAG,1492R:GGTTACCTTGTTACGACT;扩增条件:94℃ 5 min,(94℃ 30 s,56℃ 30 s,72℃ 45 s)×30,72℃ 5 min。
1.2.2 肠道微生物群检测采用QIAamp cador Pathogen Mini Kit试剂盒提取粪便或肛拭子标本基因组DNA约30 ng,质检合格后使用其对应的融合引物进行PCR扩增,然后使用Agencourt AMPure XP磁珠对扩增产物纯化后完成建库。Agilent 2100 Bioanalyzer检测合格的文库在HiSeq平台进行16S rRNA测序。数据过滤后得到的Cleandata用于后期分析。分析时先将双末端测序得到的成对reads用FLASH软件组装成一条序列,得到高变区的Tags。再用USEARCH软件将拼接好的Tags聚类为OTU,并将其与数据库进行比对。最后,根据OTU比对和注释结果对标本进行物种注释、物种复杂度分析、组间物种差异分析,以及关联分析。此部分由深圳华大基因科技有限公司完成。
1.3 统计学方法应用R软件对微生物丰度差异及通路差异进行Wilcoxon秩和检验,P≤0.05为差异具有统计学意义。
2 结果 2.1 ICU患者肠道CRE定植共收集ICU患者粪便或肛拭子标本241份,其中CRE阳性标本17份,阳性率为7.05%。17株CRE阳性标本中大肠埃希菌9株(52.94%),肺炎克雷伯菌7株(41.18%),阴沟肠杆菌1株(5.88%)。CRE来源科室分别为综合ICU(12株)、呼吸科ICU(3株)、儿科ICU(2株),冠心病ICU、精神科ICU及急诊ICU未筛查出。见表 1。
| 表 1 肠道CRE定植菌株科室来源分布 Table 1 Department distribution of gut-colonized CRE strains |
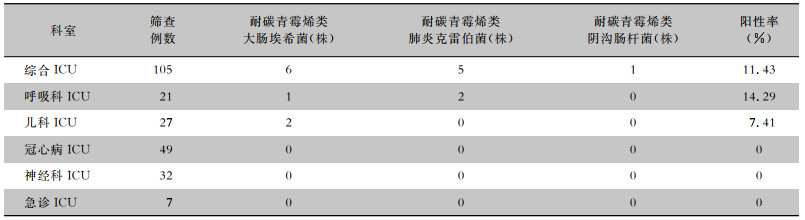
|
采用16S扩增子V3~V4区测序共分离到726 OTUs,未定植组有631 OTUs,定植组有480 OTUs,两组间共有385 OTUs,见图 1。Shannon多样性指数曲线提示数据量足够大,可以反映样品中绝大多数的微生物物种信息,见图 2。
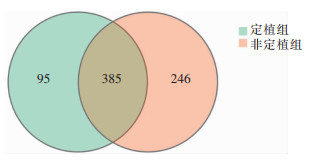 |
| 图 1 CRE定植组与非定植组Venn图 Figure 1 Venn diagram of CRE colonized- and non-colonized groups |
 |
| 图 2 CRE定植组与非定植组Alpha多样性稀释曲线图 Figure 2 Dilution curves of Alpha diversity in CRE colonized- and non-colonized groups |
物种组成分析显示,检出OTUs绝大多数属于厚壁菌门、变形菌门、拟杆菌门、疣微菌门及放线菌门,见图 3a。CRE定植组与非定植组共有OTUs主要属于肠球菌科、拟杆菌科、毛螺菌科和紫单胞菌科。定植组肠杆菌科、肠球菌科和拟杆菌科的优势更明显,而非定植组瘤胃菌科、普雷沃菌科、肽链球菌科及梭状芽孢杆菌科的优势更明显,见图 3b。
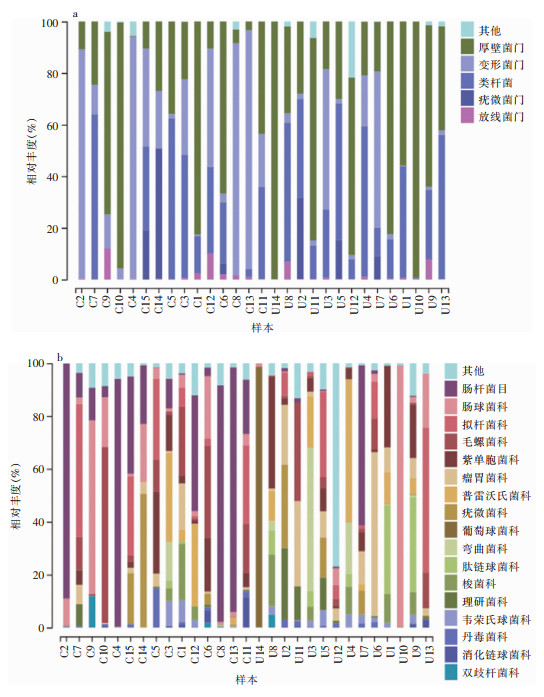 |
| 注:a为门水平上物种组成柱形图;b为科水平上物种组成柱形图。 图 3 CRE定植组与未定植组物种组成柱形图 Figure 3 Histogram of species composition of CRE colonized- and non-colonized groups |
物种差异分析显示,CRE定植组γ‐变形菌纲、拟杆菌纲、梭状芽孢杆菌相对丰度较大,而非定植组梭状芽孢杆菌、拟杆菌纲相对丰度较大;Wilcoxon秩和检验后得到两组间γ-变形菌纲(W=193.000,P<0.001,FDR=0.004)和互营养菌纲(W=37.500,P=0.001,FDR=0.018)相对丰度差异有统计学意义,见图 4。
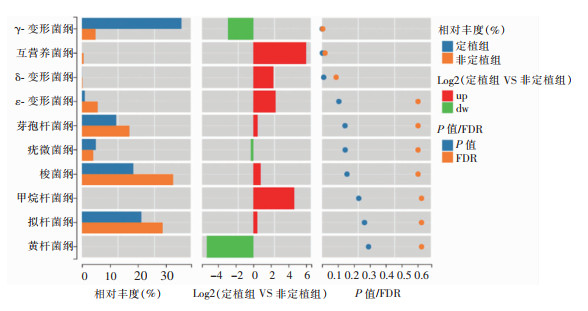 |
| 图 4 物种差异分析图 Figure 4 Analysis chart of species differences |
在种水平上,选取丰度前10的物种对比作图发现,CRE定植前后,肺炎克雷伯菌(W=195.000,P<0.001)、解糖肠球菌(W=153.000,P=0.038)及弯曲杆菌(W=63.500,P=0.050)差异有统计学意义,见图 5。
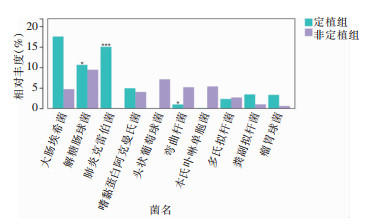 |
| 图 5 关键物种差异比较柱形图 Figure 5 Histogram comparison of differences in key species |
KEGG功能差异分析结果显示,CRE定植组与未定植组富集到的KEGG通路非常相似,碳水化合物代谢、氨基酸代谢、辅助因子和维生素的代谢相对丰度较高,而传染病、免疫系统、消化系统和神经退行性疾病相对丰度较低。对CRE定植组与未定植组的各个通路分别进行Wilcoxon秩和检验,未发现ICU患者肠道CRE定植对肠道微生物KEGG功能通路有显著影响,见图 6。
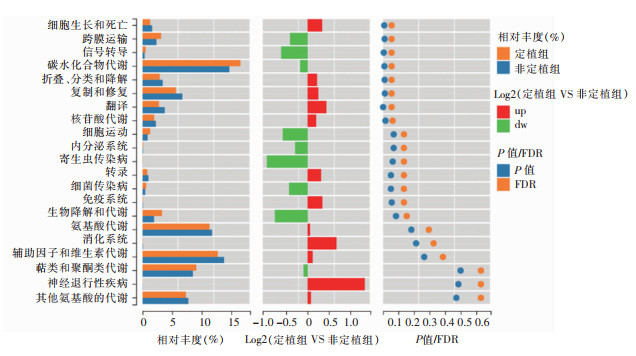 |
| 图 6 KEGG功能差异分析图 Figure 6 Analysis chart of KEGG functional difference |
患者肠道CRE定植是发生CRE感染的一个重要危险因素[15]。在流行地区,住院患者的CRE定植率为3%~ 7%[16],而ICU患者的CRE定植率及分离率高于普通病房[17]。本研究中ICU总体CRE定植率为7.05%,低于国内文献[18-19]报道的10.90%和20.00%。呼吸科ICU与综合ICU定植率最高,儿科ICU次之,而冠心病ICU、神经科ICU和急诊ICU未发现CRE定植患者,这可能与各科室床位数及采样例数有关。此外,由于ICU工作人员多且流动性大,ICU患者病情危重、免疫力低下、侵入性治疗操作多,这些原因导致ICU患者较普通科室患者更易发生感染。通过对ICU患者进行CRE主动筛查,并对筛查阳性患者严格执行感染预防与控制干预措施可以显著减少ICU患者CRE感染的发生[16]。本研究筛查到的CRE为大肠埃希菌(52.94%)、肺炎克雷伯菌(41.18%)和阴沟肠杆菌(5.88%),与本地区报道的CRE流行病学调查[20]结果基本一致。
研究[21]报道,健康个体往往具有较高的微生物多样性。另有研究[22]将CRE携带者与健康人群进行对比,发现CRE携带者的肠道菌群多样性降低。本研究发现在ICU患者中,CRE非定植组比定植组具有更多的OTUs,CRE非定植组的微生物群组分更丰富。人体肠道微生物的组成相对恒定,以厚壁菌门和拟杆菌门为主,其次是放线菌门和疣微菌门[23]。本研究发现在ICU患者中,无论是CRE定植还是非定植患者,除了上述4类细菌以外,变形菌门都占有重要比例。有研究[24]指出变形菌门的丰度与炎症相关,可能在一定程度上解释了这一现象。相对于CRE定植组,本研究在非定植组中发现瘤胃菌科更具优势,而这种菌对抗感染有帮助[21]。
为了探究CRE定植对ICU患者肠道微生态结构的影响,本研究对物种差异进行了分析,研究结果显示两组间γ-变形菌纲和互营养菌纲相对丰度存在统计学差异(P<0.05)。γ-变形菌纲的军团菌,如嗜肺军团菌(军团菌病的病原体)和贝氏柯克斯菌(Q热的病原体),被广泛认为是人类病原体[25]。而目前对于互营养菌纲知之甚少。本研究显示,在种水平上,肺炎克雷伯菌、解糖肠球菌及弯曲杆菌具有统计学差异(P<0.05),与Sindi等[26]的研究结果类似,Sindi等[26]将造成这一差异的原因归结为抗菌药物的使用。研究[22]表明,某些肠杆菌目家族成员比其他成员更容易驻留在人类肠道中,一些细菌聚集在一起,形成一个由代谢产物驱动的复杂的网络互惠共生[27],而菌群的差异可以决定微生物群特异性代谢能力的改变,这些改变与全身感染的患病率增加有关[22]。本研究通过对KEGG功能差异进行分析,发现CRE定植组与非定植组各个通路的相对丰度高度相似,CRE定植组在营养物质代谢方面相对丰度较高,而与疾病相关的功能通路相对丰度很低。对两组间差异进行统计学分析,并未发现肠道CRE定植对ICU患者肠道微生物KEGG功能通路有显著影响,后期可通过增加样本量,同时对患者临床信息进行分析以期有新的发现。
本研究表明,肠道CRE定植组患者肠道微生物多样性降低,且与非定植组患者相比,肺炎克雷伯菌、解糖肠球菌及弯曲杆菌丰度上具有统计学差异。这些结果为以特定的细菌表达谱来区分CRE是否定植提供理论基础,但是否可以将以上几种菌作为特定的标志,以及通过改变细菌组成是否会影响CRE定植都有待进一步研究。另外,通过重新引入特定菌株和/或用益生菌或粪便菌群移植来改变肠道微生态,从而影响CRE定植及感染状态,也为CRE的预防及治疗提供了新的思路。
另外,本研究存在一定的局限性,收集的病例数相对较少且缺乏对临床数据的分析。后期可通过增加研究病例,同时结合患者临床数据如基础疾病及抗菌药物使用情况等进行分析,以期发现更加有意义的成果,为临床CRE诊治和防控提供支持依据。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Shrivastava SR, Shrivastava PS, Ramasamy J. World Health Organization releases global priority list of antibiotic-resistant bacteria to guide research, discovery, and development of new antibiotics[J]. J Med Soc, 2018, 32(1): 76-77. DOI:10.4103/jms.jms_25_17 |
| [2] |
Martin RM, Cao J, Brisse S, et al. Molecular epidemiology of colonizing and infecting isolates of Klebsiella pneumoniae[J]. mSphere, 2016, 1(5): e00261-16. |
| [3] |
Dickstein Y, Edelman R, Dror T, et al. Carbapenem-resistant Enterobacteriaceae colonization and infection in critically ill patients: a retrospective matched cohort comparison with non-carriers[J]. J Hosp Infect, 2016, 94(1): 54-59. DOI:10.1016/j.jhin.2016.05.018 |
| [4] |
Madney Y, Aboubakr S, Khedr R, et al. Carbapenem-resis-tant Enterobacteriaceae (CRE) among children with cancer: predictors of mortality and treatment outcome[J]. Antibiotics (Basel), 2023, 12(2): 405. DOI:10.3390/antibiotics12020405 |
| [5] |
Freire MP, Song ATW, Oshiro ICV, et al. Surgical site infection after liver transplantation in the era of multidrug-resistant bacteria: what new risks should be considered?[J]. Diagn Microbiol Infect Dis, 2021, 99(1): 115220. DOI:10.1016/j.diagmicrobio.2020.115220 |
| [6] |
Li NN, Xu SQ, Zhang SS, et al. MSI2 deficiency in ILC3s attenuates DSS-induced colitis by affecting the intestinal micro-biota[J]. Front Immunol, 2022, 13: 963379. |
| [7] |
Pandrea I, Brooks K, Desai RP, et al. I've looked at gut from both sides now: gastrointestinal tract involvement in the patho-genesis of SARS-CoV-2 and HIV/SIV infections[J]. Front Immunol, 2022, 13: 899559. DOI:10.3389/fimmu.2022.899559 |
| [8] |
Ma XZ, Lu XJ, Zhang WY, et al. Gut microbiota in the early stage of Crohn's disease has unique characteristics[J]. Gut Pathog, 2022, 14(1): 46. DOI:10.1186/s13099-022-00521-0 |
| [9] |
Li NJ, Dai ZL, Wang Z, et al. Gut microbiota dysbiosis contributes to the development of chronic obstructive pulmonary disease[J]. Respir Res, 2021, 22(1): 274. DOI:10.1186/s12931-021-01872-z |
| [10] |
Wellman AS, Metukuri MR, Kazgan N, et al. Intestinal epithelial sirtuin 1 regulates intestinal inflammation during aging in mice by altering the intestinal microbiota[J]. Gastroentero-logy, 2017, 153(3): 772-786. DOI:10.1053/j.gastro.2017.05.022 |
| [11] |
Gierse LC, Meene A, Skorka S, et al. Impact of pneumococcal and viral pneumonia on the respiratory and intestinal tract microbiomes of mice[J]. Microbiol Spectr, 2023, 11(3): e0344722. DOI:10.1128/spectrum.03447-22 |
| [12] |
Shin J, Lee JH, Park SH, et al. Efficacy and safety of fecal microbiota transplantation for clearance of multidrug-resistant organisms under multiple comorbidities: a prospective compa-rative trial[J]. Biomedicines, 2022, 10(10): 2404. DOI:10.3390/biomedicines10102404 |
| [13] |
中华人民共和国卫生部. 医院感染诊断标准(试行)摘登(1)[J]. 新医学, 2005, 36(8): 495. Ministry of Health of the Ministry of Health of the People's Republic of China. Hospital infection diagnostic standards (trial) excerpt (1)[J]. New Chinese Medicine, 2005, 36(8): 495. |
| [14] |
Humphries R, Bobenchik AM, Hindler JA, et al. Overview of changes to the Clinical and Laboratory Standards Institute Performance Standards for Antimicrobial Susceptibility Testing, M100, 31st edition[J]. J Clin Microbiol, 2021, 59(12): e0021321. DOI:10.1128/JCM.00213-21 |
| [15] |
Zhu RP, Xu XH, Lian SY, et al. Intestinal colonization with carbapenem-resistant Enterobacteriaceae in acute leukemia patients: risk factors and molecular characteristics[J]. Infect Drug Resist, 2022, 15: 4275-4283. DOI:10.2147/IDR.S376413 |
| [16] |
Bhargava A, Hayakawa K, Silverman E, et al. Risk factors for colonization due to carbapenem-resistant Enterobacteriaceae among patients exposed to long-term acute care and acute care facilities[J]. Infect Control Hosp Epidemiol, 2014, 35(4): 398-405. DOI:10.1086/675614 |
| [17] |
杨玉琪, 刘家云, 徐修礼, 等. 某院耐碳青霉烯类肠杆菌科细菌的临床分布特点及药物敏感性分析[J]. 检验医学与临床, 2021, 18(1): 1-5, 11. Yang YQ, Liu JY, Xu XL, et al. Analysis on clinical distribution characteristics and drug susceptibility of carbapenem-resistant Enterobacteriaceae bacteria in a hospital[J]. Laboratory Medicine and Clinic, 2021, 18(1): 1-5, 11. |
| [18] |
陈美恋, 王守军, 匡季秋, 等. 重症监护病区CRE主动筛查及其效果评价[J]. 中华医院感染学杂志, 2017, 27(18): 4123-4126. Chen ML, Wang SJ, Kuang JQ, et al. Active screening of CRE in intensive care unit and its effect[J]. Chinese Journal of Nosocomiology, 2017, 27(18): 4123-4126. |
| [19] |
杨璐, 李云轩, 宋婉红, 等. 肠道定植耐碳青霉烯肠杆菌科细菌(CRE)与感染病原菌关系[J]. 中国抗生素杂志, 2022, 47(10): 1077-1083. Yang L, Li YX, Song WH, et al. The relationship between carbapenem-resistant Enterobacteriaceae(CRE) colonized in intestinal tract and pathogenic bacteria of infection[J]. Chinese Journal of Antibiotics, 2022, 47(10): 1077-1083. |
| [20] |
兰敏, 赵志军, 康宇婷, 等. 耐碳青霉烯类肠杆菌目细菌分子流行病学特征及耐药性[J]. 中国感染控制杂志, 2022, 21(11): 1053-1059. Lan M, Zhao ZJ, Kang YT, et al. Molecular epidemiological characteristics and drug resistance of carbapenem-resistant Enterobacterales[J]. Chinese Journal of Infection Control, 2022, 21(11): 1053-1059. |
| [21] |
Ubeda C, Pamer EG. Antibiotics, microbiota, and immune defense[J]. Trends Immunol, 2012, 33(9): 459-466. |
| [22] |
Korach-Rechtman H, Hreish M, Fried C, et al. Intestinal dysbiosis in carriers of carbapenem-resistant Enterobacteriaceae[J]. mSphere, 2020, 5(2): e00173-20. |
| [23] |
Qin JJ, Li RQ, Raes J, et al. A human gut microbial gene cata-logue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. |
| [24] |
Niechcial A, Schwarzfischer M, Wawrzyniak M, et al. Spermidine ameliorates colitis via induction of anti-inflammatory macrophages and prevention of intestinal dysbiosis[J]. J Crohns Colitis, 2023: jjad058. Epub ahead of print. DOI: 10.1093/ecco-jcc/jjad058.
|
| [25] |
Duron O, Doublet P, Vavre F, et al. The importance of revisi-ting legionellales diversity[J]. Trends Parasitol, 2018, 34(12): 1027-1037. |
| [26] |
Sindi AA, Alsayed SM, Abushoshah I, et al. Profile of the gut microbiome containing carbapenem-resistant Enterobacteriaceae in ICU patients[J]. Microorganisms, 2022, 10(7): 1309. |
| [27] |
Willing BP, Russell SL, Finlay BB. Shifting the balance: antibiotic effects on host-microbiota mutualism[J]. Nat Rev Microbiol, 2011, 9(4): 233-243. |



