肺炎克雷伯菌(Klebsiella pneumoniae, Kp),是一种有荚膜的革兰阴性粗短杆菌,属于肠杆菌目克雷伯菌属,是临床上重要的机会致病菌。临床上Kp因其高耐药率及高传播性成为引起社区、医院感染的重要致病菌,约占克雷伯菌属分离率的95%以上[1]。研究[1-2]表明Kp具有广泛的生态分布、多样的DNA组成、丰富的抗菌药物耐药基因(AMR)和极高的质粒丰度,是耐药基因在环境与革兰阴性菌之间传播的主要媒介。喹诺酮耐药基因首次分离于Kp菌株;耐碳青霉烯类基因KPC、NDM-1和OXA-48等通过Kp在环境与临床病原菌之间进行传播。随着产超广谱β-内酰胺酶(ESBLs)、碳青霉烯类耐药(CRKp)、高毒力菌株(hvKp)的出现大大加剧了Kp感染治疗的难度。随着基因组学及高通量测序技术的发展,依据Kp临床分离株核苷酸间的差异可分为三个系统群:肺炎克雷伯菌(KpⅠ)、类肺炎克雷伯菌(KpⅡ)和变栖克雷伯菌(KpⅢ),KpⅡ又可进一步分为KpⅡ-A和KpⅡ-B。国内外研究发现KpⅠ、KpⅡ和KpⅢ系统群菌株的生态分布、基因型、耐药性、毒力特征以及致病性存在显著差异,因此菌株的分群研究对个体化精准医疗的推进和针对性治疗指南制定极为有益。本文通过总结近几年国内外的最新研究以及临床报道,对KpⅠ、KpⅡ、KpⅢ的特征进行综述,为感染性疾病精准医疗、医院Kp感染性疾病防控指南制定提供理论依据。
1 Kp临床分离株感染现状全球医院面临的抗菌药物耐药性危机是由ESKAPE病原体(屎肠球菌、金黄色葡萄球菌、肺炎克雷伯菌、鲍曼不动杆菌、铜绿假单胞菌、肠杆菌属细菌)驱动的,这些病原体是导致大多数患者感染后使用抗菌药物治疗失败的原因[3]。2019年中国细菌耐药监测网数据显示Kp居临床分离的革兰阴性菌第二位,仅次于大肠埃希菌,同时耐碳青霉烯类肺炎克雷伯菌(CRKp)检出率自2013年以来持续升高[4]。Kp可引起呼吸系统、泌尿生殖系统以及伤口的感染,也可导致菌血症和肝脓肿等;免疫力低下的群体(如新生儿、老年人、肿瘤、自身免疫性疾病群体和有创操作的患者等)易感。一项涵盖中国14个省25所三级甲等医院的多中心临床研究[3]显示,在664个临床分离标本中,耐碳青霉烯类肠杆菌目细菌(CRE)中Kp占比为73.9%,耐药现状严峻。
在分子生物学领域,当DNA杂交结果大于70%时考虑为同一物种。系统进化树研究[5]表明,临床分离鉴定的Kp可分为三个系统群:KpⅠ、KpⅡ和KpⅢ,系统群间核苷酸差异为3%~4%。KpⅠ菌株是临床常见的病原菌;约94%的KpⅡ菌株分离自人类(50%以上为肠道定植);KpⅢ菌株能够固氮,广泛存在于植物、水体、土壤、食草生物的黏膜表面,与环境密切相关;Martínez-Romero等[6]通过计算机分析和基因组数据Jaccard分析证明了KpⅡ和KpⅢ从环境感染人的可能性。KpⅠ、KpⅡ和KpⅢ三种细菌感染的流行病学存在差异,关于Kp临床分离株分群的流行病学研究有助于临床科室防控工作的实施,使广大患者受益。
2 KpⅠ、KpⅡ和KpⅢ的鉴定临床常用的细菌鉴定技术是基于生化试验的VITEK-2鉴定仪和基于蛋白质组学的基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS)。基于生化试验的鉴定系统尚不能实现KpⅠ、KpⅡ和KpⅢ的分群,因此临床上使用药敏卡进行鉴定会降低KpⅡ和KpⅢ分离率。MALDI-TOF-MS系统通过将待鉴定菌株质谱图与数据库中的质谱图对比实现菌种鉴定,因此鉴定菌种范围主要与装配的鉴定文库相关。目前装配有Bruker文库6.0.0.0版本(包含KpⅠ、KpⅡ和KpⅢ菌株标准谱图)的MALDI-TOF-MS系统可实现KpⅠ、KpⅡ和KpⅢ的分群[7-8]。但受标准谱图对应的菌株数据量的限制,包含KpⅠ、KpⅡ和KpⅢ菌株标准谱图的细菌文库在临床尚未广泛应用。基于以上现状,KpⅡ和KpⅢ在临床分离率较低。
目前KpⅠ、KpⅡ和KpⅢ鉴别的分子生物学技术包括PCR法、进化树分析及基因测序等。PCR法主要是基于染色体上β- 内酰胺酶多样性设计的多重PCR和rpoB基因的系统发育树分析来鉴定KpⅠ(blaSHV)、KpⅡ(blaOKP)和KpⅢ(blaLEN),由于部分KpⅢ携带的β-内酰胺酶为blaOKP,因此该技术不能很好地区分KpⅡ和KpⅢ[9]。基因组学分析主要通过全基因测序分析(WGS)进行细菌病原体的鉴定分群,此外KlebSeq、单核苷酸多态性(SNPs)、ANI均可用于克雷伯菌属筛选和流行病学监测[10]。但目前PCR法、进化树分析和基因测序等分子生物学手段主要用于科学研究,尚未在临床普及。
3 KpⅠ的药物敏感性、菌株毒力与致病性 3.1 药物敏感性早在20世纪60年代就发现Kp临床分离株携带对青霉素天然耐药的A类染色体编码的β-内酰胺酶基因[11]。KpⅠ的blaSHV耐药基因介导菌株对氨苄西林等天然耐药。20世纪80年代德国从一例ICU患者分离的Kp中发现了第一个可水解包括第三代头孢菌素类及氨曲南在内的抗生素的ESBLs基因(blaSHV),随后ESBLs编码基因blaSHV-2、blaTEM-3、blaCTX-M被发现[3]。根据世界卫生组织(WHO)的报告[10],携带ESBLs的KpⅠ在世界范围内覆盖率达到50%,社区分离率达到30%,严重威胁人类健康。碳青霉烯类药物作为产ESBLs菌株的常规治疗药物,在药物选择的压力下,碳青霉烯类耐药菌株随之出现,其中CRKp是常见的耐碳青霉烯类肠杆菌目细菌(CRE)。CRKp出现的主要机制是产碳青霉烯酶,细菌基因组基因突变、药物外排泵的改变、AmpC酶和ESBLs异常表达也可导致CRKp出现。全基因组测序发现[12]CRKp基因组包括16个碳青霉烯类耐药基因(12个位于质粒中,4个位于染色体中)。碳青霉烯酶是指能够明显水解亚胺培南或美罗培南等碳青霉烯类抗生素的一类β-内酰胺酶,按照Ambler分子分类方法,可分为A、B、D 3类酶。其中A类、D类为丝氨酸酶,属于Bush分群中的第2f和2d亚组;B类为金属酶,属于Bush分群中的第3组,可由染色体、质粒或转座子介导。blaKPC是KpⅠ最常见的碳青霉烯酶,属A类丝氨酸碳青霉烯酶,目前我国流行的主要是blaKPC-1和blaKPC-2[13];D类酶blaOXA-48与CRKp密切相关,blaOXA-48阳性的KpⅠ对亚胺培南高度耐药[14];B类金属酶blaIMP、blaNDM-1和blaVIM等均与碳青霉烯类耐药KpⅠ相关[13]。除此之外,KpⅠ中外排泵OqxAB和AcrAB的表达和/或活化与多种抗菌药物(如氟喹诺酮类、呋喃妥因、替格环素、氯霉素和碳青霉烯类) 的耐药性相关;外膜OmpK36的特异性突变或丢失在很大程度上加剧了KpⅠ碳青霉烯类耐药的问题[3],并可以使MIC远高于单独的碳青霉烯酶水平,增加临床治疗难度。
3.2 菌株毒力与致病性Kp是备受关注的医院获得性病原体,已鉴定的毒力因子包括荚膜、脂多糖、铁载体、菌毛[2]。荚膜作为KpⅠ最重要的毒力因子之一,主要帮助细菌抵御吞噬和血清杀菌作用。基于血清学鉴定可区分78种血清型,其中K1和K2型与高侵袭性疾病和致病性相关,在高毒性克隆菌株中高度保守[15-17]。荚膜调控基因rmpA、rmpA2和magA等与KpⅠ高黏液表型密切相关[17]。脂多糖(LPS)即内毒素,是革兰阴性菌外膜的主要成分,可参与TLR介导的信号转导,LPS修饰可帮助细菌逃逸免疫系统识别,wcaG基因与脂多糖及荚膜的形成相关[18]。铁载体系统包括铁螯合分子以及用于内化的表面受体,可以竞争性地从宿主蛋白质或其他来源清除铁。Kp中有四个铁载体系统:entero- bactin、yersiniabactin、aerobactin和salmochelin(分别由Ent、ybt、iuc和iro编码)[19]。Ent编码的核心结构可破坏中性粒细胞的除铁功能并诱导炎症反应;iro介导Ent末端糖基化,使细胞保持除铁活性;ybt和iuc的亲铁性比Ent低。KpⅠ中常见的菌毛有Ⅰ型菌毛(fm)和Ⅲ型菌毛(mrk),Ⅲ型菌毛和脂多糖是生物膜中最重要的表面结构,Ⅰ型菌毛的转录对菌体抗吞噬过程至关重要,有助于细菌免受宿主免疫反应的攻击、吞噬[19]。此外,KpⅠ外排泵也可能发挥毒力作用,如AcrAB可影响KpⅠ经小鼠呼吸系统感染后的毒力。近来,以ST23为代表的高黏液表型为特征的KpⅠ的出现,给临床治疗带来极大挑战,通常该菌株携带四种获得性铁载体系统以及rmpA和rmpA2编码基因[20]。高毒性肺炎克雷伯菌(hvKpⅠ)具有丰富的毒力基因和铁载体系统、对大多数抗菌药物敏感,常引起肝脓肿、脑膜炎、肺炎等疾病[21]。以往的研究[22]认为高毒力与高耐药之间负性相关,随着国内报道的同时具有高毒性、耐多药性和高度传播性的ST11型耐碳青霉烯类高毒性肺炎克雷伯菌(CR-hvKp)的发现,证明了高毒性与高耐药性可同时存在,严重威胁人类健康。
4 KpⅡ的药物敏感性、菌株毒力与致病性 4.1 药物敏感性Kp临床分离株可根据基因组间差异分为三群,第二群对应于类肺炎克雷伯菌(K. quasipneumoniae, KpⅡ),KpⅡ可分为两个亚群:K. quasipneumoniae subsp. quasipneumoniae (KpⅡ-A)和K.quasipneumoniae subsp. similipneumoniae(KpⅡ-B)[12]。KpⅡ染色体上携带blaOKP基因,介导菌株对青霉素天然耐药,blaOKP-A和blaOKP-B分别对应于KpⅡ-A和KpⅡ-B。与KpⅠ相比,KpⅡ药物敏感性较高,其中一部分菌株能够产ESBLs,碳青霉烯酶编码基因blaNDM和blaOXA-181曾在2株KpⅡ菌株中检测到[23-24]。Fuga等[25]报道在巴西地区2014年从一例患者的肛拭子中分离出含有blaNDM-1基因的KpⅡ-B,2017年从一例灌注液标本中检出含blaKPC-2基因的KpⅡ-A。2016年从马来西亚一例患者分离的blaOXA181型CRKpⅡ中blaCTX-M-15基因和OMPK37孔蛋白缺失[24];2019年沙特阿拉伯[23]分离的KpⅡ中的blaNDM-1由mcr-9质粒携带;2019年尼日利亚一家医院新生儿病区中携带blaNDM-5基因的KpⅡ的出现和多黏菌素耐药的blaKPC-9基因型KpⅡ菌株的报道[26],揭示了质粒在耐药性传播中的重要性。2020年中国从某新生儿病区[27]分离的ST2727菌株,分类上属于KpⅡ-B,导致一例新生儿败血症死亡,同一病区多例患者感染,虽然ST2727菌株为非高毒力非耐药性菌株,但其暴发性流行揭示了KpⅡ在医院环境中的诊断和监测价值。巴西里约热内卢的一项研究发现ST3397临床分离株是毒力基因magA缺乏、blaNDM-blaCTX-M15多重耐药的KpⅡ-B菌株,表现出对全部抗菌药物耐药,蝇蚊在耐药性传递中的作用不可忽视。
4.2 菌株毒力与致病性KpⅡ菌株致病因素包括荚膜、脂多糖、铁载体、菌毛等,KpⅡ最初被认为是一种共生的肠道定植菌群,不具有致病性,然而,近年来的研究表明KpⅡ在临床上可发挥致病性,感染最常见的来源是泌尿道、血液、呼吸道及部分创口等。该菌株致病性与KpⅠ类似,但毒力基因丰度低。2015年报道了第一例KpⅡ-B高黏液菌株,该菌株为血流感染来源,且该菌株中高黏液调控基因rmpA和rmpA2缺乏[5]。2016年,巴西分离了一株产生ESBLs、黏菌素耐药、高毒性和高黏液KpⅡ菌株[12],同年Breurec等[28]报道的K1型KpⅡ菌株具有高毒性,含有结合元件ICEKp1编码的毒力因子、铁载体系统和rmpA基因,可引起感染性肝脓肿,但其毒力耐药基因的水平转移机制尚未清楚。临床研究表明高毒力KpⅡ相关的社区获得性肺炎(CAP)病死率相对较高,一株高毒力KpⅡ临床分离株菌毛调控基因mrkB和mrkC丰度较高,但铁载体系统缺乏,这与高毒力KpⅠ存在差异[28],推测KpⅡ菌株具有独立的毒力调节系统,但有待研究。
5 KpⅢ的药物敏感性、菌株毒力与致病性 5.1 药物敏感性KpⅢ菌株具有固氮作用和促进植物生长的作用,栖息环境多样,可广泛存在于植物、昆虫、动物、人体、土壤、水和生态环境中[29],2004年,综合考虑菌株基因多态性和生态环境多态性将KpⅢ命名为变栖克雷伯菌(K. variicola, KpⅢ)。与其他Kp菌株一样,KpⅢ染色体上携带耐药基因blaLEN,对青霉素类天然耐药。随着KpⅢ从临床和环境中分离率增高,产ESBLs和碳青霉烯类耐药变栖克雷伯菌(CRKpⅢ)的报告也在增加,KpⅢ在环境与临床之间传递耐药基因的潜力逐渐被发现。blaCTX-M是目前ESBLs基因中流行最广泛的类型,以其对头孢噻肟的超强水解活性而得名,Farzana等[30]从一株KpⅢ检出了抗菌药物耐药基因blaNDM-1和blaCTX-M-15。随后ESBLs基因blaSHV-5、blaSHV-12、blaSHV-30等均从KpⅢ菌株中检出[7]。Kumwenda等[31]从CRKpⅢ菌株中检测到KPC-2和NDM-1型基因,随后报道从CRKpⅢ菌株内检测到NDM-5。Zurfluh等[32]首次在从亚洲进口到瑞士的香菜中检测到含有OXA-181的CRKpⅢ,碳青霉烯类耐药基因NDM-9、GES-6、GES-2等在河水或污水来源的CRKpⅢ中检出[33]。报道[7]显示,碳青霉烯酶NDM-1和β-内酰胺酶CTX-M-15与变栖克雷伯菌的高耐药和高毒力相关。在2018年,Guo等[34]在一株KpⅢ-X39发现了染色体和质粒编码的CTX-M-24基因。综上所述,KpⅢ菌株的耐药现状不容忽视。
5.2 菌株毒力与致病性KpⅢ与环境关系密切,其临床重要性越来越凸显。2018年Dong等[35]研究表明,KpⅢ-X39可定植于玉米的根、茎和叶并在特定条件下感染人致病。目前已发现的KpⅢ毒力因子主要为荚膜、脂多糖、铁载体、菌毛,在细菌的入侵、致病、免疫逃逸中发挥作用。2015年从一例住院的老年患者体内分离出第一株高毒力变栖克雷伯菌株(hvKpⅢ),该菌株基因组DNA可检测到经典的毒力因子(铁载体、铁摄取系统、菌毛和黏附素),在5%羊血的琼脂平板上拉丝试验阳性[6]。分离于中国的一株血培养来源的黏菌素耐药高毒力KpⅢ (WCHKV030666)中检出来源于质粒的黏液表型毒力因子(rmpA和rmpA2)和3种载体系统(yersiniabactin、aerobactin和salmochelin)[35]。值得注意的是,一株高毒力高耐药KpⅢ并未检测到黏液表型毒力因子(rmpA和rmpA2),但其致病性远高于ST23型高毒力肺炎克雷伯菌[36]。2018年我国四川分离了一株血液来源的高毒力高耐药菌株(WCHKV030666),同年在孟加拉国新生儿病房检出一株暴发流行的高毒力多重耐药变栖克雷伯菌(ST171)导致54.5%的新生儿死亡[29]。推测特殊的遗传背景、特殊的毒力系统可能与高毒力KpⅢ毒力系统的形成相关。现有临床数据表明KpⅢ是一种机会致病菌,可引起血流相关感染、呼吸系统感染、新生儿暴发感染、原发性牙根管感染[7]。临床上分离的KpⅢ菌株70%左右来自尿路相关感染,KpⅢ相关血流感染致死率较KpⅠ和KpⅡ高[29]。由于KpⅢ临床感染现状严重,在感染研究以及感染控制领域关注度逐渐增高,是一种新发现的人类病原体,因此菌株特性、流行病特征、致病性研究有助于更好地控制感染性疾病的发生。
6 结语KpⅡ和KpⅢ是从Kp中分离出来的新的物种,可追溯到600万年前。KpⅠ、KpⅡ和KpⅢ是三种不同的细菌,受临床现有检测技术的限制,KpⅡ和KpⅢ临床分离率较低,临床报道较少。KpⅠ、KpⅡ和KpⅢ定植和感染的流行病学大不相同,菌株毒力、耐药性及致病特点存在差异,见表 1。Brisse等[37]研究发现,KpⅠ耐药性最强,KpⅡ次之,KpⅢ耐药性最弱。KpⅠ是临床常见的病原菌,属机会致病菌,可引起社区获得性或医院获得性感染,感染部位广泛,携带有丰富的毒力和耐药基因,部分菌株表现为产ESBLs或碳青霉烯类药物耐药,耐药现状严重。KpⅡ最常见于泌尿道和呼吸道感染,部分菌株能够产ESBLs,94%的KpⅡ分离自人类,其中50%以上来自肠道定植,远高于KpⅠ(24%)和KpⅢ(39%)[24]。随着研究[38]的深入,KpⅢ被分为经典变栖克雷伯菌(cKpⅢ)、高黏液表型变栖克雷伯菌(hmvKpⅢ)与高毒力变栖克雷伯菌(hvKpⅢ),部分菌株可以引起肺炎、血流感染、脑膜炎、尿道感染等,KpⅢ相关血流感染致死率较KpⅠ和KpⅡ高2倍左右。因其独特的毒力表型,微生物学者Rodríguez-Medina等[7]认为KpⅢ是一种新发现的人类病原体,其流行病特征与肺炎克雷伯菌不同,可能存在有独立的毒力系统。因此,KpⅠ、KpⅡ和KpⅢ的毒力特征及耐药性和致病性研究可为感染的多维度控制模型和措施,为有效的目标性治疗策略提供理论和实验室依据。
| 表 1 KpⅠ、KpⅡ和KpⅢ的区别 |
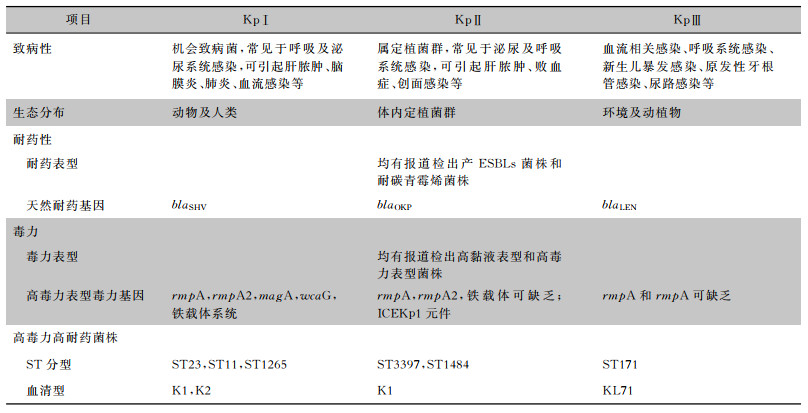
|
KpⅠ、KpⅡ、KpⅢ具有不同的生态分布,KpⅡ、KpⅢ具有与KpⅠ不同的毒力调节系统和独特的基因分布,与其耐药特点、菌株致病性与致病能力密切相关。相信随着基因测序技术的开展和普及,越来越多的KpⅡ、KpⅢ将会被鉴定出来,对KpⅡ、KpⅢ的认识将不断加深,促进精准医疗和个体化医疗的发展。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Wyres KL, Holt KE. Klebsiella pneumoniae as a key traffic-ker of drug resistance genes from environmental to clinically important bacteria[J]. Curr Opin Microbiol, 2018, 45: 131-139. DOI:10.1016/j.mib.2018.04.004 |
| [2] |
Martin RM, Bachman MA. Colonization, infection, and the accessory genome of Klebsiella pneumoniae[J]. Front Cell Infect Microbiol, 2018, 8: 4. DOI:10.3389/fcimb.2018.00004 |
| [3] |
Wang GY, Zhao G, Chao XY, et al. The characteristic of viru-lence, biofilm and antibiotic resistance of Klebsiella pneumo-niae[J]. Int J Environ Res Public Health, 2020, 17(17): 6278. DOI:10.3390/ijerph17176278 |
| [4] |
全国细菌耐药监测网. 全国细菌耐药监测网2014-2019年老年患者常见临床分离细菌耐药性监测报告[J]. 中国感染控制杂志, 2021, 20(2): 112-123. China Antimicrobial Resistance Surveillance System. Antimicrobial resistance of clinically isolated bacteria from elderly patients: surveillance report from China Antimicrobial Resistance Surveillance System in 2014-2019[J]. Chinese Journal of Infection Control, 2021, 20(2): 112-123. |
| [5] |
Holt KE, Wertheim H, Zadoks RN, et al. Genomic analysis of diversity, population structure, virulence, and antimicrobial resistance in Klebsiella pneumoniae, an urgent threat to public health[J]. Proc Natl Acad Sci U S A, 2015, 112(27): E3574-E3581. |
| [6] |
Martínez-Romero E, Rodríguez-Medina N, Beltrán-Rojel M, et al. Klebsiella variicola and Klebsiella quasipneumoniae with capacity to adapt to clinical and plant settings[J]. Salud Publica Mex, 2018, 60(1): 29-40. |
| [7] |
Rodríguez-Medina N, Barrios-Camacho H, Duran-Bedolla J, et al. Klebsiella variicola: an emerging pathogen in humans[J]. Emerg Microbes Infect, 2019, 8(1): 973-988. DOI:10.1080/22221751.2019.1634981 |
| [8] |
Rodrigues C, Passet V, Rakotondrasoa A, et al. Identification of Klebsiella pneumoniae, Klebsiella quasipneumoniae, Klebsiella variicola and related phylogroups by MALDI-TOF mass spectrometry[J]. Front Microbiol, 2018, 9: 3000. DOI:10.3389/fmicb.2018.03000 |
| [9] |
Barrios-Camacho H, Silva-Sánchez J, Cercas-Ayala E, et al. PCR system for the correct differentiation of the main bacterial species of the Klebsiella pneumoniae complex[J]. Arch Microbiol, 2021, 204(1): 73. |
| [10] |
Long SW, Linson SE, Ojeda Saavedra M, et al. Whole-genome sequencing of human clinical Klebsiella pneumoniae isolates reveals misidentification and misunderstandings of Klebsiella pneumoniae, Klebsiella variicola, and Klebsiella quasipneumoniae[J]. mSphere, 2017, 2(4): e00290-17. |
| [11] |
Ashurst JV, Dawson A. Klebsiella pneumonia[M]//StatPearls[Internet]. Treasure Island (FL): StatPearls Publi-shing, 2022.
|
| [12] |
Ferreira ML, Araújo BF, Cerdeira LT, et al. Genomic features of a clinical ESBL-producing and colistin-resistant hypermucoviscous K. quasipneumoniae subsp. similipneumoniae from Brazil[J]. Braz J Infect Dis, 2019, 23(3): 207-209. DOI:10.1016/j.bjid.2019.05.003 |
| [13] |
Meng XJ, Yang J, Duan JP, et al. Assessing molecular epidemiology of carbapenem-resistant Klebsiella pneumoniae (CR-KP) with MLST and MALDI-TOF in central China[J]. Sci Rep, 2019, 9(1): 2271. DOI:10.1038/s41598-018-38295-8 |
| [14] |
Atterby C, Osbjer K, Tepper V, et al. Carriage of carbapenemase -and extended-spectrum cephalosporinase-producing Escherichia coli and Klebsiella pneumoniae in humans and livestock in rural Cambodia; gender and age differences and detection of blaOXA-48 in humans[J]. Zoonoses Public Health, 2019, 66(6): 603-617. DOI:10.1111/zph.12612 |
| [15] |
Harada S, Ishii Y, Saga T, et al. Molecular epidemiology of Klebsiella pneumoniae K1 and K2 isolates in Japan[J]. Diagn Microbiol Infect Dis, 2018, 91(4): 354-359. DOI:10.1016/j.diagmicrobio.2018.03.010 |
| [16] |
Bialek-Davenet S, Criscuolo A, Ailloud F, et al. Genomic defi-nition of hypervirulent and multidrug-resistant Klebsiella pneumoniae clonal groups[J]. Emerg Infect Dis, 2014, 20(11): 1812-1820. DOI:10.3201/eid2011.140206 |
| [17] |
Gu DX, Dong N, Zheng ZW, et al. A fatal outbreak of ST11 carbapenem-resistant hypervirulent Klebsiella pneumoniae in a Chinese hospital: a molecular epidemiological study[J]. Lancet Infect Dis, 2018, 18(1): 37-46. DOI:10.1016/S1473-3099(17)30489-9 |
| [18] |
Candan ED, Aks z N. Klebsiella pneumoniae: characteristics of carbapenem resistance and virulence factors[J]. Acta Biochim Pol, 2015, 62(4): 867-874. DOI:10.18388/abp.2015_1148 |
| [19] |
Paczosa MK, Mecsas J. Klebsiella pneumoniae: going on the offense with a strong defense[J]. Microbiol Mol Biol Rev, 2016, 80(3): 629-661. DOI:10.1128/MMBR.00078-15 |
| [20] |
Sanikhani R, Moeinirad M, Shahcheraghi F, et al. Molecular epidemiology of hypervirulent Klebsiella pneumoniae: a systematic review and Meta-analysis[J]. Iran J Microbiol, 2021, 13(3): 257-265. |
| [21] |
Gonzalez-Ferrer S, Peñaloza HF, Budnick JA, et al. Finding order in the chaos: outstanding questions in Klebsiella pneumoniae pathogenesis[J]. Infect Immun, 2021, 89(4): e00693-20. |
| [22] |
Furlan JPR, Gallo IFL, de Campos TA, et al. Genomic chara-cterization of a multidrug-resistant and hypermucoviscous/hypervirulent Klebsiella quasipneumoniae subsp. similipneumoniae ST4417 isolated from a sewage treatment plant[J]. Microb Drug Resist, 2020, 26(11): 1321-1325. DOI:10.1089/mdr.2019.0417 |
| [23] |
Faccone D, Martino F, Albornoz E, et al. Plasmid carrying mcr-9 from an extensively drug-resistant NDM-1-producing Klebsiella quasipneumoniae subsp. quasipneumoniae clinical isolate[J]. Infect Genet Evol, 2020, 81: 104273. DOI:10.1016/j.meegid.2020.104273 |
| [24] |
Lau MY, Ponnampalavanar S, Lee WS, et al. First detection of Klebsiella quasipneumoniae producing OXA-181 carbapenemase in Malaysia[J]. J Infect Chemother, 2020, 26(10): 1058-1061. DOI:10.1016/j.jiac.2020.05.009 |
| [25] |
Fuga B, Cerdeira L, Andrade F, et al. Genome sequences of clinical isolates of NDM-1-producing Klebsiella quasipneumo-niae subsp. similipneumoniae and KPC-2-producing Klebsiella quasipneumoniae subsp. quasipneumoniae from Brazil[J]. Microbiol Resour Announc, 2020, 9(10): e00089-20. |
| [26] |
Shankar C, Karunasree S, Manesh A, et al. First report of whole-genome sequence of colistin-resistant Klebsiella quasipneumoniae subsp. similipneumoniae producing KPC-9 in India[J]. Microb Drug Resist, 2019, 25(4): 489-493. DOI:10.1089/mdr.2018.0116 |
| [27] |
Perlaza-Jiménez L, Wu Q, Torres VVL, et al. Forensic genomics of a novel Klebsiella quasipneumoniae type from a neonatal intensive care unit in China reveals patterns of colonization, evolution and epidemiology[J]. Microb Genom, 2020, 6(10): mgen000433. |
| [28] |
Breurec S, Melot B, Hoen B, et al. Liver abscess caused by infection with community-acquired Klebsiella quasipneumoniae subsp. quasipneumoniae[J]. Emerg Infect Dis, 2016, 22(3): 529-531. DOI:10.3201/eid2203.151466 |
| [29] |
Barrios-Camacho H, Aguilar-Vera A, Beltran-Rojel M, et al. Molecular epidemiology of Klebsiella variicola obtained from different sources[J]. Sci Rep, 2019, 9(1): 10610. DOI:10.1038/s41598-019-46998-9 |
| [30] |
Farzana R, Jones LS, Rahman MA, et al. Outbreak of hypervirulent multidrug-resistant Klebsiella variicola causing high mortality in neonates in Bangladesh[J]. Clin Infect Dis, 2019, 68(7): 1225-1227. DOI:10.1093/cid/ciy778 |
| [31] |
Kumwenda GP, Sugawara Y, Abe R, et al. First identification and genomic characterization of multidrug-resistant carbapenemase-producing Enterobacteriaceae clinical isolates in Malawi, Africa[J]. J Med Microbiol, 2019, 68(12): 1707-1715. DOI:10.1099/jmm.0.001087 |
| [32] |
Zurfluh K, Poirel L, Nordmann P, et al. First detection of Klebsiella variicola producing OXA-181 carbapenemase in fresh vegetable imported from Asia to Switzerland[J]. Antimicrob Resist Infect Control, 2015, 4: 38. DOI:10.1186/s13756-015-0080-5 |
| [33] |
Mondal AH, Siddiqui MT, Sultan I, et al. Prevalence and diversity of blaTEM, blaSHV and blaCTX-M variants among multidrug resistant Klebsiella spp. from an urban riverine environment in India[J]. Int J Environ Health Res, 2019, 29(2): 117-129. DOI:10.1080/09603123.2018.1515425 |
| [34] |
Guo YT, Zhai Y, Zhang Z, et al. Complete genomic analysis of a kingdom-crossing Klebsiella variicola isolate[J]. Front Microbiol, 2018, 9: 2428. DOI:10.3389/fmicb.2018.02428 |
| [35] |
Dong N, Lin DC, Zhang R, et al. Carriage of blaKPC-2 by a virulence plasmid in hypervirulent Klebsiella pneumoniae[J]. J Antimicrob Chemother, 2018, 73(12): 3317-3321. |
| [36] |
de Campos TA, de Almeida FM, de Almeida APC, et al. Multidrug-resistant (MDR) Klebsiella variicola strains isolated in a Brazilian hospital belong to new clones[J]. Front Microbiol, 2021, 12: 604031. DOI:10.3389/fmicb.2021.604031 |
| [37] |
Brisse S, van Himbergen T, Kusters K, et al. Development of a rapid identification method for Klebsiella pneumoniae phylogenetic groups and analysis of 420 clinical isolates[J]. Clin Microbiol Infect, 2004, 10(10): 942-945. DOI:10.1111/j.1469-0691.2004.00973.x |
| [38] |
Garza-Ramos U, Moreno-Dominguez S, Hernández-Castro R, et al. Identification and characterization of imipenem-resistant Klebsiella pneumoniae and susceptible Klebsiella variicola isolates obtained from the same patient[J]. Microb Drug Resist, 2016, 22(3): 179-184. DOI:10.1089/mdr.2015.0181 |



