Wh ole-genome sequencing and colistin resistance of carbapenem-resistant Klebsiella pneumoniae
-
摘要:
目的 通过全基因组测序分析临床耐碳青霉烯类肺炎克雷伯菌(CRKP)的分子流行病学及黏菌素耐药基因,为临床诊疗提供参考。 方法 收集2021—2023年安徽省某三级甲等综合医院住院患者临床标本分离的57株CRKP,对所有菌株进行药敏试验。采用全基因测序技术分析CRKP菌株多位点序列分型、荚膜血清型、耐药基因和毒力基因,并对所有菌株序列进行单核苷酸多态性分析,采用聚合酶链式反应(PCR)方法扩增与黏菌素耐药相关基因。 结果 57株CRKP对除替加环素外的14种抗菌药物均为耐药。测序结果显示,93.0%(53/57)的CRKP携带blaKPC-2,ST11型CRKP菌株检出率最高(51/57,89.5%)。单核苷酸多态性聚类分析表明,57株CRKP分为11个克隆群,其中有4个克隆群均为ST11-KL64型CRKP。40株(70.2%)CRKP携带多种毒力基因。5株黏菌素耐药CRKP,其耐药机制是在mgrB基因70位点处插入一个ISKpn26元件。 结论 CRKP菌株以产KPC-2 ST11-KL64型为主要流行型,在重症监护病房有播散性流行。ISKpn26元件插入引起mgrB基因突变与该地区CRKP黏菌素耐药有关。 Abstract:Objective To analyze the molecular epidemiology and colistin-resistant genes of carbapenem-resistant Klebsiella pneumoniae (CRKP) by whole-genome sequencing, and to provide reference for clinical diagnosis and treatment. Methods 57 CRKP strains isolated from clinical specimens of hospitalized patients in a tertiary general first-class hospital in Anhui Province from 2021 to 2023 were collected and antimicrobial susceptibility testing was performed. Multilocus sequence typing, capsule serotype, resistance genes, and virulence genes of CRKP strains were analyzed by whole-genome sequencing technique, and single nucleotide polymorphism analysis was conducted on sequences of all strains. Colistin resistance-related genes were amplified by polymerase chain reaction (PCR). Results 57 CRKP strains exhibited resistance to 14 antimicrobial agents, with the exception of tigecycline. The sequencing results showed that 93.0% (53/57) of CRKP carried blaKPC-2, and the ST11 type CRKP strain had the highest detection rate (51/57, 89.5%). Single nucleotide polymorphism clustering analysis showed that the 57 CRKP strains were divided into 11 clone groups, of which 4 clone groups were all ST11-KL64 type CRKP. 40 (70.2%) CRKP strains carried multiple virulence genes. Five strains of CRKP were colistin-resistant strains, the resistance mechanism involved the insertion of ISKpn26 element at site 70 of the mgrB gene. Conclusion The CRKP strain is primarily characterized by the production of KPC-2 ST11-KL64, with disseminated transmission in intensive care unit. The insertion of ISKpn26 element leading to mgrB gene mutation is related to resistance of CRKP to colistin in this region. -
肺炎克雷伯菌(Klebsiella pneumoniae, KP)是一种临床上常见的革兰阴性条件病原菌,可感染人类和多种家畜,是医疗保健相关感染和新生儿败血症的主要病原菌[1]。在健康人群中,KP主要定植于胃肠道,口咽部浓度较低[2]。在免疫功能低下或免疫缺陷的患者中,KP可侵入血液或组织并引起多种感染,包括尿路感染、菌血症、肺炎和肝脓肿[3]。此外,KP是呼吸机相关肺炎(ventilator-associated pneumonia, VAP)和重症监护病房(intensive care unit, ICU)获得性肺炎的主要病原体[4],因此KP受到广泛关注。
碳青霉烯类药物被认为是治疗严重KP感染的有效抗生素。然而,耐碳青霉烯类肺炎克雷伯(carbapenem-resistant Klebsiella pneumoniae, CRKP)因其快速传播而被认为是一个重大的公共卫生问题[5]。2022年我国KP对亚胺培南和美罗培南的平均耐药率分别为22.6%、24.2%,blaKPC和blaNDM编码的KP碳青霉烯酶(KPC)和新德里金属β-内酰胺酶(NDM)是全球大多数国家导致KP耐碳青霉烯类药物的最常见因素,而苯唑西林酶-48(oxacillinase-48)在非洲最常见[6-8]。此外,美国和中国均有报道,blaKPC和blaNDM在KP中共存[9-10]。虽然黏菌素被认为是治疗临床多重耐药KP感染的最后一道防线,但黏菌素耐药已成为世界范围内的一个重要问题[11]。已有研究[12]报道,KP对黏菌素耐药,其耐药性是通过遗传变异介导的相关染色体突变。鉴于多重耐药菌感染临床治疗的障碍和对公共卫生的负面影响,了解KP的全基因组特征有助于更全面地评估其风险[13]。
全基因组测序技术现已广泛应用于细菌学研究[14]。细菌基因组的功能注释可以在遗传水平上产生丰富的数据[15]。该技术在监测病原菌基因组进化和研究致病机制方面发挥着积极作用。本研究对安徽某省级三级甲等综合医院2021—2023年临床分离CRKP菌株全基因组进行测序,分析菌株克隆关系、耐药性和毒力基因分布,以期为制定控制CRKP传播的有效策略和降低医院重症感染发病率提供重要依据。
1. 对象与方法
1.1 研究对象
收集2021年1月1日—2023年12月31日安徽省某三级甲等综合医院住院患者临床标本分离的非重复KP 1 022株,对所有KP临床分离株进行传代培养,通过药敏试验筛选出57株CRKP作为研究对象。以上菌株均在-80℃保存。
1.2 研究方法
1.2.1 细菌鉴定及药敏试验
所有KP均采用德国布鲁克基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)仪鉴定。使用美国BD Phoenix M50全自动微生物药物敏感性分析仪及配套药敏卡分析各种抗菌药物的最低抑菌浓度(minimum inhibitory concentration, MIC)。质控菌株为KP ATCC 700603和KP ATCC BAA-1705。药敏试验结果参照2023年美国临床实验室标准化协会(CLSI) M100文件进行判读。替加环素药敏结果的判读按照美国食品药品监督管理局(Food and Drug Administration, FDA)指南:MIC≤2 μg/mL,敏感;4 μg/mL,中介;≥8 μg/mL,耐药。黏菌素药敏结果判读按照欧洲药敏试验委员会(European Committee on Antimicrobial Susceptibility Testing, EUCAST)指南(第10版):MIC>2 μg/mL,耐药;≤2 μg/mL,敏感。
1.2.2 拉丝试验
拉丝试验用于识别KP高黏液性表型[16]。将接种KP菌株的哥伦比亚血琼脂培养皿置于37℃培养24 h后,使用一次性接种环拉伸KP菌落的黏液线。如果黏液线长度>5 mm,则为拉丝试验阳性。
1.2.3 细菌全基因组测序、组装和注释
对收集的CRKP临床分离株进行全基因组测序。将CRKP的单菌落置于卢尼亚-贝尔塔尼(Luria-Bertani, LB)肉汤中,37℃摇动过夜。采用经少量修饰的十六烷基三甲基溴化铵(cetyltrimethylammonium bromide, CTAB)方法提取基因组DNA[17],使用TruSeq DNA样品制备试剂盒(Illumina,美国)和模板制备试剂盒(Pacific Biosciences,美国)测定DNA的浓度、质量和完整性,并生成测序文库。随后,由上海生物技术公司使用Illumina NovaSeq 6000平台进行基因组测序。使用AdapterRemoval[18]和SOAPec[19]去除适配器污染和数据过滤后进行数据组装。过滤后的读数由SPAdes[20]和A5-miseq[21]组装以构建支架和重叠群。
1.2.4 测序结果分析
将测序结果上传至相关网站进行分析。使用ResFinder(http://genepi.food.dtu.dk/resfinder)鉴定耐药基因,应用https://cge.food.dtu.dk/services/MLST/分析多位点序列分型(Multilocus Sequence Typing, MLST)。使用http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi在线数据库分析相关毒力基因,并将携带两种以上毒力基因的CRKP定义为耐碳青霉烯类高毒力肺炎克雷伯菌(carbapenem-resistant hypervirulent Klebsiella pneumoniae, CR-hvKP)。
1.2.5 单核苷酸多态性(single-nucleotide polymorphism, SNP)分析
将所有菌株序列提交CSI Phylogeny (https://cge.food.dtu.dk/services/CSIPhylogeny/)进行SNP分析。将分析结果导入FigTree 1.4.3软件(http://tree.bio.ed.ac.uk/software/fifigtree/)生成最大似然树,根据近与远的关系对菌株进行分组。进化树使用iTOL[22](https://itol.embl.de)美化加工。
1.2.6 黏菌素耐药相关基因检测和分析
检测CRKP菌株质粒介导的黏菌素耐药基因mcr-1~9,以及染色体介导的双组分系统(phoP/Q和pmrA/B)、mgrB基因。采用聚合酶链式反应(PCR)扩增黏菌素耐药相关基因[23-25](见表 1)。PCR扩增产物经琼脂糖凝胶电泳显示并送到生工生物工程(上海)股份有限公司测序,测序结果随后使用国家生物信息中心网站上的基本局部比对搜索工具(blaST)程序进行分析,插入序列(insertion sequence, IS)通过IS finder网站进行评估。
表 1 黏菌素耐药相关基因引物序列及扩增产物大小Table 1 Primer sequences of colistin resistance-related genes and size of amplified products基因 序列(5’-3’) 产物大小(bp) 参考文献 基因 序列(5’-3’) 产物大小(bp) 参考文献 mcr-1 AGTCCGTTTGTTCTTGTGGC 320 [23] mcr-8 TTGTCGTCGTGGGCGAAAC 514 [23] AGATCCTTGGTCTCGGCTTG CTGTCGCAAGTTGGGCTAAAG mcr-2 AGCCGAGTCTAAGGACTTGATGAATTTG 576 [23] mcr-9 CGGCGAACTACGCTTACAG 465 [23] GCGGTATCGACATCATAGTCATCTTG CGCACAGTTTCGGGTTATCAC mcr-3 AAATAAAAATTGTTCCGCTTATG 929 [23] phoP ATTGAAGAGGTTGCCGCCCGC 136 [24] AATGGAGATCCCCGTTTTT GCTTGATCGGCTGGTCATTCACC mcr-4 AATTGTCGTGGGAAAAGCCGC 1 062 [23] phoQ ATATGCTGGCGAGATGGGAAAACGG 138 [24] CTGCTGACTGGGCTATTACCGTCAT CCAGCCAGGGAACATCACGCT mcr-5 GTGAAACAGGTGATCGTGACTTACCG 271 [23] pmrA TACGCCGAAAGAGTATGCCC 170 [24] CGTGCTTTACACCGATCATGTGCT GGATCCGCGATTTGCCAATC mcr-6 ACTGACCAAGCCGAGTCTAAG 259 [23] pmrB TGCCAGCTGATAAGCGTCTT 95 [24] GCATCACGGGATTGACATAGC TTCTGGTTGTTGTGCCCTTC mcr-7 GCGACCTCCTACCTGAATG 345 [23] mgrB AAGGCGTTCATTCTACCACC 253 [25] CCCTTTGGCGACGACTTTG TTAAGAAGGCCGTGCTATCC 2. 结果
2.1 CRKP感染患者临床特征
临床分离的57株CRKP,患者主要为男性(71.9%),年龄≥60岁的患者占56.1%,其中≥75岁的患者占22.8%,中位年龄66岁。CRKP感染患者主要来自ICU(52.6%)和呼吸内科(21.1%)。标本来源以呼吸道和血标本为主,分别占56.1%、24.6%。CRKP患者的人口和临床统计学特征见表 2。表 2
表 2 CRKP患者的人口和临床统计学特征Table 2 Demographic and clinical statistic characteristics of CRKP patients临床特征 例数(n=57) 百分比(%) 年龄≥60岁 32 56.1 性别 男性 41 71.9 女性 16 28.1 标本类型 痰 22 38.6 血 14 24.6 气管抽吸物 6 10.5 腹腔积液/腹腔引流液 6 10.5 支气管肺泡灌洗液 4 7.0 尿 3 5.3 穿刺液 2 3.5 病房 ICU 30 52.6 呼吸内科 12 21.1 感染病科 4 7.0 神经外科 4 7.0 血液内科 3 5.3 肾脏内科 2 3.5 其他病房 2 3.5 基础疾病 呼吸系统疾病 32 56.1 消化系统疾病 8 14.0 神经系统疾病 5 8.8 肿瘤 4 7.0 血液病 3 5.3 心血管疾病 2 3.5 泌尿系统疾病 2 3.5 其他疾病 1 1.8 2.2 CRKP菌株耐药表型分析
57株CRKP对多种抗菌药物耐药,所有菌株对碳青霉烯类、β-内酰胺类抗生素以及头孢哌酮/舒巴坦耐药,对环丙沙星和左氧氟沙星耐药率分别为91.2%、87.7%,对妥布霉素和阿米卡星的耐药率分别为21.2%、15.8%。见表 3、4。
表 3 57株CRKP药敏试验结果[株(%)]Table 3 Antimicrobial susceptibility testing results of 57 CRKP strains (No. of isolates [%])抗菌药物 R I S 哌拉西林他/唑巴坦 45(78.9) 5(8.8) 7(12.3) 头孢他啶 57(100) 0(0) 0(0) 头孢曲松 57(100) 0(0) 0(0) 头孢吡肟 57(100) 0(0) 0(0) 头孢哌酮/舒巴坦 57(100) 0(0) 0(0) 氨曲南 4(7.0) 0(0) 53(93.0) 厄他培南 57(100) 0(0) 0(0) 亚胺培南 57(100) 0(0) 0(0) 美罗培南 57(100) 0(0) 0(0) 妥布霉素 12(21.1) 0(0) 45(78.9) 阿米卡星 9(15.8) 0(0) 48(84.2) 替加环素 0(0) 0(0) 57(100) 环丙沙星 52(91.2) 1(1.8) 4(7.0) 左氧氟沙星 50(87.7) 0(0) 7(12.3) 黏菌素 5(8.8) 0(0) 52(91.2) 注:R为耐药;S为敏感;I为中介。 表 4 5株黏菌素耐药CRKP的MIC值(μg/mL)Table 4 MIC values of 5 colistin-resistant CRKP strains (μg/mL)抗菌药物 KP02 KP17 KP20 KP46 KP47 哌拉西林/他唑巴坦 ≥128 ≥128 ≥128 ≥128 ≥128 头孢他啶 ≥64 ≥64 ≥64 ≥64 ≥64 头孢曲松 ≥64 ≥64 ≥64 ≥64 ≥64 头孢吡肟 ≥32 ≥32 ≥32 ≥32 ≥32 头孢哌酮/舒巴坦 ≥64 ≥64 ≥64 ≥64 ≥64 氨曲南 ≥16 ≥16 ≥16 ≥16 ≥16 厄他培南 ≥8 ≥8 ≥8 ≥8 ≥8 亚胺培南 ≥16 ≥16 ≥16 ≥16 ≥16 美洛培南 ≥16 ≥16 ≥16 ≥16 ≥16 妥布霉素 ≤1 ≥16 ≥16 ≥16 ≥16 阿米卡星 ≤2 ≥64 ≥64 ≥64 ≥64 替加环素 1 1 1 1 1 环丙沙星 ≥4 ≥4 ≥4 ≥4 ≥4 左氧氟沙星 ≥8 ≥8 ≥8 ≥8 ≥8 黏菌素 16 32 8 16 16 2.3 CRKP单核苷酸多态性分析
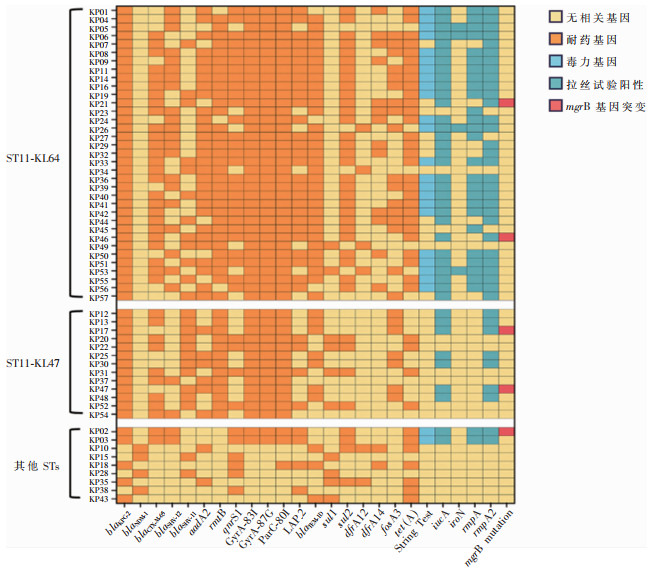
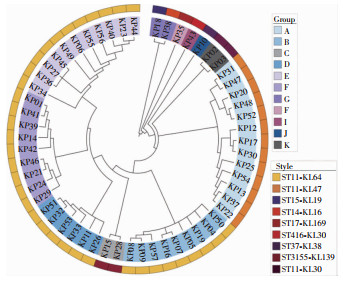
全基因组测序和核心基因组系统发育分析结果显示,57株菌株以ST11型CRKP菌株为主(51株,89.5%),分别是ST11-KL64、ST11-KL47、ST11-KL30。其中ST11-KL64型CRKP形成B、D、E、F四个克隆群,从2021年12月—2023年8月由外科ICU、内科ICU和神经外科等科室检出。13株ST11-KL47型CRKP形成克隆群A,该克隆群菌株均是2021年3—5月从外科ICU患者标本中分离。同时还发现其他ST型别呈散在分布。见图 1。
2.4 CRKP耐药和毒力基因分布情况
57株CRKP均携带碳青霉烯类耐药基因,以携带blaKPC-2(53/57,93.0%)最为常见,其次是blaNDM-1(7.0%,4/57)。此外,53株(93.0%)CRKP菌株携带5种以上耐药基因。菌株iucA、iroN、rmpA和rmpA2毒力基因检测显示,40株(70.2%)携带两种及以上毒力基因,均为CR-HvKP,其中4株携带4种毒力基因。见图 2。
2.5 黏菌素耐药相关基因的分子分析
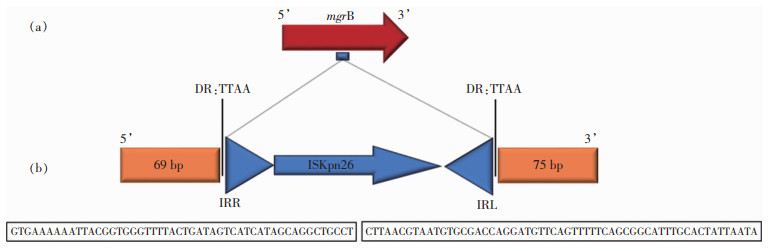
40株CR-hvKP中,5株对黏菌素耐药。为探讨潜在的黏菌素耐药机制,研究分析pmrA、pmrB、phoP、phoQ和mgrB基因的核苷酸序列,并与质控菌株KP ATCC 700603和1株黏菌素敏感KP临床菌株的基因序列进行比较,均未发现mcr-1~9阳性和pmrA、pmrB、phoP、phoQ相关基因突变,但5株黏菌素耐药CR-hvKP mgrB基因均被插入相同的1 200 bp的ISKpn26序列,且均是插在相同的位置,即mgrB基因序列的69和70个核苷酸之间,并且方向相同。见图 3。
3. 讨论
CRKP对全世界公共卫生安全构成重大威胁,了解本地CRKP克隆株流行病学数据,对于制定和评估相关临床治疗方案必不可少。本研究前瞻性调查2021—2023年安徽某三级甲等教学医院收集的57株CRKP菌株,报告其分子流行特征和相关基因分布。本研究中,患者年龄≥60岁者占56.1%,≥75岁以上的患者占22.8%,可能与老年患者免疫功能下降有关。耐药表型结果显示,CRKP对碳青霉烯类、头孢菌素和氟喹诺酮类药物的耐药率较高,对替加环素和黏菌素的耐药率较低。
MLST是一种基于核苷酸序列分型的方法,可以表明细菌之间的遗传关系[26],了解CRKP菌株的进化起源和传播。本研究中,ST11仍是最常见序列类型,占89.5%(51/57),表明ST11型产KPC-2 KP克隆正在以惊人的速度传播[27]。同时菌株携带4个具有代表性毒力基因,检测显示,40株产KPC-2的CRKP(40/57,70.2%)携带多个毒力基因,与华中地区三级甲等医院发现59.3%(83株)的CRKP分离株携带rmpA、rmpA2、iroN和iucA 4个毒力基因的研究[28]结果基本一致。目前,此4种毒力基因在产KPC-2 ST11型CRKP菌株中高检出比例存在的机制尚不清楚,需要进一步研究。
本研究通过MLST和荚膜型检测,40株CR-HvKP出现了以ST11-KL64(31/40,77.5%)为主和ST11-KL47(7/40,17.5%)为主的两类克隆群。其中与ST11-KL47型CR-HvKP相比,ST11-KL64型CR-HvKP携带的耐药基因和毒力基因更丰富,表明两种发育不同序列可能已经形成。通过系统发育树分析,ST11-KL47型和ST1-KL64型CRKP流行的科室涉及外科ICU、内科ICU和神经外科等高风险科室,已成为医院传播的主要流行菌株。尤其ST11-KL64型CRKP在无症状CRKP携带者检出极高,是导致患者发生CRKP感染的主要型别[29],应是临床感染防控的重点监测对象。除此之外,本研究检出2株产KPC-2的ST11-KL30型CR-HvKP,而该类型CR-HvKP尚未被报道,表明CR-HvKP进化的多样性。
对5株黏菌素耐药CRKP的分子机制进行研究,发现菌株特异性mgrB基因被破坏性插入ISKpn26元件, 该元件已被证实因其插入mgrB序列是KP黏菌素耐药的普遍机制[30-31]。本研究中5株菌的感染者也存在长时间使用黏菌素的治疗过程,具有ISKpn26插入序列的5株菌被分为3个不同的克隆群(A、F、K),由此推测ISKpn26插入元件可能具有水平和克隆双重遗传传播模式。Zhu等[32]研究指出,IS元件的插入位点以+74位点为优先插入点,与本研究中发现的插入位置为+70位点有所区别,说明IS元件插入mgrB基因的位点具有可变性。
本研究的关键发现是IS元件在ST11-CRKP菌株的mgrB基因失活或突变中的普遍作用。此前有文献[33]报道ISkpn14对mgrB基因的插入失活并证明插入序列起源于含有碳青霉烯酶基因的质粒,特别是blaKPC-2,这也表明IS元件可能在介导耐药中起作用。迫切需要进一步的研究来阐明病原体从多重耐药病原体升级为泛耐药表型的机制[34-35]。当然本研究样本量非常有限,未来需要更大规模或多中心的研究成果来支持上述结果。
综上所述,ST11-KL64 CR-HvKP已成为最普遍的高毒力CRKP,该菌在医院环境中有较高的适应性,可以造成医院感染的传播,感染防控人员需要对此引起足够的重视。同时在黏菌素的压力下,会进一步导致CRKP对黏菌素耐药。由于黏菌素是治疗多重耐药菌感染的最后一道防线,一旦对黏菌素耐药的CR-HvKP在医院环境中大量扩散,将会对临床造成威胁。因此,临床需要采取感染控制措施和使用更严格的政策控制此类病原菌的传播,预防和控制耐黏菌素KP的传播。
利益冲突:所有作者均声明不存在利益冲突。
-
表 1 黏菌素耐药相关基因引物序列及扩增产物大小
Table 1 Primer sequences of colistin resistance-related genes and size of amplified products
基因 序列(5’-3’) 产物大小(bp) 参考文献 基因 序列(5’-3’) 产物大小(bp) 参考文献 mcr-1 AGTCCGTTTGTTCTTGTGGC 320 [23] mcr-8 TTGTCGTCGTGGGCGAAAC 514 [23] AGATCCTTGGTCTCGGCTTG CTGTCGCAAGTTGGGCTAAAG mcr-2 AGCCGAGTCTAAGGACTTGATGAATTTG 576 [23] mcr-9 CGGCGAACTACGCTTACAG 465 [23] GCGGTATCGACATCATAGTCATCTTG CGCACAGTTTCGGGTTATCAC mcr-3 AAATAAAAATTGTTCCGCTTATG 929 [23] phoP ATTGAAGAGGTTGCCGCCCGC 136 [24] AATGGAGATCCCCGTTTTT GCTTGATCGGCTGGTCATTCACC mcr-4 AATTGTCGTGGGAAAAGCCGC 1 062 [23] phoQ ATATGCTGGCGAGATGGGAAAACGG 138 [24] CTGCTGACTGGGCTATTACCGTCAT CCAGCCAGGGAACATCACGCT mcr-5 GTGAAACAGGTGATCGTGACTTACCG 271 [23] pmrA TACGCCGAAAGAGTATGCCC 170 [24] CGTGCTTTACACCGATCATGTGCT GGATCCGCGATTTGCCAATC mcr-6 ACTGACCAAGCCGAGTCTAAG 259 [23] pmrB TGCCAGCTGATAAGCGTCTT 95 [24] GCATCACGGGATTGACATAGC TTCTGGTTGTTGTGCCCTTC mcr-7 GCGACCTCCTACCTGAATG 345 [23] mgrB AAGGCGTTCATTCTACCACC 253 [25] CCCTTTGGCGACGACTTTG TTAAGAAGGCCGTGCTATCC 表 2 CRKP患者的人口和临床统计学特征
Table 2 Demographic and clinical statistic characteristics of CRKP patients
临床特征 例数(n=57) 百分比(%) 年龄≥60岁 32 56.1 性别 男性 41 71.9 女性 16 28.1 标本类型 痰 22 38.6 血 14 24.6 气管抽吸物 6 10.5 腹腔积液/腹腔引流液 6 10.5 支气管肺泡灌洗液 4 7.0 尿 3 5.3 穿刺液 2 3.5 病房 ICU 30 52.6 呼吸内科 12 21.1 感染病科 4 7.0 神经外科 4 7.0 血液内科 3 5.3 肾脏内科 2 3.5 其他病房 2 3.5 基础疾病 呼吸系统疾病 32 56.1 消化系统疾病 8 14.0 神经系统疾病 5 8.8 肿瘤 4 7.0 血液病 3 5.3 心血管疾病 2 3.5 泌尿系统疾病 2 3.5 其他疾病 1 1.8 表 3 57株CRKP药敏试验结果[株(%)]
Table 3 Antimicrobial susceptibility testing results of 57 CRKP strains (No. of isolates [%])
抗菌药物 R I S 哌拉西林他/唑巴坦 45(78.9) 5(8.8) 7(12.3) 头孢他啶 57(100) 0(0) 0(0) 头孢曲松 57(100) 0(0) 0(0) 头孢吡肟 57(100) 0(0) 0(0) 头孢哌酮/舒巴坦 57(100) 0(0) 0(0) 氨曲南 4(7.0) 0(0) 53(93.0) 厄他培南 57(100) 0(0) 0(0) 亚胺培南 57(100) 0(0) 0(0) 美罗培南 57(100) 0(0) 0(0) 妥布霉素 12(21.1) 0(0) 45(78.9) 阿米卡星 9(15.8) 0(0) 48(84.2) 替加环素 0(0) 0(0) 57(100) 环丙沙星 52(91.2) 1(1.8) 4(7.0) 左氧氟沙星 50(87.7) 0(0) 7(12.3) 黏菌素 5(8.8) 0(0) 52(91.2) 注:R为耐药;S为敏感;I为中介。 表 4 5株黏菌素耐药CRKP的MIC值(μg/mL)
Table 4 MIC values of 5 colistin-resistant CRKP strains (μg/mL)
抗菌药物 KP02 KP17 KP20 KP46 KP47 哌拉西林/他唑巴坦 ≥128 ≥128 ≥128 ≥128 ≥128 头孢他啶 ≥64 ≥64 ≥64 ≥64 ≥64 头孢曲松 ≥64 ≥64 ≥64 ≥64 ≥64 头孢吡肟 ≥32 ≥32 ≥32 ≥32 ≥32 头孢哌酮/舒巴坦 ≥64 ≥64 ≥64 ≥64 ≥64 氨曲南 ≥16 ≥16 ≥16 ≥16 ≥16 厄他培南 ≥8 ≥8 ≥8 ≥8 ≥8 亚胺培南 ≥16 ≥16 ≥16 ≥16 ≥16 美洛培南 ≥16 ≥16 ≥16 ≥16 ≥16 妥布霉素 ≤1 ≥16 ≥16 ≥16 ≥16 阿米卡星 ≤2 ≥64 ≥64 ≥64 ≥64 替加环素 1 1 1 1 1 环丙沙星 ≥4 ≥4 ≥4 ≥4 ≥4 左氧氟沙星 ≥8 ≥8 ≥8 ≥8 ≥8 黏菌素 16 32 8 16 16 -
[1] Sands K, Carvalho MJ, Portal E, et al. Characterization of antimicrobial-resistant Gram-negative bacteria that cause neonatal sepsis in seven low- and middle-income countries[J]. Nat Microbiol, 2021, 6(4): 512-523. doi: 10.1038/s41564-021-00870-7 [2] Sequeira RP, McDonald JAK, Marchesi JR, et al. Commensal bacteroidetes protect against Klebsiella pneumoniae colonization and transmission through IL-36 signalling[J]. Nat Microbiol, 2020, 5(2): 304-313. doi: 10.1038/s41564-019-0640-1 [3] Liang XX, Chen P, Deng BG, et al. Outcomes and risk factors of bloodstream infections caused by carbapenem-resistant and non-carbapenem-resistant Klebsiella pneumoniae in China[J]. Infect Drug Resist, 2022, 15: 3161-3171. doi: 10.2147/IDR.S367588 [4] Zhou M, Xiao M, Hou RY, et al. Bundles of care for prevention of ventilator-associated pneumonia caused by carbapenem-resistant Klebsiella pneumoniae in the ICU[J]. Am J Transl Res, 2021, 13(4): 3561-3572. [5] Yang XM, Sun QL, Li JP, et al. Molecular epidemiology of carbapenem-resistant hypervirulent Klebsiella pneumoniae in China[J]. Emerg Microbes Infect, 2022, 11(1): 841-849. doi: 10.1080/22221751.2022.2049458 [6] Hammer CC, Brainard J, Hunter PR. Risk factors and risk factor cascades for communicable disease outbreaks in complex humanitarian emergencies: a qualitative systematic review[J]. BMJ Glob Health, 2018, 3(4): e000647. doi: 10.1136/bmjgh-2017-000647 [7] Kumarasamy KK, Toleman MA, Walsh TR, et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK: a molecular, biological, and epidemiological study[J]. Lancet Infect Dis, 2010, 10(9): 597-602. doi: 10.1016/S1473-3099(10)70143-2 [8] van Duin D, DoiY. The global epidemiology of carbapenemase-producing Enterobacteriaceae[J]. Virulence, 2017, 8(4): 460-469. doi: 10.1080/21505594.2016.1222343 [9] Fu L, Wang SM, Zhang ZK, et al. Whole genome sequence of blaNDM and blaKPC co-producing Klebsiella pneumoniae isolate KSH203 with capsular serotype K25 belonging to ST11 from China[J]. J Glob Antimicrob Resist, 2020, 20: 272-274. doi: 10.1016/j.jgar.2020.01.006 [10] Felipe Vásquez-Ponce, Dantas K, Becerra J, et al. Detecting KPC-2 and NDM-1 coexpression in Klebsiella pneumoniae complex from human and animal hosts in South America[J]. Microbiology Spectrum, 2022, 10(5): 01159-22. [11] Livermore DM, Warner M, Mushtaq S, et al. What remains against carbapenem-resistant Enterobacteriaceae? Evaluation of chloramphenicol, ciprofloxacin, colistin, fosfomycin, minocycline, nitrofurantoin, temocillin and tigecycline[J]. Int J Antimicrob Agents, 2011, 37(5): 415-419. doi: 10.1016/j.ijantimicag.2011.01.012 [12] Cheong HS, Kim SY, Wi YM, et al. Colistin heteroresistance in Klebsiella pneumoniae isolates and diverse mutations of PmrAB and PhoPQ in resistant subpopulations[J]. J Clin Med, 2019, 8(9): 1444. doi: 10.3390/jcm8091444 [13] Wyres KL, Holt KE. Klebsiella pneumoniae as a key traffic-ker of drug resistance genes from environmental to clinically important bacteria[J]. Curr Opin Microbiol, 2018, 45: 131-139. doi: 10.1016/j.mib.2018.04.004 [14] Rossen JWA, Friedrich AW, Moran-Gilad J, et al. Practical issues in implementing whole-genome-sequencing in routine dia- gnostic microbiology[J]. Clin Microbiol Infect, 2018, 24(4): 355-360. doi: 10.1016/j.cmi.2017.11.001 [15] Balloux F, Brønstad Brynildsrud O, van Dorp L, et al. From theory to practice: translating whole-genome sequencing (WGS) into the clinic[J]. Trends Microbiol, 2018, 26(12): 1035-1048. doi: 10.1016/j.tim.2018.08.004 [16] Li G, Shi JM, Zhao Y, et al. Identification of hypervirulent Klebsiella pneumoniae isolates using the string test in combination with Galleria mellonella infectivity[J]. Eur J Clin Microbiol Infect Dis, 2020, 39(9): 1673-1679. doi: 10.1007/s10096-020-03890-z [17] Kelley WS, Stump KH. A rapid procedure for isolation of large quantities of Escherichia coli DNA polymerase I utilizing a lambdapolA transducing phage[J]. J Biol Chem. 1979. 254(9): 3206-3210. doi: 10.1016/S0021-9258(18)50745-6 [18] Lindgreen S. AdapterRemoval: easy cleaning of next-generation sequencing reads[J]. BMC Res Notes, 2012, 5: 337. doi: 10.1186/1756-0500-5-337 [19] Luo RB, Liu BH, Xie YL, et al. SOAPdenovo2: an empirica-lly improved memory-efficient short-read de novo assembler[J]. Gigascience, 2012, 1(1): 18. [20] Bankevich A, Nurk S, Antipov D, et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing[J]. J Comput Biol, 2012, 19(5): 455-477. doi: 10.1089/cmb.2012.0021 [21] Coil D, Jospin G, Darling AE. A5-miseq: an updated pipeline to assemble microbial genomes from Illumina MiSeq data[J]. Bioinformatics, 2015, 31(4): 587-589. doi: 10.1093/bioinformatics/btu661 [22] Letunic I, Bork P. Interactive tree of life v2: online annotation and display of phylogenetic trees made easy[J]. Nucleic Acids Res, 2011, 39(S2): W475-W478. [23] 肖晓, 杭修兵, 王梦, 等. 耐碳青霉烯类肠杆菌目细菌耐药性、临床感染特征及mcr基因分析[J]. 中国感染控制杂志, 2023, 22(1): 31-37. doi: 10.12138/j.issn.1671-9638.20233395 Xiao X, Hang XB, Wang M, et al. Antimicrobial resistance, clinical infection characteristics and mcr genes of carbapenem-resistant Enterobacterales[J]. Chinese Journal of Infection Control, 2023, 22(1): 31-37. doi: 10.12138/j.issn.1671-9638.20233395 [24] Kong YY, Li C, Chen HF, et al. In vivo emergence of colistin resistance in carbapenem-resistant Klebsiella pneumoniae mediated by premature termination of the mgrB gene regulator[J]. Front Microbiol, 2021, 12: 656610. doi: 10.3389/fmicb.2021.656610 [25] Cannatelli A, D'Andrea MM, Giani T, et al. In vivo emergence of colistin resistance in Klebsiella pneumoniae producing KPC-type carbapenemases mediated by insertional inactivation of the PhoQ/PhoP mgrB regulator[J]. Antimicrob Agents Chemother, 2013, 57(11): 5521-5526. doi: 10.1128/AAC.01480-13 [26] Zeng LY, Yang CR, Zhang JS, et al. An outbreak of carba-penem-resistant Klebsiella pneumoniae in an intensive care unit of a major teaching hospital in Chongqing, China[J]. Front Cell Infect Microbiol, 2021, 11: 656070. doi: 10.3389/fcimb.2021.656070 [27] Zhang YW, Jin LY, Ouyang PW, et al. Evolution of hypervirulence in carbapenem-resistant Klebsiella pneumoniae in China: a multicentre, molecular epidemiological analysis[J]. J Antimicrob Chemother, 2020, 75(2): 327-336. doi: 10.1093/jac/dkz446 [28] Xiang YT, Tian HP, Chen QS, et al. Clinical and molecular characteristics of Klebsiella pneumoniae infection in a tertiary general hospital of Wuhan, China[J]. Eur J Clin Microbiol Infect Dis, 2024, 43(2): 269-278. doi: 10.1007/s10096-023-04719-1 [29] Chu WW, Hang XB, Li X, et al. Bloodstream infections in patients with rectal colonization by carbapenem-resistant Enterobacteriaceae: a prospective cohort study[J]. Infect Drug Resist, 2022, 15: 6051-6063. doi: 10.2147/IDR.S383688 [30] Poirel L, Jayol A, Bontron S, et al. The mgrB gene as a key target for acquired resistance to colistin in Klebsiella pneumoniae[J]. J Antimicrob Chemother, 2015, 70(1): 75-80. doi: 10.1093/jac/dku323 [31] Bray AS, Smith RD, Hudson AW, et al. MgrB-dependent colistin resistance in Klebsiella pneumoniae is associated with an increase in host-to-host transmission[J]. mBio, 2022, 13(2): e0359521. doi: 10.1128/mbio.03595-21 [32] Zhu LL, Li P, Zhang GY, et al. Role of the ISKpn element in mediating mgrB gene mutations in ST11 hypervirulent colistin-resistant Klebsiella pneumoniae[J]. Front Microbiol, 2023, 14: 1277320. doi: 10.3389/fmicb.2023.1277320 [33] Li Z, Lei Z, Liu X, et al. Disruption of mgrB gene by ISkpn14 sourced from a blaKPC-2 carrying plasmid mediating polymyxin resistance against carbapenem-resistant Klebsiella pneumoniae during treatment: study on the underlying mechanisms[J]. BMC Microbiol, 2024, 24(1): 422. doi: 10.1186/s12866-024-03572-2 [34] Lv LC, Wan M, Wang CZ, et al. Emergence of a plasmid-encoded resistance-nodulation-division efflux pump conferring resistance to multiple drugs, including tigecycline, in Klebsiella pneumoniae[J]. mBio, 2020, 11(2): e02930-19. [35] Sun SJ, Gao H, Liu YD, et al. Co-existence of a novel plasmid-mediated efflux pump with colistin resistance gene mcr in one plasmid confers transferable multidrug resistance in Klebsiella pneumoniae[J]. Emerg Microbes Infect, 2020, 9(1): 1102-1113. doi: 10.1080/22221751.2020.1768805

 下载:
下载: