Mechanism of horizontal transfer of linezolid resistance gene optrA mediated by plasmids in Enterococcus faecalis
-
摘要:
目的 研究临床上耐利奈唑胺粪肠球菌(LREf)携带的optrA基因通过质粒介导在粪肠球菌间的水平转移机制。 方法 收集昆明市某三级甲等医院2022年8月—2023年8月留取的非重复LREf,使用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)仪进行鉴定,VITEK 2 Compact、纸片扩散法、微量肉汤稀释法进行药敏试验,聚合酶链式反应(PCR)检测optrA基因,全基因组测序(WGS)分析LREf的分子生物学特征,以LREf为供体菌、临床来源粪肠球菌为受体菌进行接合实验。 结果 共收集17株LREf,12株LREf的质粒检测出optrA基因,检测出多种耐药基因及毒力基因。12株LREf菌株以ST16 (50.0%)为主要分型。接合试验的24株接合子中有8株接合成功,接合率为33.3%,进一步分析发现,菌株接合前后位于质粒上的optrA基因上下游均存在IS1216E插入序列,且形成IS1216E-fexA-optrA-erm(A)-IS1216E样的转座单元。 结论 optrA基因可通过同时携带erm(A)、fexA基因的质粒介导在临床粪肠球菌间进行水平转移,而插入序列IS1216E在其水平转移过程中发挥着重要作用。 Abstract:Objective To study the horizontal transfer mechanism of optrA gene-carrying linezolid-resistant Enterococcus faecalis (LREf) among clinical Enterococcus faecalis mediated by plasmids. Methods Non-repeated LREf from a tertiary first-class hospital in Kunming City from August 2022 to August 2023 were collected and identified by matrix-assisted laser desorption/ionisation-time of flight mass spectrometry (MALDI-TOF MS). Antimicrobial susceptibility testing was performed by VITEK 2 Compact, disc diffusion method and microbroth dilution method, optrA gene was detected by polymerase chain reaction (PCR), molecular biology characteristics of LREf was analyzed by whole-genome sequencing (WGS), LREf as the donor strain and clinically isolated Enterococcus faecalis as the recipient strain for conjugation test. Results A total of 17 LREf strains were collected, optrA gene was detected from plasmids of 12 LREf strains. Multiple resistance genes and virulence genes were detected. 12 LREf strains were mainly ST16 type (50.0%). Among the 24 transconjugates in the conjugation test, 8 were successfully conjugated, with a conjugation rate of 33.3%. Further analysis revealed that IS1216E insertion sequences presented at upstream and downstream of the optrA gene on the plasmid before and after conjugation of strains, and formed IS1216E-fexA-optrA-erm (A)-IS1216E-like transposon units. Conclusion The optrA gene can be horizontally transferred among clinical Enterococcus faecalis by mediating of plasmid of genes carrying both erm (A) and fexA, and the insertion sequence IS1216E plays an important role in its horizontal transfer process. -
Keywords:
- Enterococcus faecalis /
- linezolid /
- plasmid /
- optrA /
- whole-genome sequencing /
- horizontal transfer
-
粪肠球菌(Enterococcus faecalis)是一种革兰阳性兼性厌氧球菌,广泛存在于自然环境、人和动物的胃肠道中,被认为是导致尿路感染、心内膜炎及医院感染的常见病原体之一[1]。粪肠球菌不仅对多种抗菌药物具有耐药性,还能通过质粒和转座子的转移获得额外耐药性,导致耐多药粪肠球菌大量出现,甚至形成高危肠球菌复合体(high-risk enterococcal complexes, HiRECC)在医院环境中进一步传播[2]。
利奈唑胺是2000年第一个被美国食品药品监督管理局批准用于临床治疗的人工合成口恶唑烷酮类抗菌药物,其通过结合在细菌50S核糖体亚基23S rRNA成分的V域,从而抑制多种革兰阳性细菌的初始核糖体组装和蛋白质合成来发挥抗菌作用[3]。目前大多数革兰阳性细菌仍然对利奈唑胺敏感,然而由于口恶唑烷酮类药物在人类医学中的频繁使用,粪肠球菌可通过多种机制降低对利奈唑胺的敏感性,导致耐利奈唑胺粪肠球菌(linezolid-resistant Enterococcus faecalis, LREf)的不断出现和散播。根据中国细菌耐药监测网(CHINET)统计2022年1—12月LREf检出率达3.4%[4]。此外,中国细菌耐药监测研究(CARST)2021—2022年革兰阳性菌监测报告显示,粪肠球菌对利奈唑胺的耐药率达7.7%[5]。
目前,肠球菌对利奈唑胺耐药机制主要分为以下两种,第一种为染色体突变导致,包括23S rRNA结合位点的突变(如G2576U、G2447U和G2504A)和编码50S核糖体蛋白的基因突变(如L3、L4、L22的rplC、rplD、rplV);第二种机制为获得可转移性口恶唑烷酮耐药基因, 包括cfr基因家族、optrA、poxtA,这些耐药基因可在质粒、转座子、整合子和插入序列等可移动遗传元件介导下在肠球菌之间和其他革兰阳性菌之间传播[6-7]。optrA基因编码ATP结合盒(ABC)-F蛋白,通过保护细菌核糖体而不受抗菌药物抑制,对利奈唑胺和氟苯尼考具有交叉耐药性[8]。自2015年中国首次在人类和食源性动物分离的粪肠球菌和屎肠球菌中发现optrA基因以来[9],该基因已陆续在人类和食源性动物分离的肠球菌质粒或染色体DNA中检出[10],但主要定位于质粒上且通常位于IS6家族或ISL3家族这类插入序列元件的上游或下游[11]。
本研究以昆明某三级甲等医院2022年8月—2023年8月收集的携带optrA基因的LREf菌株为研究对象,分析LREf菌株的耐药基因、毒力基因、致病蛋白、ST型、遗传环境等分子生物学特征,研究经质粒介导的optrA基因在LREf菌株的水平转移机制,为LREf菌株感染的临床治疗与防控提供科学的理论依据。
1. 资料与方法
1.1 菌株来源
收集2022年8月—2023年8月在昆明某三级甲等医院临床分离的非重复肠球菌17株,通过纸片法将菌株冻存于-80℃冰箱,在37℃的LB肉汤中进行常规培养。这些菌株来源于不同的标本类型,如尿、分泌物、引流液、脓液、胆汁及穿刺液等。利用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF MS)仪进行粪肠球菌鉴定[12]。
1.2 主要仪器与试剂
VITEK 2 Compact及配套的AST-GP67药敏卡(法国梅里埃);MALDI-TOF-MS细菌质谱鉴定仪(德国布鲁克);DENSICHEK电子比浊仪(法国梅里埃);脑心浸液(BHI)琼脂、BHI肉汤;氟苯尼考、夫西地酸抗生素标准品(北京索莱宝);DNA提取试剂盒(北京擎科生物科技有限公司);革兰阳性菌质粒小量提取试剂盒(北京索莱宝)、PCR仪T-100、电泳仪164-5079、ChemiDocXRS+化学发光凝胶成像仪(Bio-Rad公司);聚合酶链式反应(PCR)引物合成及扩增产物测序由生工生物工程(上海)股份有限公司完成。
1.3 药敏试验
对经MALDI-TOF MS鉴定结果为粪肠球菌的菌株进行药敏试验。使用VITEK 2 Compact全自动微生物分析仪(bioMériux,Marcy l’étoile, France)及配套的AST-GP67药敏卡(bioMériux)测定包括利奈唑胺、氨苄西林、四环素、红霉素、呋喃妥因、万古霉素、克林霉素、高浓度庆大霉素、环丙沙星、左氧氟沙星、莫西沙星、高浓度链霉素、替加环素13种抗菌药物的敏感性,药敏结果根据美国临床实验室标准化协会(CLSI)M100-S32指南标准判读。此外,采用纸片扩散法测定粪肠球菌对氯霉素的敏感性,纸片含量为30 μg。为确定粪肠球菌对利奈唑胺的最低抑菌浓度(MIC),使用微量肉汤稀释法复核其利奈唑胺MIC值。根据CLSI指南判读标准,粪肠球菌对利奈唑胺MIC≤2 μg/mL为敏感,MIC≥8 μg/mL为耐药。
1.4 optrA基因检测
采用PCR法检测17株LREf菌株的optrA基因。使用革兰阳性菌质粒小量提取试剂盒提取17株LREf菌株的质粒DNA,使用特异性引物检测optrA基因,检测质粒DNA中optrA基因。引物序列为:F:TTGTCCAAAGCCACCTTTGCAA,R:AACTCTACACCATCCTTATCAAAAAC,产物大小为1 968 bp。扩增条件为:预变性,98℃,2 min;进行35个循环的变性(98℃,10 s)、退火(63℃,10 s)、延伸(72℃,2 min);最后进行终延伸(72℃,5 min)。PCR阳性产物送至北京擎科生物科技公司进行Sanger测序。将测序结果所得序列通过NCBI网站(https://www.ncbi.nlm.nih.gov/)的BLAST与GenBank数据库进行比对。
1.5 LREf菌株全基因组测序(WGS)和生物信息学分析
将optrA基因定位于质粒上的12株LREf菌株使用TSINGKE基因组DNA提取试剂盒提取菌株DNA,送至上海美吉生物公司使用Illumina HiSeq 2500平台进行WGS。应用SPAdes软件对数据进行测序结果拼接,RAST网站进行拼接后的注释[13]。根据WGS结果,使用基因组流行病学中心(https://cge.cbs.dtu.dk)的在线工具ResFinder 4.4(https://cge.food.dtu.dk/services/ResFinder/)、LRE-Finder[14]、VirulenceFinder 2.0[15]、MLST 2.0[16]、PathogenFinder(https://cge.food.dtu.dk/services/PathogenFinder/)对菌株携带的耐药基因、毒力基因及23S rRNA的突变情况、ST类型进行预测。应用建树软件megaX[17],比对方法clustalW,建树方法NJ,设定bootstrap为1 000绘制系统发育树。
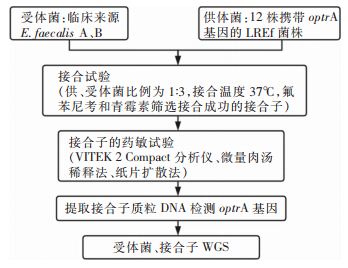
1.6 接合试验及分析
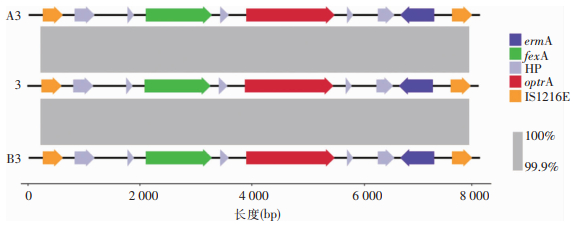
以12株携带optrA基因的LREf菌株作为供体菌,临床来源E.faecalis A、B作为受体菌进行接合试验,并通过药敏试验、optrA基因检测、WGS对接合子进一步分析。具体的接合过程可见流程图 1。使用在线网站CGview(https://cgview.ca/)绘制3号供体菌质粒基因组结构圈图。选取接合成功的3号供体菌及其相应的接合子A3、B3进行基因组结构共线性分析,最后通过easyfig进行绘图,应用矢量图形制作软件(Adobe illustrator)对图形进行美化。
1.7 统计学分析
应用SPSS 26.0进行数据分析。计数资料采用例(百分比)表示。
2. 结果
2.1 细菌的分离鉴定和药物敏感性
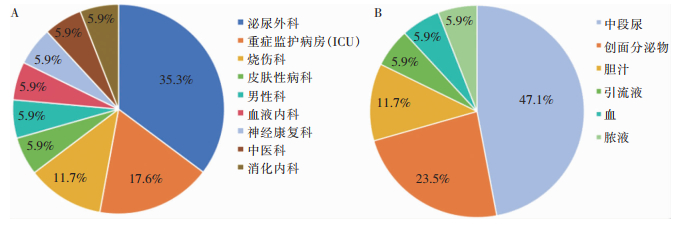
本研究共收集2022年8月—2023年8月来自昆明某三级甲等医院经MALDI-TOF MS鉴定和微量肉汤稀释法测定利奈唑胺MIC≥8 μg/mL的粪肠球菌17株。17株LREf对14种抗菌药物的耐药率分别为:利奈唑胺、红霉素、四环素、氯霉素、克林霉素均为100%(17株),环丙沙星为58.8%(10株),高浓度庆大霉素为52.9%(9株),左氧氟沙星为58.8%(10株),高浓度链霉素为52.9%(9株),其中对利奈唑胺的MIC值为8 μg/mL或16 μg/mL,未发现高水平耐药现象。所有粪肠球菌对3种及以上抗菌药物耐药,均表现为多重耐药。17株LREf菌株的科室分布和标本来源见图 2。
2.2 optrA基因检测结果
共有12株(70.6%)LREf的质粒DNA检测出optrA基因。将optrA基因PCR阳性产物的测序结果在NCBI数据库中进行BLAST比对,发现其与接合质粒pE349携带的optrA基因序列(GenBank accession no.KP399637)具有较高相似性,符合度为99%。17株LREf菌株optrA基因定位电泳图见图 3。
2.3 WGS结果和生物信息学分析
2.3.1 耐药基因型、毒力基因、致病蛋白
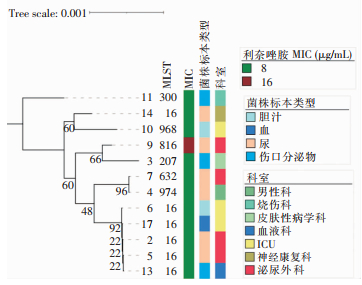
12株LREf菌株携带耐药基因15种,其中,optrA、tet(M)、fexA、lsa(A)携带率最高(100%),其次是erm(A)、dfrG,携带率分别为91.7%、83.3%,未检测到cfr、cfr(B)、poxtA基因。共检测细菌黏附素毒力基因(ace、agg、elrA)、蛋白酶基因(srtA)、生物膜形成、吸附酶组装菌毛基因(ebpB、ebpA、ebpC)等在内的20种毒力基因,其中有10个毒力基因(elrA、srtA、ace、efaAfs、ccf、cob、cad、camE、ebpA和tpx)在所有的LREf中均可检测到。此外,在这些菌株中共检测到包括性信息素接合蛋白、Tn5251转录蛋白、Tn916样转座蛋白、铁化合物ABC转运蛋白在内的29种致病蛋白。所有的LREf菌株中均包含铁氧化还原蛋白与性信息素接合蛋白,其次较多的为Tn916样转座蛋白与PTS系统蛋白。见表 1。
表 1 12株LREf的菌株特征Table 1 Characteristics of 12 LREf strains菌株编号 科室来源 标本类型 利奈唑胺MIC
(μg/mL)耐药基因 毒力基因 2 泌尿外科 中段尿 8 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(L)、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM 3 男性科 中段尿 8 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、erm(B)、lsa(A)、lsa(E)、fexA、tet(M)、dfrG、ant(6’)-la、lnu(B)、tet(L) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylB、fsrB、gelE 4 皮肤性病科 创面分泌物 8 optrA、lsa(A)、ermA、cat、fexA、tet(L)、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylB、fsrB、gelE、hylA、cylA、cylL、cylM、cad 5 泌尿外科 中段尿 8 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM、cad 6 ICU 胆汁 8 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM 7 泌尿外科 中段尿 8 optrA、ant(6’)-la、erm(A)、erm(B)、fexA、tet(M)、dfrG、lsa(A) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylB、fsrB、gelE 9 泌尿外科 中段尿 16 optrA、cat、fexA、tet(M)、lsa(A)、tet(L) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpB、tpx、efaAfs、hylB、hylA、fsrB、gelE、cylA、cylL、cylM 10 ICU 胆汁 8 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、ant(6’)-la、erm(A)、lsa(A)、lsa(E)、cat、fexA、tet(M)、dfrG、lnu(B) elrA、srtA、ace、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA 11 烧伤科 创面分泌物 8 optrA、ant(6’)-la、erm(A)、erm(B)、lsa(A)、fexA、tet(M) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA、hylB、fsrB、gelE 13 血液内科 创面分泌物 8 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、strA、erm(A)、erm(B)、lsa(A)、lsa(E)、fexA、cat、tet(M)、dfrG、Inu(B) elrA、srtA、ace、ccf、cob、cad、camE、cylA、cylL、cylM、ebpA、ebpC、tpx、efaAfs、hylA 14 神经康复科 中段尿 16 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、lsa(E)、lnu(B)、cat、fexA、tet(M)、dfrG、erm(B)、tet(L) elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM、cad、fsrB、gelE 17 ICU 血 8 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、lsa(E)、lnu(B)、fexA、tet(M)、dfrG、erm(B) elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM、cad 2.3.2 ST型分析和系统发育树结果
应用MLST 2.0对菌株的全基因组结果进行分析,12株LREf菌株可分为7种ST型,其中ST16为主要分型(占50.0%)。MEGA X软件构建的12株LREf进行系统发育进化树见图 4。
2.4 接合试验
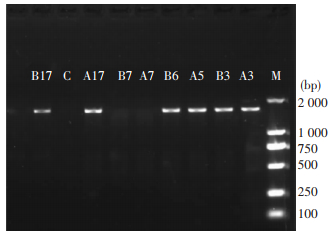
24株接合子中有8株接合子携带optrA基因,ST型与受体菌A、B的ST型一致,均为ST4,接合成功率为30.0%。其中与受体A接合成功的有4株,分别为3、5、7、17(对应接合子命名为A3、A5、A7、A17);与受体菌B接合成功的有4株,分别为3、6、7、17(对应接合子命名为B3、B6、B7、B17)。8株接合子对利奈唑胺、青霉素、氯霉素、红霉素、四环素表现为耐药,对呋喃妥因、替加环素、万古霉素表现为敏感。其中对利奈唑胺的MIC值均为8 μg/mL,较受体菌A、B有7倍的提高(从1 μg/mL升高至8 μg/mL)。对8株接合子的耐药基因分析发现,除含有optrA基因外,接合子上还含有erm(A)、erm(B)、dfrG、lsa(A)、ant(6’)-la、tet(M)和fexA。此外,在这8株接合子的质粒DNA中有6株接合子检测出optrA基因,optrA基因检测结果见图 5。供体菌、受体菌、接合子的耐药表型、基因型、ST分型见表 2。
表 2 供体菌、受体菌、接合子的耐药表型、基因型、ST分型Table 2 Resistance phenotypes, genotypes, and ST typing of donor strains, recipient strains, and transconjugants菌株编号 MIC(μg/mL) ST型 耐药基因 optrA位置 LZD PEN AMP CHL# CIP ERY LVX TCY TGC/VA/NIT
A1 32 2 23 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 ant(6’)-la、lsa(A)、erm(B)、tet(M) - B 1 32 4 21 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 ant(6’)-la、lsa(A)、erm(B)、tet(M) - 3 8 2 2 10 ≤1 ≥8 1 ≥16 ≤0.12/≤0.5/≤16 ST207 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、erm(B)、lsa(A)、lsa(E)、fexA、tet(M)、dfrG、ant(6’)-la、Inu(B)、tet(L) 质粒 A3 8 32 4 6 ≤1 ≥8 2 ≥16 ≤0.12/2/≤16 ST4 optrA、aph(6’)-(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 B3 8 32 4 7 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、erm(B)、lsa(A)、fexA、tet(M)、lsa(E)、Inu(B)、dfrG 质粒 5 8 4 2 11 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST16 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 A5 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 6 8 4 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST16 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 B6 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、fexA、tet(M)、lsa(E)、lnu(B)、dfrG、erm(B) 质粒 7 8 1 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST632 optrA、ant(6’)-la、erm(A)、erm(B)、fexA、tet(M)、dfrG、lsa(A) 质粒 A7 8 32 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/32 ST4 optrA、ant(6’)-la、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG 染色体 B7 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/32 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、fexA、tet(M)、lsa(E)、lnu(B)、dfrG、erm(B) 染色体 17 8 2 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST16 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、lsa(E)、lnu(B)、fexA、tet(M)、dfrG、erm(B) 质粒 A17 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(2’)-Ia、erm(A)、lsa(A)、fexA、tet(M)、erm(B)、dfrG 质粒 B17 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、fexA、tet(M)、lsa(E)、lnu(B)、dfrG、erm(B) 质粒 注:A、B分别为两株受体菌;LZD为利奈唑胺(≥8 μg/mL为耐药,≤2 μg/mL为敏感),PEN为青霉素(≥16 μg/mL为耐药,≤8 μg/mL为敏感),AMP为氨苄西林(≥16 μg/mL为耐药,≤8 μg/mL为敏感),CIP为环丙沙星(≥4 μg/mL为耐药,≤1 μg/mL为敏感),ERY为红霉素(≥8 μg/mL为耐药,≤0.5 μg/mL为敏感),LVX为左氧氟沙星(≥8 μg/mL为耐药,≤2 μg/mL为敏感),NIT为呋喃妥因(≥128 μg/mL为耐药,≤32 μg/mL为敏感),TCY为四环素(≥16 μg/mL为耐药,≤4 μg/mL为敏感),TGC为替加环素(≥16 μg/mL为耐药,≤4 μg/mL为敏感),VA为万古霉素(≥32 μg/mL为耐药,≤4 μg/mL为敏感);CHL为氯霉素(抑菌圈直径≤12 mm为耐药,≥18 mm为敏感);#表示药敏方法采用纸片扩散法;-表示菌株不携带optrA。 2.5 接合子质粒基因组分析
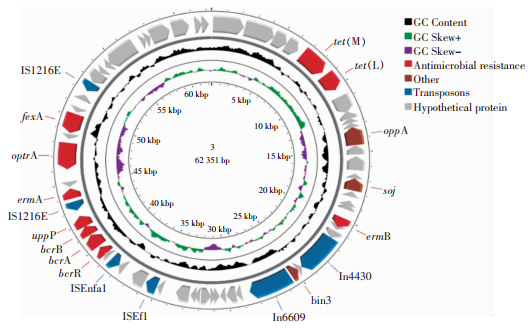
为进一步分析质粒介导的optrA基因在粪肠球菌之间的水平转移机制,选取3号供体菌与其相应的接合子A3、B3进行质粒基因组分析。3号供体菌质粒的基因组结构见图 6,其长度为62 351 bp,其GC含量为34.9%,含有70个编码区(coding sequences),该质粒除含有optrA基因外,还包括fexA、tet(L)、erm(A)、erm(B)等耐药基因和杆菌肽耐药操纵子bcrA、bcrB和bcrR。在optrA基因两侧存在有两个大小和方向相同的插入元件IS1216E,形成IS1216E-fexA-optrA-erm(A)-IS1216E样的转座单元。此外还存在一些可移动遗传元件,如插入序列ISEnfa1、ISEf1和转座子Tn6009、Tn4430,可能共同参与促进optrA基因的水平传播。
将3号供体菌与对应的A3、B3接合子进行基因组比较分析,表明3号供体菌与接合子A3、B3中均含有optrA、erm(A)、fexA耐药基因,且fexA基因位于optrA基因的上游,与optrA基因同向;erm(A)基因位于optrA基因下游,与optrA基因方向相反,形成了IS1216E-fexA-optrA-erm(A)-IS1216E样的转座单元。以上结果说明插入序列IS1216E在介导粪肠球菌质粒上optrA基因的水平转移过程中发挥着重要作用。见图 7。
3. 讨论
肠球菌属不仅对包括头孢菌素和磺胺类药物在内的多种抗菌药物具有天然耐药性,还能通过质粒和转座子的转移获得额外的耐药性,导致在医院环境中进一步传播,被认为是医院感染控制的重大挑战。粪肠球菌可通过水平基因转移(horizontal gene transfer, HGT)获得耐药基因,进而获得相应抗菌药物耐药性[18]。HGT通常由可移动遗传元件(mobile genetic element, MGE)如质粒、转座子、噬菌体、整合子和插入序列等介导,其能促进粪肠球菌耐药基因在种甚至不同种属间发生水平传播[19]。
与之前的研究不同,本研究接合试验中使用供体菌、受体菌均为临床来源的粪肠球菌,以分析optrA基因在临床粪肠球菌间的水平转移机制。结果显示,接合的24株中有8株接合成功,接合率为33.3%。进一步研究发现8株接合子有6株的optrA基因在接合后同样位于粪肠球菌质粒上,表明利奈唑胺耐药基因optrA经质粒介导在粪肠球菌水平传播,验证了质粒接合是细菌转移耐药基因的重要方式之一。但从总体上看,接合效率较低,原因可能包括以下几个方面。首先optrA基因可能位于非接合质粒上。基于质粒接合完整性以及转移能力的差异,可将质粒分为接合性质粒、可移动质粒和不可移动质粒[20]。其中仅接合性质粒可以自主和独立地进行接合转移,位于非接合质粒上便不具备转移能力。其次,由于本研究中所选择的受体菌为临床菌株,与工程菌JH2-2相比,临床菌株包含许多质粒,这些质粒在接合过程中可能会因质粒的不相容性导致质粒间发生竞争抑制,从而未能使携带optrA基因的质粒与其成功接合。最后,LREf菌株大多数表现为多重耐药,质粒携带其他耐药基因的同时会对宿主菌的生长代谢及其环境适应性造成一定影响而不利于携带optrA基因质粒的菌株进行接合转移[21]。
为了进一步研究利奈唑胺耐药基因optrA经质粒介导在粪肠球菌水平传播的机制,对3号供体菌与其相应的接合子A3、B3进行质粒基因组结构分析。研究发现,在接合前后optrA基因上下游均存在着两个方向相同、大小相同的IS1216E插入序列,可与fexA、erm(A)形成IS1216E-fexA-optrA-erm(A)-IS1216E转座单元。首先,fexA、erm(A)和optrA基因共定位表明,LREf的出现可能是由于使用非口恶唑烷酮类(大环内酯类、氟苯尼考)等抗生素选择压力导致。几乎所有质粒optrA基因的上游都存在fexA。氟苯尼考作为一种兽用抗生素,在治疗家畜感染时广泛、反复的使用对环境细菌施加了选择压力,可能与利奈唑胺耐药基因的产生呈正相关[22]。其次,接合前后所形成的IS1216E-fexA-optrA-erm(A)-IS1216E转座单元有助于optrA基因的转移。两个IS1216E可以组合形成一个环状中间体,使optrA基因整合到质粒中,在optrA基因的水平转移中起到重要作用。IS1216E是IS1216的一种亚型,属IS6家族,其广泛存在于多重耐药革兰阳性菌中,是位于optrA基因侧翼最常见的插入序列。无论有无额外基因,optrA基因通常被两个相同方向和大小的IS1216E拷贝括起来。当这些IS1216E拷贝重组时,产生一个易转位单位(translocatable units, TU),TU可以整合到质粒、整合结合元件(integrative and conjuative element, ICE)或不同的染色体位点上,如果整合到接合质粒或ICE中,可能导致optrA基因在相同或不同菌种之间传播[23]。IS6家族除了能促进位于质粒的optrA基因水平转移外,也可能促进临床上其他重要的抗生素耐药基因交换,包括但不限于optrA基因[24-25]。因此,定期监测包括IS6家族在内的IS介导的抗菌药物耐药基因的动态移动性对控制抗菌药物耐药性在菌株间的传播至关重要。
本研究对昆明某院检出的携带optrA基因LREf菌株的分子特征及传播机制进行分析。携带optrA基因的LREf菌株对多种抗菌药物具有耐药性,携带多种耐药基因和毒力基因。ST16是该院携带optrA基因LREf菌株中较为流行的ST型。质粒接合是optrA基因在LREf菌株中水平转移的重要方式,其中质粒上的插入序列IS1216E在optrA基因的水平转移中起着重要作用。多重耐药粪肠球菌的出现,给临床抗感染治疗带来了极大的挑战,对公众健康构成潜在威胁。因此,在临床使用药物时应根据实际情况合理用药,对肠球菌携带的optrA基因进行持续监测并制定有效的防控措施。本项研究也存在一些局限性:首先,由于粪肠球菌中利奈唑胺耐药性仍然很少见,导致纳入研究的LREf菌株数量相对较少。其次,本研究为一项单中心研究,需要进一步的多中心研究分析含有optrA基因LREf菌株的流行病学特征。
利益冲突:所有作者均声明不存在利益冲突。
-
表 1 12株LREf的菌株特征
Table 1 Characteristics of 12 LREf strains
菌株编号 科室来源 标本类型 利奈唑胺MIC
(μg/mL)耐药基因 毒力基因 2 泌尿外科 中段尿 8 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(L)、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM 3 男性科 中段尿 8 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、erm(B)、lsa(A)、lsa(E)、fexA、tet(M)、dfrG、ant(6’)-la、lnu(B)、tet(L) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylB、fsrB、gelE 4 皮肤性病科 创面分泌物 8 optrA、lsa(A)、ermA、cat、fexA、tet(L)、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylB、fsrB、gelE、hylA、cylA、cylL、cylM、cad 5 泌尿外科 中段尿 8 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM、cad 6 ICU 胆汁 8 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(M)、dfrG elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM 7 泌尿外科 中段尿 8 optrA、ant(6’)-la、erm(A)、erm(B)、fexA、tet(M)、dfrG、lsa(A) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylB、fsrB、gelE 9 泌尿外科 中段尿 16 optrA、cat、fexA、tet(M)、lsa(A)、tet(L) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpB、tpx、efaAfs、hylB、hylA、fsrB、gelE、cylA、cylL、cylM 10 ICU 胆汁 8 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、ant(6’)-la、erm(A)、lsa(A)、lsa(E)、cat、fexA、tet(M)、dfrG、lnu(B) elrA、srtA、ace、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA 11 烧伤科 创面分泌物 8 optrA、ant(6’)-la、erm(A)、erm(B)、lsa(A)、fexA、tet(M) elrA、srtA、ace、agg、ccf、cob、cad、camE、ebpA、ebpC、tpx、efaAfs、hylA、hylB、fsrB、gelE 13 血液内科 创面分泌物 8 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、strA、erm(A)、erm(B)、lsa(A)、lsa(E)、fexA、cat、tet(M)、dfrG、Inu(B) elrA、srtA、ace、ccf、cob、cad、camE、cylA、cylL、cylM、ebpA、ebpC、tpx、efaAfs、hylA 14 神经康复科 中段尿 16 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、lsa(E)、lnu(B)、cat、fexA、tet(M)、dfrG、erm(B)、tet(L) elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM、cad、fsrB、gelE 17 ICU 血 8 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、lsa(E)、lnu(B)、fexA、tet(M)、dfrG、erm(B) elrA、srtA、ace、agg、ccf、cob、camE、ebpA、ebpC、tpx、efaAfs、hylA、cylA、cylL、cylM、cad 表 2 供体菌、受体菌、接合子的耐药表型、基因型、ST分型
Table 2 Resistance phenotypes, genotypes, and ST typing of donor strains, recipient strains, and transconjugants
菌株编号 MIC(μg/mL) ST型 耐药基因 optrA位置 LZD PEN AMP CHL# CIP ERY LVX TCY TGC/VA/NIT
A1 32 2 23 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 ant(6’)-la、lsa(A)、erm(B)、tet(M) - B 1 32 4 21 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 ant(6’)-la、lsa(A)、erm(B)、tet(M) - 3 8 2 2 10 ≤1 ≥8 1 ≥16 ≤0.12/≤0.5/≤16 ST207 optrA、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、erm(B)、lsa(A)、lsa(E)、fexA、tet(M)、dfrG、ant(6’)-la、Inu(B)、tet(L) 质粒 A3 8 32 4 6 ≤1 ≥8 2 ≥16 ≤0.12/2/≤16 ST4 optrA、aph(6’)-(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 B3 8 32 4 7 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、erm(B)、lsa(A)、fexA、tet(M)、lsa(E)、Inu(B)、dfrG 质粒 5 8 4 2 11 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST16 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 A5 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 6 8 4 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST16 optrA、aac(6’)-aph(2”)、ant(6’)-la、aph(3’)-Ⅲ、lsa(A)、lsa(E)、lnu(B)、erm(A)、erm(B)、fexA、tet(M)、dfrG 质粒 B6 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、fexA、tet(M)、lsa(E)、lnu(B)、dfrG、erm(B) 质粒 7 8 1 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST632 optrA、ant(6’)-la、erm(A)、erm(B)、fexA、tet(M)、dfrG、lsa(A) 质粒 A7 8 32 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/32 ST4 optrA、ant(6’)-la、lsa(A)、erm(A)、erm(B)、fexA、tet(M)、dfrG 染色体 B7 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/32 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、fexA、tet(M)、lsa(E)、lnu(B)、dfrG、erm(B) 染色体 17 8 2 2 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/1/≤16 ST16 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、lsa(E)、lnu(B)、fexA、tet(M)、dfrG、erm(B) 质粒 A17 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(2’)-Ia、erm(A)、lsa(A)、fexA、tet(M)、erm(B)、dfrG 质粒 B17 8 32 4 6 ≥4 ≥8 ≥8 ≥16 ≤0.12/2/≤16 ST4 optrA、ant(6’)-la、aac(6’)-aph(2”)、aph(3’)-Ⅲ、erm(A)、lsa(A)、fexA、tet(M)、lsa(E)、lnu(B)、dfrG、erm(B) 质粒 注:A、B分别为两株受体菌;LZD为利奈唑胺(≥8 μg/mL为耐药,≤2 μg/mL为敏感),PEN为青霉素(≥16 μg/mL为耐药,≤8 μg/mL为敏感),AMP为氨苄西林(≥16 μg/mL为耐药,≤8 μg/mL为敏感),CIP为环丙沙星(≥4 μg/mL为耐药,≤1 μg/mL为敏感),ERY为红霉素(≥8 μg/mL为耐药,≤0.5 μg/mL为敏感),LVX为左氧氟沙星(≥8 μg/mL为耐药,≤2 μg/mL为敏感),NIT为呋喃妥因(≥128 μg/mL为耐药,≤32 μg/mL为敏感),TCY为四环素(≥16 μg/mL为耐药,≤4 μg/mL为敏感),TGC为替加环素(≥16 μg/mL为耐药,≤4 μg/mL为敏感),VA为万古霉素(≥32 μg/mL为耐药,≤4 μg/mL为敏感);CHL为氯霉素(抑菌圈直径≤12 mm为耐药,≥18 mm为敏感);#表示药敏方法采用纸片扩散法;-表示菌株不携带optrA。 -
[1] Carvalhaes CG, Sader HS, Flamm RK, et al. Assessment of tedizolid in vitro activity and resistance mechanisms against a collection of Enterococcus spp. causing invasive infections, including isolates requiring an optimized dosing strategy for daptomycin from U.S. and European medical centers, 2016 to 2018[J]. Antimicrob Agents Chemother, 2020, 64(4): e00175-20. [2] El-Zamkan MA, Mohamed HMA. Antimicrobial resistance, virulence genes and biofilm formation in Enterococcus species isolated from milk of sheep and goat with subclinical mastitis[J]. PLoS One, 2021, 16(11): e0259584. doi: 10.1371/journal.pone.0259584 [3] Egan SA, Shore AC, O'Connell B, et al. Linezolid resistance in Enterococcus faecium and Enterococcus faecalis from hospitalized patients in Ireland: high prevalence of the MDR genes optrA and poxtA in isolates with diverse genetic backgrounds[J]. J Antimicrob Chemother, 2020, 75(7): 1704-1711. doi: 10.1093/jac/dkaa075 [4] 胡付品, 郭燕, 朱德妹, 等. 2021年CHINET中国细菌耐药监测[J]. 中国感染与化疗杂志, 2022, 22(5): 521-530. Hu FP, Guo Y, Zhu DM, et al. CHINET surveillance of antimicrobial resistance among the bacterial isolates in 2021[J]. Chinese Journal of Infection and Chemotherapy, 2022, 22(5): 521-530. [5] 李耘, 郑波, 薛峰, 等. 中国细菌耐药监测研究(CARST)2021—2022年革兰氏阳性菌监测报告[J]. 中国临床药理学杂志, 2023, 39(23): 3509-3524. Li Y, Zheng B, Xue F, et al. Antimicrobial susceptibility of Gram-positive organisms: results from China antimicrobial resistance surveillance trial (CARST) program, 2021-2022[J]. The Chinese Journal of Clinical Pharmacology, 2023, 39(23): 3509-3524. [6] Hu YX, Won D, Nguyen LP, et al. Prevalence and genetic analysis of resistance mechanisms of linezolid-nonsusceptible Enterococci in a tertiary care hospital examined via whole-genome sequencing[J]. Antibiotics (Basel), 2022, 11(11): 1624. doi: 10.3390/antibiotics11111624 [7] Lei CW, Chen X, Liu SY, et al. Clonal spread and horizontal transfer mediate dissemination of phenicol-oxazolidinone-tetracycline resistance gene poxtA in Enterococci isolates from a swine farm in China[J]. Vet Microbiol, 2021, 262: 109219. doi: 10.1016/j.vetmic.2021.109219 [8] Sharkey LKR, Edwards TA, O'Neill AJ. ABC-F proteins mediate antibiotic resistance through ribosomal protection[J]. mBio, 2016, 7(2): e01975. [9] Wang Y, Lv Y, Cai JC, et al. A novel gene, optrA, that confers transferable resistance to oxazolidinones and phenicols and its presence in Enterococcus faecalis and Enterococcus faecium of human and animal origin[J]. J Antimicrob Chemother, 2015, 70(8): 2182-2190. doi: 10.1093/jac/dkv116 [10] Sadowy E. Linezolid resistance genes and genetic elements enhancing their dissemination in Enterococci and Streptococci[J]. Plasmid, 2018, 99: 89-98. doi: 10.1016/j.plasmid.2018.09.011 [11] Freitas AR, Tedim AP, Novais C, et al. Comparative geno-mics of global optrA-carrying Enterococcus faecalis uncovers a common chromosomal hotspot for optrA acquisition within a diversity of core and accessory genomes[J]. Microb Genom, 2020, 6(6): e000350. [12] Kornienko M, Ilina E, Lubasovskaya L, et al. Analysis of nosocomial Staphylococcus haemolyticus by MLST and MALDI-TOF mass spectrometry[J]. Infect Genet Evol, 2016, 39: 99-105. doi: 10.1016/j.meegid.2015.12.015 [13] Aziz RK, Bartels D, Best AA, et al. The RAST server: rapid annotations using subsystems technology[J]. BMC Genomics, 2008, 9: 75. doi: 10.1186/1471-2164-9-75 [14] Hasman H, Clausen PTLC, Kaya H, et al. LRE-finder, a web tool for detection of the 23S rRNA mutations and the optrA, cfr, cfr(B) and poxtA genes encoding linezolid resis-tance in Enterococci from whole-genome sequences[J]. J Antimicrob Chemother, 2019, 74(6): 1473-1476. doi: 10.1093/jac/dkz092 [15] Joensen KG, Scheutz F, Lund O, et al. Real-time whole-genome sequencing for routine typing, surveillance, and outbreak detection of verotoxigenic Escherichia coli[J]. J Clin Microbiol, 2014, 52(5): 1501-1510. doi: 10.1128/JCM.03617-13 [16] Larsen MV, Cosentino S, Rasmussen S, et al. Multilocus sequence typing of total-genome-sequenced bacteria[J]. J Clin Microbiol, 2012, 50(4): 1355-1361. doi: 10.1128/JCM.06094-11 [17] Kumar S, Stecher G, Li M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol, 2018, 35(6): 1547-1549. doi: 10.1093/molbev/msy096 [18] Hegstad K, Mikalsen T, Coque TM, et al. Mobile genetic elements and their contribution to the emergence of antimicrobial resistant Enterococcus faecalis and Enterococcus faecium[J]. Clin Microbiol Infect, 2010, 16(6): 541-554. doi: 10.1111/j.1469-0691.2010.03226.x [19] Schwarz S, Zhang WJ, Du XD, et al. Mobile oxazolidinone resistance genes in Gram-positive and Gram-negative bacteria[J]. Clin Microbiol Rev, 2021, 34(3): e0018820. doi: 10.1128/CMR.00188-20 [20] Smillie C, Garcillán-Barcia MP, Francia MV, et al. Mobility of plasmids[J]. Microbiol Mol Biol Rev, 2010, 74(3): 434-452. doi: 10.1128/MMBR.00020-10 [21] Young S, Rohr JR, Harwood VJ. Vancomycin resistance plasmids affect persistence of Enterococcus faecium in water[J]. Water Res, 2019, 166: 115069. doi: 10.1016/j.watres.2019.115069 [22] Wang YY, Li XW, Fu YL, et al. Association of florfenicol residues with the abundance of oxazolidinone resistance genes in livestock manures[J]. J Hazard Mater, 2020, 399: 123059. doi: 10.1016/j.jhazmat.2020.123059 [23] Shang YH, Li DX, Shan XX, et al. Analysis of two pheromone-responsive conjugative multiresistance plasmids carrying the novel mobile optrA locus from Enterococcus faecalis[J]. Infect Drug Resist, 2019, 12: 2355-2362. doi: 10.2147/IDR.S206295 [24] Liu SH, Yang XX, Li RC, et al. IS6 family insertion sequences promote optrA dissemination between plasmids va-rying in transfer abilities[J]. Appl Microbiol Biotechnol, 2024, 108(1): 132. doi: 10.1007/s00253-023-12858-w [25] Che Y, Yang Y, Xu XQ, et al. Conjugative plasmids interact with insertion sequences to shape the horizontal transfer of antimicrobial resistance genes[J]. Proc Natl Acad Sci U S A, 2021, 118(6): e2008731118. doi: 10.1073/pnas.2008731118

 下载:
下载: