Drug resistance and genomic characteristics of clinically isolated hypermucoviscous Klebsiella pneumoniae
-
摘要:
目的 分析临床分离高黏液肺炎克雷伯菌(hmKp)的耐药情况及基因组特征。 方法 收集2019—2023年分离自淮安市国家致病菌识别网哨点医院临床标本的肺炎克雷伯菌,使用VITEK 2 Compact对菌株进行鉴定,拉丝试验判断黏液表型,微量肉汤稀释法测定hmKp的耐药性。应用全基因组测序技术进行分子分型,并对菌株携带的毒力和耐药基因进行注释。 结果 共收集hmKp 60株,主要分离自痰标本(33.33%)和血标本(28.33%)。60株菌株基因组平均大小为5.6 Mb,平均GC含量为57.09%。多位点序列分型(ST)显示,有28种ST型,优势ST型为ST11(11.67%)、ST15(11.67%)和ST412(10.00%)。核心基因组多位点序列分型(cgMLST)分析发现,部分菌株高度同源,未发现暴发菌株。多重耐药菌株占60.00%,耐亚胺培南菌株占28.33%。共携带64种耐药基因,其中84.38%的耐药基因位于可移动原件上,磷霉素相关耐药基因、超广谱β-内酰胺类抗生素相关耐药基因在菌株中携带率较高,分别为100%、98.33%。碳青霉烯类耐药基因blaKPC-2,21.67%由ST11、ST1和ST15菌株携带;blaNDM-5基因3.33%由ST76菌株携带。共携带101种毒力基因,其中Colibactin和Ⅵ型分泌系统相关毒力基因种类最多。所有菌株均携带荚膜合成调节相关基因(rcsA、rcsB)、外排泵相关基因(acrA、acrB)和肠杆菌素相关基因(entABCEF、fepABCDFG、fes)。 结论 淮安地区临床分离的hmKp呈现多重耐药,优势ST型为ST11、ST15和ST412,携带多种能够进行水平转移的耐药基因和毒力基因,应加强防控措施。 Abstract:Objective To analyze the drug resistance and genomic characteristics of clinically isolated hypermucoviscous Klebsiella pneumoniae (hmKp). Methods Klebsiella pneumoniae isolated from clinical specimens from the sentinel hospitals of National Pathogen Identification Network in Huai'an City from 2019 to 2023 were collected, strains were identified by VITEK 2 Compact, mucus phenotype was determined by string test, resistance of hmKp was determined by microbroth dilution method. Molecular typing was conducted by whole-genome sequencing technology, annotation of virulence and resistance genes carried by strains was performed. Results A total of 60 strains of hmKp were collected, mainly isolated from sputum specimens (33.33%) and blood specimens (28.33%). The average genome size of 60 strains was 5.6 Mb, with an average GC content of 57.09%. Multilocus sequence typing (MLST) showed that there were 28 ST types, with the dominant ST types being ST11 (11.67%), ST15 (11.67%), and ST412 (10.00%). Core genome MLST (cgMLST) analysis revealed that some strains were highly homologous, and no outbreak strains were found. Multidrug-resistant strains accounted for 60.00%, while imipe-nem-resistant strains accounted for 28.33%. 64 types of resistance genes were carried, 84.38% of which were located on mobile element. The carriage rates of fosfomycin-related resistance genes and extended-spectrum β-lactam antibiotic-related resistance genes were relatively high in the strains, at 100% and 98.33%, respectively. Among carbapenem-resistance gene blaKPC-2, 21.67% was carried by ST11, ST1, and ST15 strains, among blaNDM-5 gene, 3.33% was carried by ST76 strain. A total of 101 virulence genes were carried, most were Colibactin and type Ⅵ secretion system-related virulence genes. All strains carried capsule synthesis regulation-related genes (rcsA, rcsB), efflux pump-related genes (acrA, acrB), and enterobacterin-related genes (entABCEF, fepABCDFG, fes). Conclusion Clinically isolated hmKp in Huai'an exhibits multidrug resistance, with dominant ST types ST11, ST15, and ST412, carrying multiple horizontally transferable resistance and virulence genes, prevention and control measures should be strengthened. -
肺炎克雷伯菌(Klebsiella pneumoniae, Kp)在我国临床分离率很高,可引起呼吸道感染、尿路感染、肝脓肿、血流感染、脑膜炎、胸腔感染、腹腔感染、皮肤伤口感染等,严重者可危及生命,是导致医院感染最常见的一种致病菌[1-2]。20世纪80年代中期在我国台湾地区首次分离出高黏液肺炎克雷伯菌(hypermucoviscous Klebsiella pneumoniae, hmKp),与经典肺炎克雷伯菌(classical Klebsiella pneumoniae, cKp)相比,hmKp在菌落外观上一般表现出高黏液性,临床上通常将具有高黏液表型的Kp判定为高毒力肺炎克雷伯菌(hypervirulent Klebsiella pneumoniae, hvKp)[3]。研究[4]发现,不是所有的hvKp菌株都表现为高黏性特征,也并非所有hmKp菌株都具有高毒力,hvKp的鉴定需要从菌株的表型、基因型, 以及临床致病能力等方面综合判断。
Kp的高黏液表型受黏液表型调节基因A(rmpA)、黏性相关基因(magA)、控制细菌荚膜多糖(CPS)合成基因等因子调控,毒力与铁载体、荚膜多糖、菌毛等多种毒力因子密切相关,对Kp的这些毒力因子进行研究有助于深入研究Kp的致病机制,对于正确认识Kp所致感染,降低Kp的危害具有重要意义[5-6]。传统观点认为,cKp菌株的耐药性较强,hvKp菌株虽然表现为高毒力表型,但对除氨苄西林外的临床常用抗生素均表现为敏感[7]。近年来,多重耐药hvKp菌株高流行暴发的趋势陆续被报道,2019年我国某院重症监护病房(ICU)出现了耐碳青霉烯类hvKp感染暴发,患者病死率为100%,96.6%的分离菌株对包括碳青霉烯类在内的多种抗生素具有耐药性[8]。hvKp菌株的高毒力与耐药性的重叠将给临床治疗带来巨大挑战。本研究对2019—2023年淮安市临床分离的hmKp耐药性及基因组特征进行研究,分析其耐药与毒力相关性因子,以期为hmKp感染临床合理用药治疗及明确hvKp的生物标志物提供理论依据。
1. 材料与方法
1.1 主要仪器与试剂
血琼脂平板(广东环凯微生物科技有限公司),革兰阴性菌鉴定板(法国梅里埃公司),VITEK 2 Compact全自动细菌生化鉴定仪(法国梅里埃公司),BD Phoenix M50全自动微生物鉴定药敏分析仪(美国BD公司)。
1.2 菌株来源与鉴定
Kp菌株来源于2019—2023年淮安市国家致病菌识别网哨点医院的临床标本。将收集的菌株划线分离于血琼脂平板,观察菌株纯度,使用VITEK 2 Compat全自动细菌生化鉴定仪进行鉴定复核。hmKp菌株判定标准:用接种环轻轻向上挑起血平板上的Kp单菌落,重复牵拉两次或两次以上,挑起的拉丝长度≥5 mm的菌株判定为hmKp菌株,并统计分析hmKp来源标本类型。
1.3 细菌全基因组测序
hmKp菌株送上海伯杰医疗科技股份有限公司进行全基因组二代测序,测序平台为Illumina NovaSeq,测序深度大于100×。下机数据使用fastp(v0.23.2)滤除低质量reads,然后使用SPAdes(v3.15.5)对过滤后的数据进行无参组装拼接。
1.4 基于全基因组测序的分子分型
将菌株全基因组序列上传至BacWGSTdb 2.0[9]数据库进行多位点序列分型(ST)分析。以HS11286(CP003200)为参考基因组进行核心基因组多位点序列分型(cgMLST)分析,设定核心基因差异数≤30的菌株为相同克隆复合群,并绘制核心基因组多位点序列分型树(MSTree)。
1.5 耐药基因和毒力基因注释
利用CARD数据库(http://arpcard.mcmaster.ca)对菌株基因组进行耐药基因注释,利用VFDB数据库(http://www.mgc.ac.cn/VFs/) 对菌株基因组进行毒力基因注释,选取与相应数据库中已知基因覆盖度(coverage值)和同源性(identity值)均大于90%的基因进行统计。
1.6 药物敏感试验
采用微量肉汤稀释法检测菌株的耐药表型。根据美国临床实验室标准化协会(CLSI)标准(2022)进行耐药折点判读。依据《多重耐药菌医院感染预防与控制技术指南(试行)》,对3类或3类以上抗菌药物具有耐药性的菌株判定为多重耐药菌株。对亚胺培南或美罗培南耐药的Kp菌株判定为耐碳青霉烯类肺炎克雷伯菌(CRKp)。质控菌株为大肠埃希菌ATCC 25922、肺炎克雷伯菌ATCC 700603。
1.7 统计方法
应用SPSS软件对耐药数据进行卡方检验,以P≤0.05为差异有统计学意义。对菌株耐药表型和耐药基因携带情况进行kappa一致性分析,kappa≤0.2表示一致性较差,0.2<kappa≤0.4表示一致性一般,0.4<kappa≤0.6表示一致性中等,0.6<kappa≤0.8表示有较高的一致性,kappa>0.8表示有很好的一致性。
2. 结果
2.1 hmKp鉴定结果
经生化鉴定和拉丝试验,共检出hmKp 60株。分离自10种临床标本,以痰(33.33%,20株)和血(28.33%,17株)标本为主,其次为支气管肺泡灌洗液(8.33%,5株)、引流液(6.67%,4株)、尿(6.67%,4株)、粪便(6.67%,4株)、咽拭子(5.00%,3株)标本,腹腔积液、穿刺液、分泌物各分离1株(各占1.67%)。
2.2 全基因组测序与ST结果
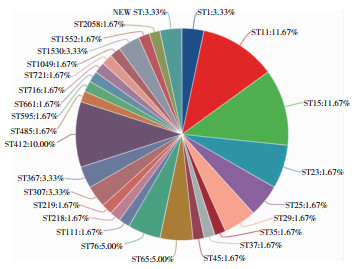
60株hmKp基因组经测序、过滤、拼接组装,基因组平均大小为5.6 (5.1~5.7)Mb,平均GC含量为57.09% (45.99%~57.82%)。60株hmKp共有26种已知ST型和2种新ST型。优势ST型为ST11(11.67%,7株)、ST15(11.67%,7株)和ST412(10.00%,6株)。见图 1。ST412菌株主要分离自痰标本。
利用BacWGSTdb 2.0数据库构建的MSTree见图 2。菌株分布分散,无聚集性菌株,相同ST型菌株间距离更近,按照核心基因组差异数≤30作为判断菌株之间具有高度同源性的标准,高度同源的菌株有ST11 3株,ST15、ST76、ST1各2株,结合这些菌株的临床背景信息,排除重复菌株和散在暴发菌株的可能性,表明这些菌株在本地区流行。
2.3 耐药表型
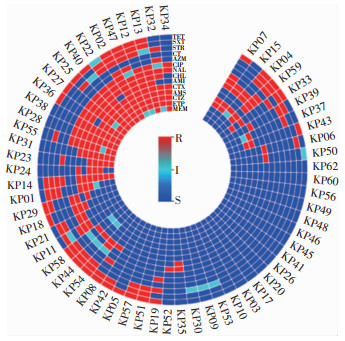
60株hmKp对10类14种抗菌药物敏感性试验结果显示,21.67%(13株)的菌株对所有测试抗菌药物敏感,其余菌株至少对1种抗菌药物耐药或中介,多重耐药菌株占60.00%(36株),最多对13种抗菌药物耐药。60株hmKp耐药表型热图见图 3。
60株hmKp检出CRKp 17株,碳青霉烯类敏感肺炎克雷伯菌(non-CRKp)43株。除复方磺胺甲
 表 1 hmKp对抗菌药物的药敏试验结果Table 1 Antimicrobial susceptibility testing results of hmKp
表 1 hmKp对抗菌药物的药敏试验结果Table 1 Antimicrobial susceptibility testing results of hmKp抗菌药物 CRKp(n=17) non-CRKp(n=43) 合计(n=60) 耐药株数 耐药率(%) 耐药株数 耐药率(%) 耐药株数 耐药率(%) 氨苄西林/舒巴坦 17 100 5 11.63 22 36.67 头孢他啶 15 88.24 2 4.65 17 28.33 头孢噻肟 17 100 9 20.93 26 43.33 亚胺培南 17 100 0 0 17 28.33 美罗培南 14 82.35 0 0 14 23.33 阿米卡星 12 70.59 3 6.98 15 25.00 链霉素 8 47.06 12 27.91 20 33.33 四环素 8 47.06 16 37.21 24 40.00 氯霉素 15 88.24 11 25.58 26 43.33 阿奇霉素 5 29.41 5 11.63 10 16.67 萘啶酸 14 82.35 11 25.58 25 41.67 环丙沙星 12 70.59 8 18.60 20 33.33 复方磺胺甲口恶唑 6 35.29 16 37.21 22 36.67 多黏菌素E 1 5.88 2 4.65 3 5.00 2.4 耐药基因携带情况及注释
60株hmKp携带了编码抗性蛋白、外排泵、酶灭活,以及抗菌药物作用位点改变的四种耐药机制对14类抗菌药物耐药的64种耐药基因(覆盖度>90%、同源率>90%)。按照抗菌药物大类统计,磷霉素相关耐药基因和超广谱β-内酰胺类抗生素相关耐药基因在菌株中携带率较高,分别为100%(60株)、98.33%(59株)。碳青霉烯类耐药基因blaKPC-2由ST11(7株)、ST1(2株)和ST15(4株)菌株携带,blaNDM-5基因由ST76(2株)菌株携带。64种耐药基因中有54种位于可移动原件上,其中喹诺酮类、利福霉素类、大环内酯类、碳青霉烯类、产超广谱β-内酰胺类、二氨基嘧啶类、磺胺类抗菌药物耐药相关基因均位于可移动原件上。见表 2。
表 2 60株hmKp耐药基因携带情况Table 2 Carriage of drug-resistance genes in 60 hmKp strains耐药机制及耐抗菌药物类别 耐药基因 位于可移动原件 携带株数 携带率(%) 耐药机制及耐抗菌药物类别 耐药基因 位于可移动原件 携带株数 携带率(%) 抗性蛋白 blaSHV-148 是 6 10.00 喹诺酮类 qnrS1 是 17 28.33 blaCTX-M-55 是 1 1.67 qnrB4 是 1 1.67 blaSHV-11 是 7 11.67 外排泵 blaSHV-62 是 1 1.67 四环素类 tet(A) 否 19 31.67 blaSHV-172 是 1 1.67 tet(D) 否 3 5.00 blaOKP-B-14 是 1 1.67 氯霉素类 cmlA1 是 1 1.67 氨基糖苷类 aac(3)-IId 是 15 25.00 floR 否 11 18.33 aac(6’)-Ib-cr 是 5 8.33 酶灭活 aph(3”)-Ib 是 4 6.67 利福霉素类 ARR-3 是 1 1.67 aph(6)-Id 是 6 10.00 ARR-2 是 1 1.67 aac(3)-IIa 是 2 3.33 大环内酯类 mph(A) 是 2 3.33 aac(3)-IV 是 2 3.33 mph(E) 是 1 1.67 aph(3')-Ia 是 8 13.33 msr(E) 是 1 1.67 aph(4)-Ia 是 2 3.33 碳青霉烯类 blaKPC-2 是 13 21.67 aadA1 是 1 1.67 blaNDM-5 是 2 3.33 aadA2 是 3 5.00 超广谱β-内酰胺类 blaSHV-40 是 6 10.00 aadA16 是 2 3.33 blaCTX-M-14 是 8 13.33 aadA5 是 1 1.67 blaCTX-M-3 是 5 8.33 磷霉素 fosA 否 58 96.67 blaSHV-182 是 5 8.33 fosA3 否 1 1.67 blaTEM-1B 是 14 23.33 fosB1 否 1 1.67 blaLAP-2 是 7 11.67 fosA5 否 2 3.33 blaSHV-187 是 8 13.33 氯霉素类 catA2 否 6 10.00 blaSHV-27 是 2 3.33 第三代头孢菌素 blaDHA-1 是 2 3.33 blaCTX-M-27 是 1 1.67 靶位改变 blaSHV-106 是 9 15.00 氨基糖苷类 armA 否 4 6.67 blaSHV-190 是 4 6.67 rmtB 否 4 6.67 blaCTX-M-65 是 8 13.33 二氨基嘧啶类 dfrA1 是 6 10.00 blaSHV-110 是 4 6.67 dfrA17 是 2 3.33 blaLEN16 是 2 3.33 dfrA27 是 1 1.67 blaCTX-M-15 是 5 8.33 dfrA12 是 1 1.67 blaOXA-1 是 4 6.67 dfrA14 是 2 3.33 blaSHV-33 是 3 5.00 磺胺类 sul1 是 13 21.67 blaSHV-164 是 3 5.00 sul2 是 5 8.33 通过菌株基因组中耐药基因携带情况,预测其具有相应耐药表型的准确率在44.07%~100%,其中磺胺类药物的预测准确率最高,产β-内酰胺类药物的预测准确率最低。磺胺类、碳青霉烯类、四环素类抗菌药物药敏试验结果与菌株携带相关耐药基因情况一致性较高;喹诺酮类、氨基糖苷类、氯霉素类、大环内酯类抗菌药物药敏试验结果与菌株携带相应耐药基因情况一致性一般;β-内酰胺类药敏试验结果与菌株携带相应耐药基因情况一致性较差。部分菌株携带相关耐药基因但对测试的抗菌药物敏感,可能与本研究测试的抗菌药物种类较少有关,如β-内酰胺类抗生素种类较多,本研究中只选择了2种;部分菌株对抗菌药物耐药,但未检测到相应的耐药基因,可能与基因注释方法或存在新的耐药基因有关。见表 3。
表 3 菌株耐药表型与携带耐药基因相关性Table 3 Correlation between resistance phenotype and carrying of resistance genes of strains抗菌药物 耐药/中介(株) 敏感(株) 药敏结果与耐药基因携带情况一致性kappa系数 耐药基因预测耐药准确率(%) 携带耐药基因 不携带耐药基因 携带耐药基因 不携带耐药基因 磺胺类 16 6 0 38 0.772 100(16/16) 碳青霉烯类 13 4 2 41 0.745 86.67(13/15) β-内酰胺类 26 0 33 1 0.026 44.07(26/59) 四环素类 18 8 3 31 0.618 85.71(18/21) 喹诺酮类 15 12 1 32 0.545 93.75(15/16) 大环内酯类 2 8 1 49 0.250 66.67(2/3) 氨基糖苷类 21 10 5 24 0.502 80.77(21/26) 多肽类 0 3 0 57 - - 氯霉素类 14 12 1 33 0.536 93.33(14/15) 注:-代表不适用kappa系数计算一致性。 2.5 毒力基因携带结果及注释
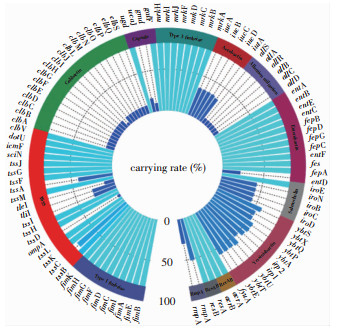
60株hmKp菌株基因组中存在与菌毛、外毒素、荚膜多糖、铁载体等致病因子相关的13类101种毒力基因,其中Colibactin和Ⅵ型分泌系统相关毒力基因种类最多,均有17个。所有菌株均携带荚膜合成调节相关基因(rcsA、rcsB)、外排泵相关基因(acrA、acrB)、肠杆菌素相关基因(entABCEF、fepABCDFG、fes),Ⅰ型和Ⅲ型菌毛相关基因的携带率均>95%。部分毒力基因与菌株ST型密切相关,如allA、allB、allC、allD、allR、allS均在ST23菌株中,tle1基因均由ST11菌株携带,Colibactin的17种毒力基因主要由ST23菌株携带,优势ST型菌株ST11、ST15、ST412均不携带Allantion utilization和Colibactin相关毒力基因。60株hmKp毒力基因携带情况见图 4。
3. 讨论
研究[10]表明,hmKp更易引起侵袭性感染,且不仅能够感染免疫功能低下者,还能在健康个体中出现危及生命的社区获得性感染。临床上hmKp检出率较高的标本包括痰、尿等,宁波地区重症感染患儿痰标本中hvKp检出率高达97.96%[11]。本研究中,60株hmKp来源标本类型广泛,以痰和血标本为主,与刘淑敏等[4]报道的结果相似。
60株hmKp ST型多样,提示本地区hmKp菌群结构分散,优势ST型为ST11、ST15和ST412,与以往研究[4, 12]不同。除ST412型菌株(主要分离自痰标本)外,菌株ST型与其来源标本类型之间无明显相关。通过cgMLST方法进一步分析,60株菌株中有部分菌株高度同源,但未发现暴发菌株。通过比较进一步表明,cgMLST比MLST具有更高的分辨力,适合亲缘关系较近菌株间的分型。ST11型hvKp是目前亚洲地区流行的耐碳青霉烯类抗生素hvKp菌株的主要型别,携带pLVPK质粒;ST15型hvKp菌株也被报道携带含有多重耐药基因和毒力基因的融合质粒,两种ST型别菌株均可水平传播并导致致命感染[13-14],应引起足够重视,继续加强此类菌株的监测力度。
本研究中菌株对氯霉素类、甲氧苄啶类、碳青霉烯类、头孢菌素类、四环素类、喹诺酮类、氨基糖苷类抗菌药物的耐药率为23.33%~43.33%,其中复方磺胺甲
本研究中菌株携带的毒力基因种类较多,部分毒力基因与菌株ST型密切相关。位于质粒上、作为hvKp标志的iroB、iucA、rmpA和rmpA2基因携带率分别为43.33%、35.00%、51.67%、30.00%[18]。rmpA、rmpA2、铁载体是导致hvKp菌株比cKp菌株毒力更强的关键因素,这几种毒力因子在本研究菌株中的携带率均较高,说明这些菌株具有较强的致病力。
综上所述,对该地区临床分离hmKp的耐药性,以及相关机制、毒力因子携带情况进行分析,为有效遏制hmKp传播和暴发流行提供了数据支撑。
利益冲突:所有作者均声明不存在利益冲突。
-
表 1 hmKp对抗菌药物的药敏试验结果
Table 1 Antimicrobial susceptibility testing results of hmKp
抗菌药物 CRKp(n=17) non-CRKp(n=43) 合计(n=60) 耐药株数 耐药率(%) 耐药株数 耐药率(%) 耐药株数 耐药率(%) 氨苄西林/舒巴坦 17 100 5 11.63 22 36.67 头孢他啶 15 88.24 2 4.65 17 28.33 头孢噻肟 17 100 9 20.93 26 43.33 亚胺培南 17 100 0 0 17 28.33 美罗培南 14 82.35 0 0 14 23.33 阿米卡星 12 70.59 3 6.98 15 25.00 链霉素 8 47.06 12 27.91 20 33.33 四环素 8 47.06 16 37.21 24 40.00 氯霉素 15 88.24 11 25.58 26 43.33 阿奇霉素 5 29.41 5 11.63 10 16.67 萘啶酸 14 82.35 11 25.58 25 41.67 环丙沙星 12 70.59 8 18.60 20 33.33 复方磺胺甲口恶唑 6 35.29 16 37.21 22 36.67 多黏菌素E 1 5.88 2 4.65 3 5.00 表 2 60株hmKp耐药基因携带情况
Table 2 Carriage of drug-resistance genes in 60 hmKp strains
耐药机制及耐抗菌药物类别 耐药基因 位于可移动原件 携带株数 携带率(%) 耐药机制及耐抗菌药物类别 耐药基因 位于可移动原件 携带株数 携带率(%) 抗性蛋白 blaSHV-148 是 6 10.00 喹诺酮类 qnrS1 是 17 28.33 blaCTX-M-55 是 1 1.67 qnrB4 是 1 1.67 blaSHV-11 是 7 11.67 外排泵 blaSHV-62 是 1 1.67 四环素类 tet(A) 否 19 31.67 blaSHV-172 是 1 1.67 tet(D) 否 3 5.00 blaOKP-B-14 是 1 1.67 氯霉素类 cmlA1 是 1 1.67 氨基糖苷类 aac(3)-IId 是 15 25.00 floR 否 11 18.33 aac(6’)-Ib-cr 是 5 8.33 酶灭活 aph(3”)-Ib 是 4 6.67 利福霉素类 ARR-3 是 1 1.67 aph(6)-Id 是 6 10.00 ARR-2 是 1 1.67 aac(3)-IIa 是 2 3.33 大环内酯类 mph(A) 是 2 3.33 aac(3)-IV 是 2 3.33 mph(E) 是 1 1.67 aph(3')-Ia 是 8 13.33 msr(E) 是 1 1.67 aph(4)-Ia 是 2 3.33 碳青霉烯类 blaKPC-2 是 13 21.67 aadA1 是 1 1.67 blaNDM-5 是 2 3.33 aadA2 是 3 5.00 超广谱β-内酰胺类 blaSHV-40 是 6 10.00 aadA16 是 2 3.33 blaCTX-M-14 是 8 13.33 aadA5 是 1 1.67 blaCTX-M-3 是 5 8.33 磷霉素 fosA 否 58 96.67 blaSHV-182 是 5 8.33 fosA3 否 1 1.67 blaTEM-1B 是 14 23.33 fosB1 否 1 1.67 blaLAP-2 是 7 11.67 fosA5 否 2 3.33 blaSHV-187 是 8 13.33 氯霉素类 catA2 否 6 10.00 blaSHV-27 是 2 3.33 第三代头孢菌素 blaDHA-1 是 2 3.33 blaCTX-M-27 是 1 1.67 靶位改变 blaSHV-106 是 9 15.00 氨基糖苷类 armA 否 4 6.67 blaSHV-190 是 4 6.67 rmtB 否 4 6.67 blaCTX-M-65 是 8 13.33 二氨基嘧啶类 dfrA1 是 6 10.00 blaSHV-110 是 4 6.67 dfrA17 是 2 3.33 blaLEN16 是 2 3.33 dfrA27 是 1 1.67 blaCTX-M-15 是 5 8.33 dfrA12 是 1 1.67 blaOXA-1 是 4 6.67 dfrA14 是 2 3.33 blaSHV-33 是 3 5.00 磺胺类 sul1 是 13 21.67 blaSHV-164 是 3 5.00 sul2 是 5 8.33 表 3 菌株耐药表型与携带耐药基因相关性
Table 3 Correlation between resistance phenotype and carrying of resistance genes of strains
抗菌药物 耐药/中介(株) 敏感(株) 药敏结果与耐药基因携带情况一致性kappa系数 耐药基因预测耐药准确率(%) 携带耐药基因 不携带耐药基因 携带耐药基因 不携带耐药基因 磺胺类 16 6 0 38 0.772 100(16/16) 碳青霉烯类 13 4 2 41 0.745 86.67(13/15) β-内酰胺类 26 0 33 1 0.026 44.07(26/59) 四环素类 18 8 3 31 0.618 85.71(18/21) 喹诺酮类 15 12 1 32 0.545 93.75(15/16) 大环内酯类 2 8 1 49 0.250 66.67(2/3) 氨基糖苷类 21 10 5 24 0.502 80.77(21/26) 多肽类 0 3 0 57 - - 氯霉素类 14 12 1 33 0.536 93.33(14/15) 注:-代表不适用kappa系数计算一致性。 -
[1] 李雪娇, 马炜, 郭杰, 等. 耐碳青霉烯类肺炎克雷伯菌的流行病学特征及分子生物学研究[J]. 第二军医大学学报, 2020, 41(10): 1109-1114. Li XJ, Ma W, Guo J, et al. Epidemiological characteristics and molecular biology of carbapenem-resistant Klebsiella pneumoniae[J]. Academic Journal of Second Military Medical University, 2020, 41(10): 1109-1114. [2] 郑茂, 陈宗耀, 鄂建飞, 等. 高毒力肺炎克雷伯菌对碳青霉烯类耐药机制研究进展[J]. 现代检验医学杂志, 2022, 37(1): 207-212. Zheng M, Chen ZY, E JF, et al. Research progress on the mechanism of hypervirulent Klebsiella pneumoniae resistance to carbapenems[J]. Journal of Modern Laboratory Medicine, 2022, 37(1): 207-212. [3] 张可依, 陶传敏. 高毒力肺炎克雷伯菌的定义和毒力相关因素研究进展[J]. 四川医学, 2021, 42(1): 86-92. Zhang KY, Tao CM. Research progress on definition and virulence related factors of hypervirulent Klebsiella pneumoniae[J]. Sichuan Medical Journal, 2021, 42(1): 86-92. [4] 刘淑敏, 杜艳, 毕建蝶. 高黏液型肺炎克雷伯菌毒力基因及分子流行病学研究[J]. 中国抗生素杂志, 2023, 48(6): 672-677. Liu SM, Du Y, Bi JD. Study on the virulence gene and molecu- lar epidemiology of hypermucoviscous Klebsiella pneumoniae[J]. Chinese Journal of Antibiotics, 2023, 48(6): 672-677. [5] 陈颖聪, 屠艳烨, 高晖, 等. 高黏液表型肺炎克雷伯菌的临床分布特征及毒力分析[J]. 浙江医学, 2024, 46(3): 257-261, 268. Chen YC, Tu YY, Gao H, et al. Clinical distribution and viru- lence analysis of hypermucoviscous Klebsiella pneumoniae[J]. Zhejiang Medical Journal, 2024, 46(3): 257-261, 268. [6] 张颖, 李轶, 郭思. 高毒力肺炎克雷伯菌分子致病机制研究进展[J]. 检验医学与临床, 2020, 17(9): 1298-1301. Zhang Y, Li Y, Guo S. Advances in molecular pathogenesis of hypervirulent Klebsiella pneumoniae[J]. Laboratory Medicine and Clinic, 2020, 17(9): 1298-1301. [7] 王华, 王俊瑞, 郑文琪. 高毒力肺炎克雷伯菌致病及耐药分子机制研究进展[J]. 微生物学通报, 2021, 48(1): 288-294. Wang H, Wang JR, Zheng WQ. Research progress in the mechanism of molecular pathogenesis and drug resistance of hypervirulent Klebsiella pneumoniae[J]. Microbiology China, 2021, 48(1): 288-294. [8] Zhao YJ, Zhang XC, Torres VVL, et al. An outbreak of carbapenem-resistant and hypervirulent Klebsiella pneumoniae in an intensive care unit of a major teaching hospital in Wenzhou, China[J]. Front Public Health, 2019, 7: 229. [9] Feng Y, Zou SM, Chen HF, et al. BacWGSTdb 2.0: a one-stop repository for bacterial whole-genome sequence typing and source tracking[J]. Nucleic Acids Res, 2021, 49(D1): D644-D650. [10] Cheng E, Ou FS, Ma C, et al. Diet and lifestyle-based prediction models to estimate cancer recurrence and death in patients with stage Ⅲ colon cancer (CALGB 89803/Alliance)[J]. J Clin Oncol, 2022, 40(7): 740-751. [11] 张杰, 吴军华, 陈杨福. 宁波地区重症感染患儿肺炎克雷伯菌临床分离株耐药性和毒力特征[J]. 中华医院感染学杂志, 2023, 33(17): 2707-2710. Zhang J, Wu JH, Chen YF. Drug resistance and virulence characteristics of clinical isolates of Klebsiella pneumoniae from children with severe infection in Ningbo[J]. Chinese Journal of Nosocomiology, 2023, 33(17): 2707-2710. [12] 金美玲. 某医院高黏液型肺炎克雷伯菌的临床与基因组特征分析[D]. 沈阳: 中国医科大学, 2023. Jin ML. Clinical features and genomic analysis of hypermucoviscous Klebsiella pneumoniae in a hospital[D]. Shenyang: China Medical University, 2023. [13] 陈宪娣, 李宜檀, 王明贞, 等. 高毒力肺炎克雷伯菌的药物敏感性及分子流行病学特征[J]. 热带医学杂志, 2023, 23(11): 1512-1517. Chen XD, Li YT, Wang MZ, et al. Study on drug susceptibi-lity and molecular epidemiology in hypervirulent Klebsiella pneumoniae[J]. Journal of Tropical Medicine, 2023, 23(11): 1512-1517. [14] Lam MMC, Wyres KL, Wick RR, et al. Convergence of virulence and MDR in a single plasmid vector in MDR Klebsiella pneumoniae ST15[J]. J Antimicrob Chemother, 2019, 74(5): 1218-1222. [15] 江培涛, 方敏, 马超. 高黏液型肺炎克雷伯菌毒力基因及临床特征分析[J]. 检验医学与临床, 2023, 20(12): 1740-1744. Jiang PT, Fang M, Ma C. Analysis on virulence genes and clinical characteristics of high-viscosity Klebsiella pneumoniae[J]. Lab Med Cli, 2023, 20(12): 1740-1744. [16] 沈垲智, 刘卫平, 王涛. 耐碳青霉烯类肺炎克雷伯菌的耐药性分析及分子分型[J]. 中国消毒学杂志, 2023, 40(10): 767-770. Shen KZ, Liu WP, Wang T. Drug resistance analysis and molecular typing of carbapenem-resistant Klebsiella pneumoniae[J]. Chinese Journal of Disinfection, 2023, 40(10): 767-770. [17] 王青, 李耘, 郑波. 中国2017—2018年耐碳青霉烯类肺炎克雷伯菌药物敏感性及耐药基因分析[J]. 中国感染控制杂志, 2021, 20(5): 437-442. doi: 10.12138/j.issn.1671-9638.20217706 Wang Q, Li Y, Zheng B. Antimicrobial susceptibility and resistance genes of carbapenem-resistant Klebsiella pneumoniae in China from 2017 to 2018[J]. Chinese Journal of Infection Control, 2021, 20(5): 437-442. doi: 10.12138/j.issn.1671-9638.20217706 [18] 周君辰, 李娟, 周海健, 等. 高毒力肺炎克雷伯菌毒力和耐药机制研究进展[J]. 疾病监测, 2022, 37(11): 1474-1477. Zhou JC, Li J, Zhou HJ, et al. Progress in research of virulence and drug resistance mechanism of hypervirulent Klebsie-lla pneumonia[J]. Disease Surveillance, 2022, 37(11): 1474-1477.

 下载:
下载: